**论文题目:**Multi-level Protein Structure Pre-training via Prompt Learning **本文作者:**王泽元(浙江大学)、张强(浙江大学)、于浩然(浙江大学)、胡双威(仁为医学)、晋旭锐(德睿制药)、宫志晨(伦敦大学学院)、陈华钧(浙江大学) **发表会议:**ICLR 2023 **论文链接:**https://openreview.net/pdf?id=XGagtiJ8XC 代码链接:https://github.com/HICAI-ZJU/PromptProtein 欢迎转载,转载请注明出处
引言
为了理解蛋白质,研究者将其结构划分为四个等级:一级结构是指氨基酸序列;二级结构关注于蛋白质局部结构如-螺旋、-折叠;三级结构描述的是蛋白质3D结构;四级结构是由两个或多个蛋白质序列聚集而成的结构。在描述某些特定特征时,每种结构都有自己的优点和驱动力,它们不能相互替代。为了让模型同时具备多种蛋白质等级理解能力,使用多任务学习是非常自然的。然而,已有的研究发现,在多任务学习中,存在着任务间相互干扰的情况,多任务目标的组合可能会降低在单一结构等级学习上的表现。例如,语言建模让模型学习的是一维序列之间相互依赖的模式,而结构预测更关注于三维空间上的相关性。另一方面如何将每种结构的优点应用到下游任务也是多任务学习中的困难。这篇文章中,我们提出了提示符引导的对任务预训练和微调框架,通过利用提示符让模型减少在预训练阶段的任务冲突,并通过提示符微调技术,让结构信息可以更好的在下游任务中使用。
方法
提示符引导的预训练和微调框架
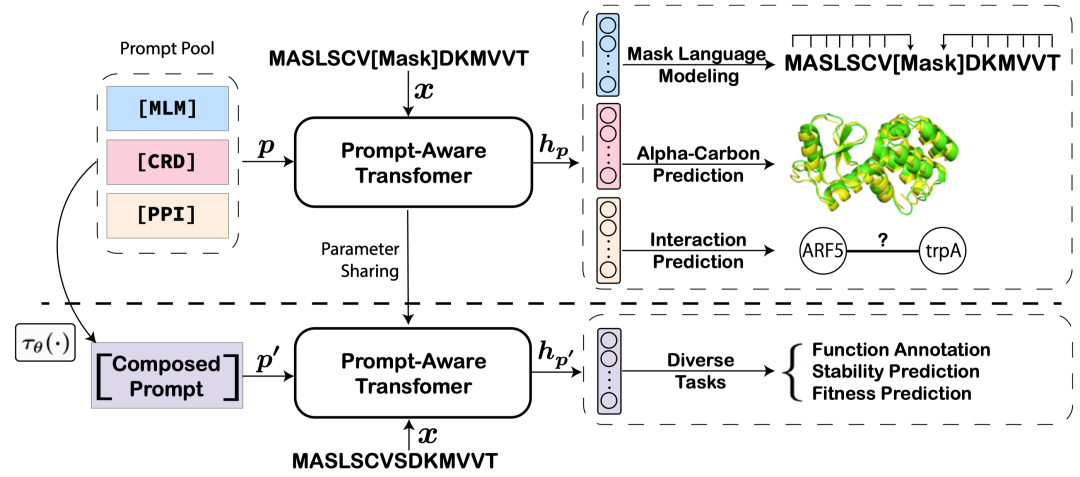
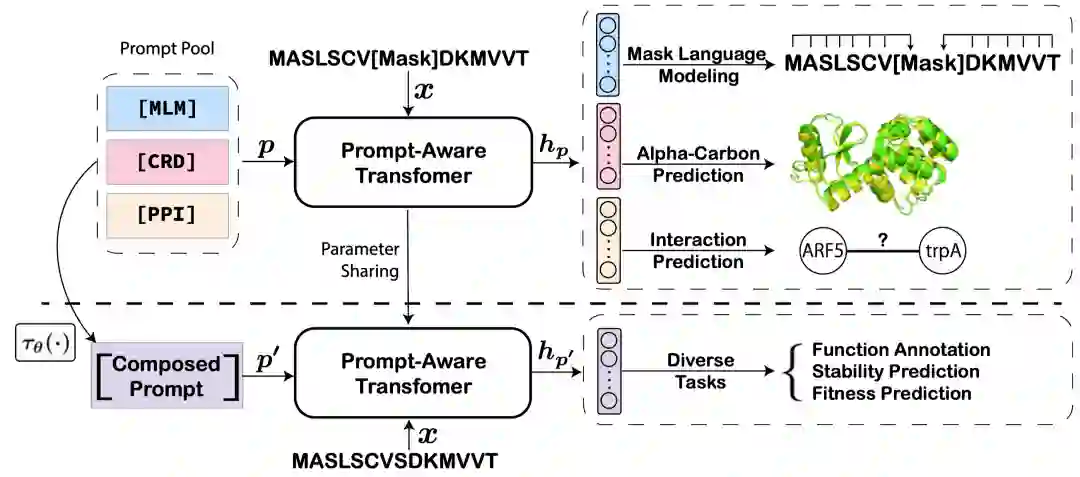
在预训练阶段,作者使用三个结构相关的任务对模型进行预训练(序列的语言建模,预测三级结构的碳坐标和预测蛋白质之间相互作用)。模型的输入不仅包含蛋白质序列,还包含任务对应的提示符。这样对于不同任务,相同蛋白质对应的表示是不同的,减少了任务间的冲突。在微调阶段,模型的参数固定,只微调任务提示符,通过任务提示符引导模型输出与下游任务最相关的结构表示。
模型框架
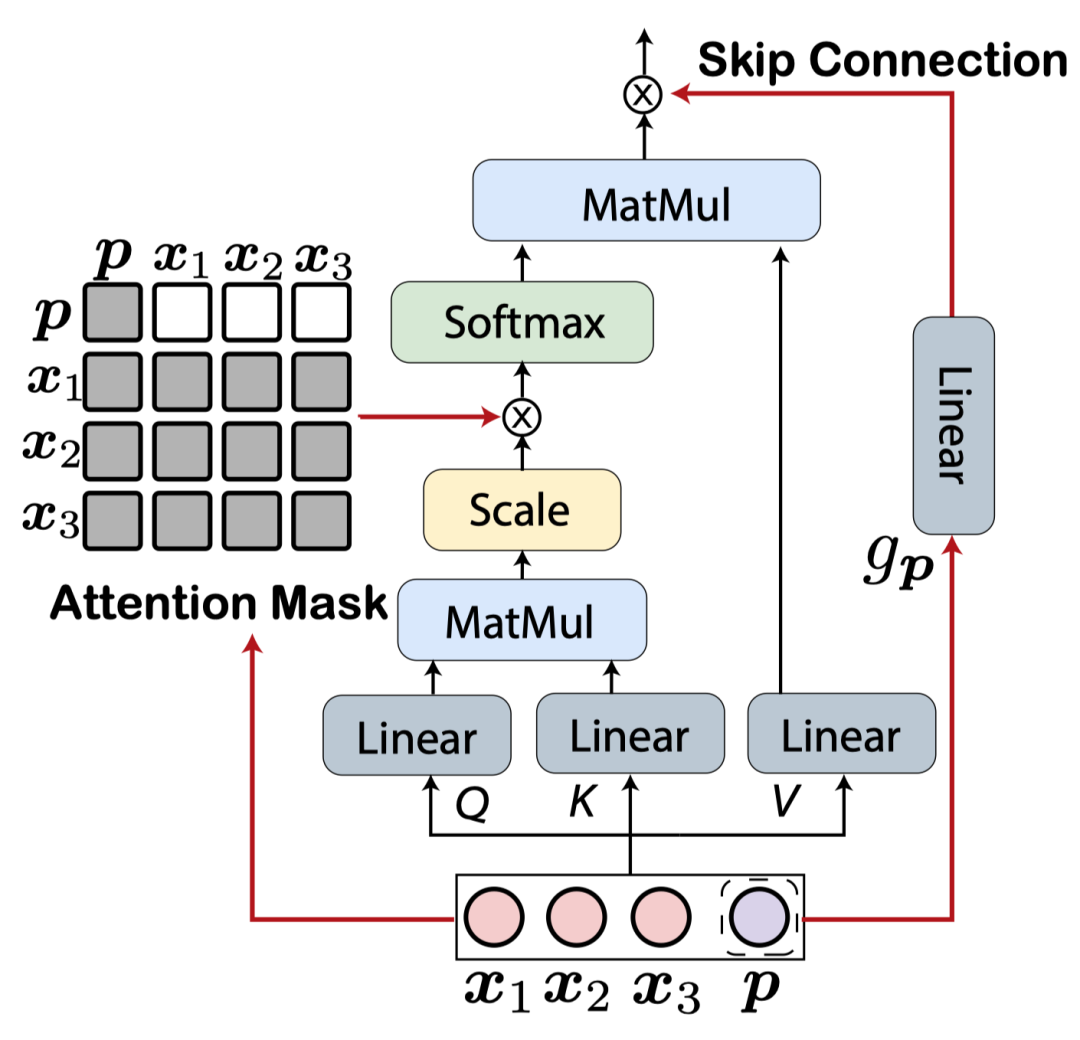
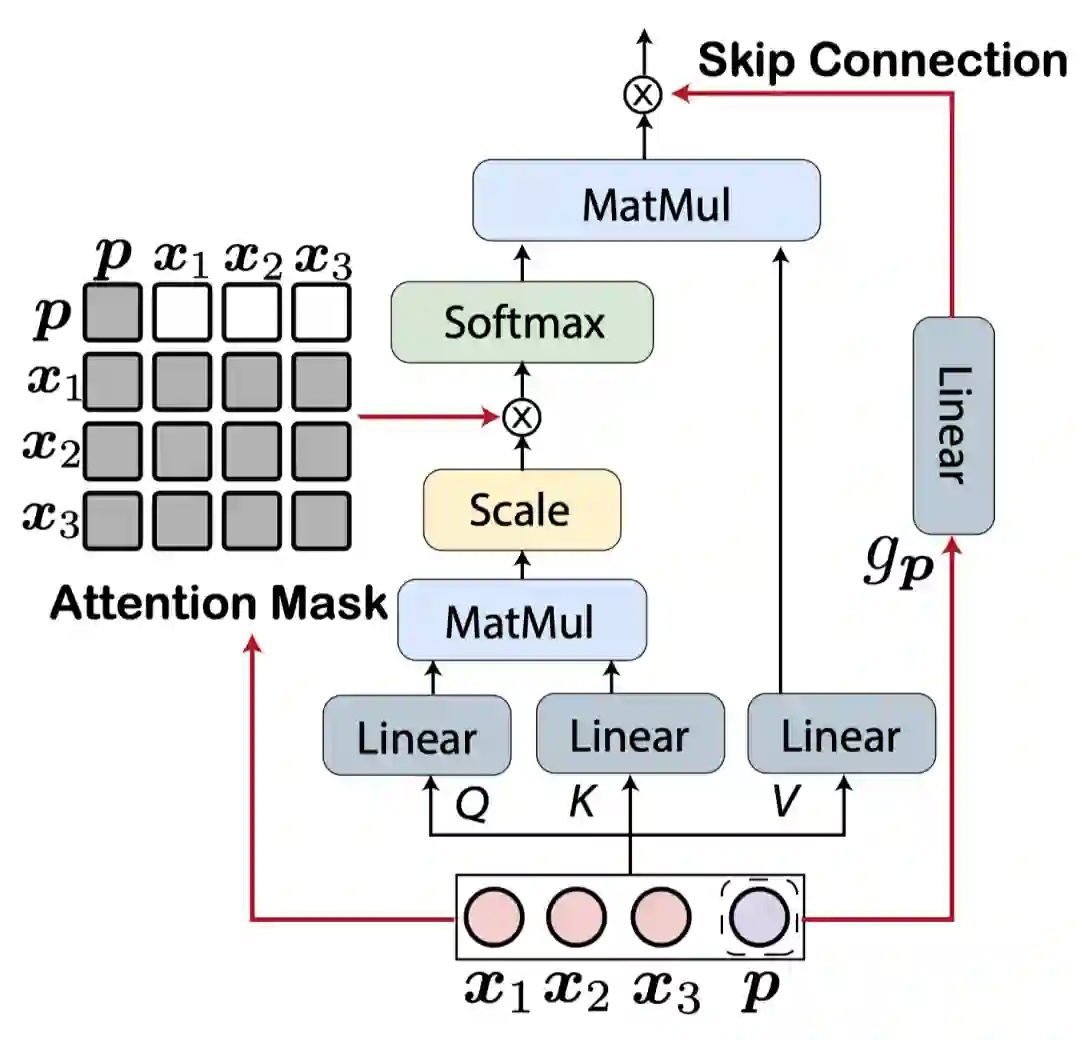
PromptProtein在传统的Transformer架构上做了调整,提出了提示符感知的注意力模块。传统的注意力网络中,每个token是根据整个序列的注意力权重来更新,这意味着任务提示符会根据蛋白质序列的变化而变化,这显然是不合理的。作者通过修改注意力掩码,让提示符的更新只依赖于自己来消除这种现象。为了鼓励不同预训练任务的独立性,减少任务间的干扰,作者还提出了带权重的跳跃链接,这使得一种任务可以仅依赖于部分层获得对应的表示。
**
**
实验
下游任务
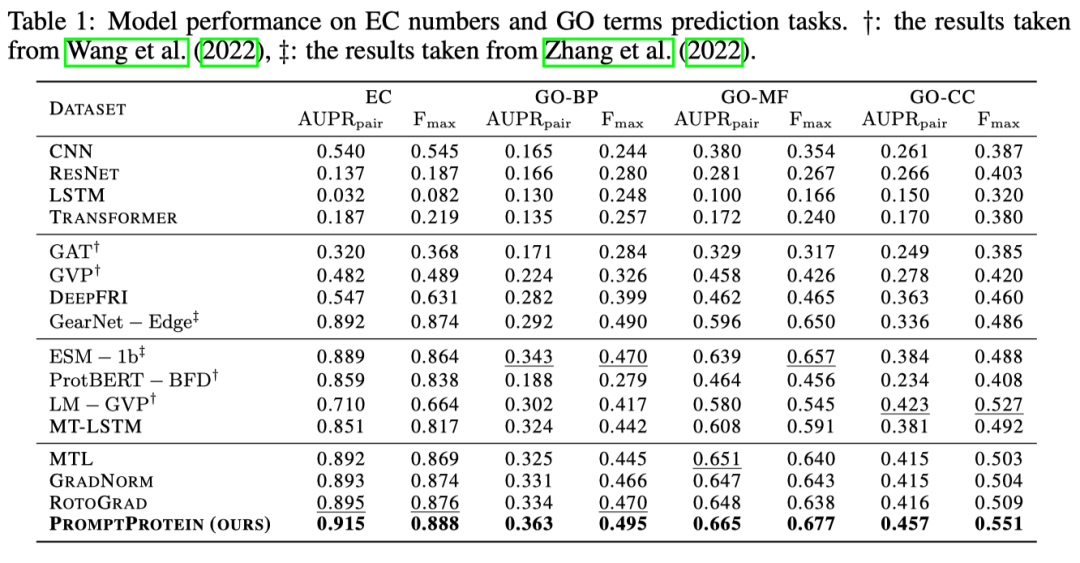
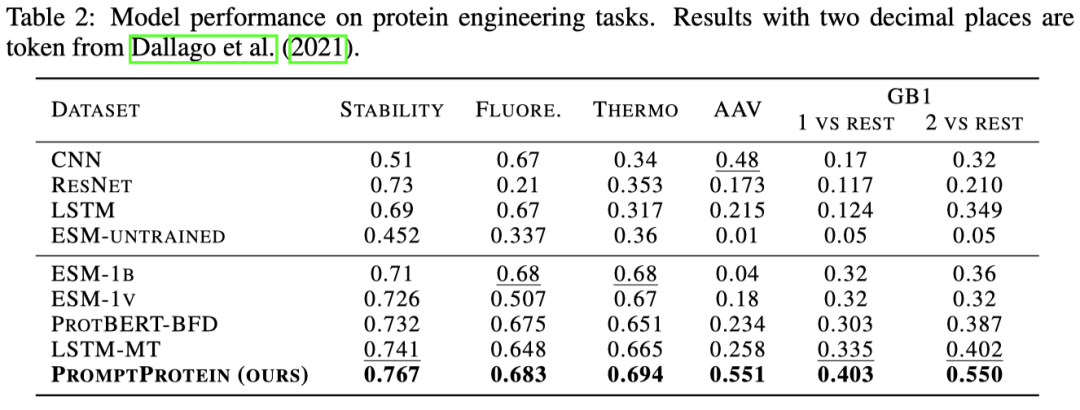
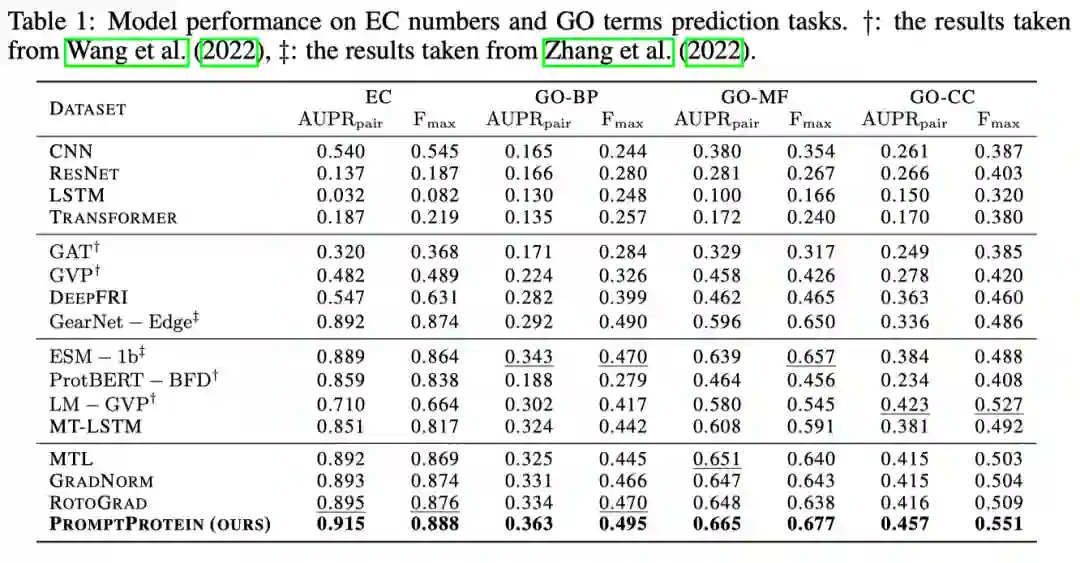
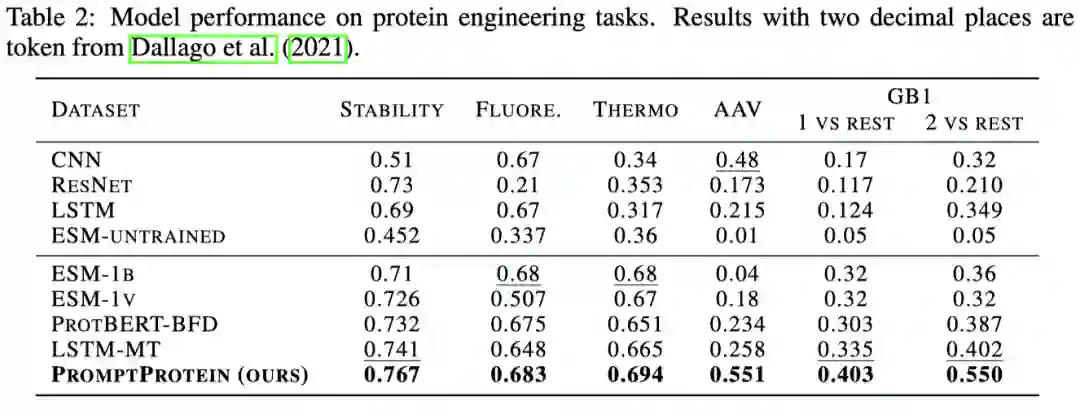
为了验证模型的学习能力,作者选择在蛋白质功能预测任务和蛋白质工程任务上进行评价。功能预测任务包含对蛋白质对应的基因本体(GO)术语和国际酶学会(EC)编号的预测,我们可以将其看作多个二分类任务。蛋白质工程任务评价的是模型区别蛋白质细微改变对其性质的影响包含了稳定性、亲和力的预测。实验结果表明多级结构信息可以有效的帮助模型提高对功能的预测准确性。虽然传统多任务学习的方法融合了这些多级信息,但是他们学习出来的是一个通用的表示,无法根据已有的下游任务选择利用哪些预训练知识。
分析实验
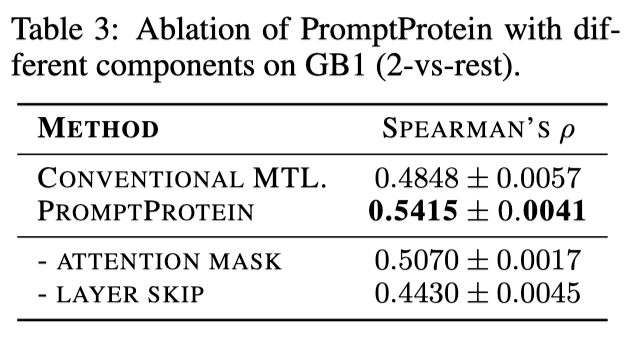
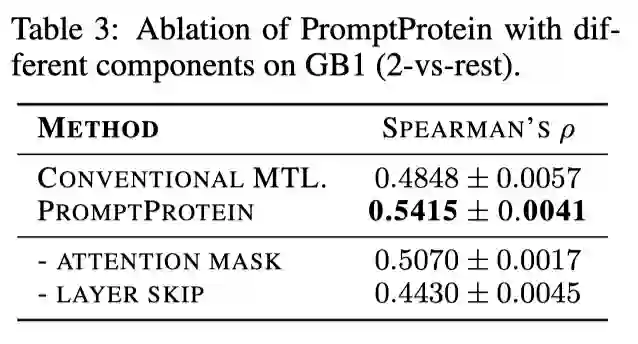
作者对预训练微调框架、模型设计做了消解。结果表明缺少任何一个模块,性能就会有不同程度的下降,证明了所有模块的有益性。
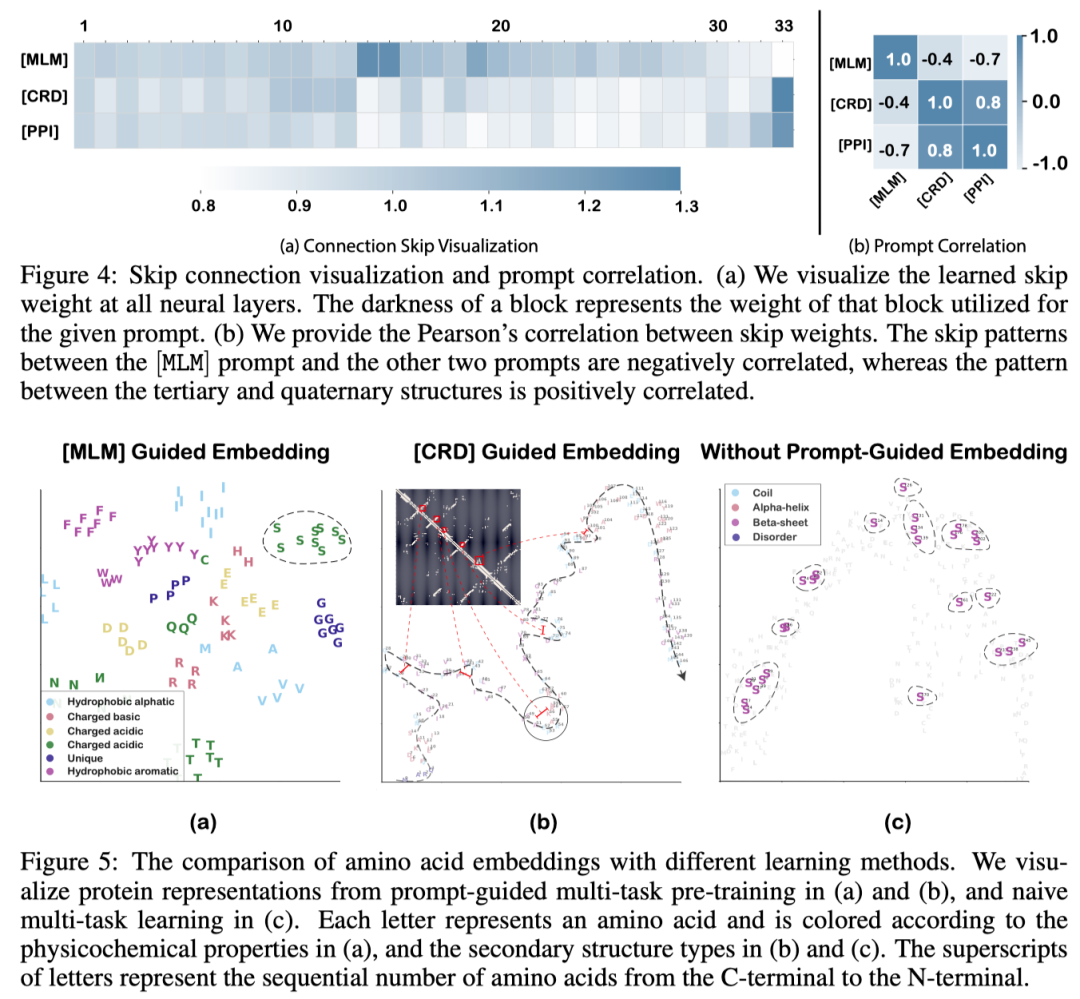
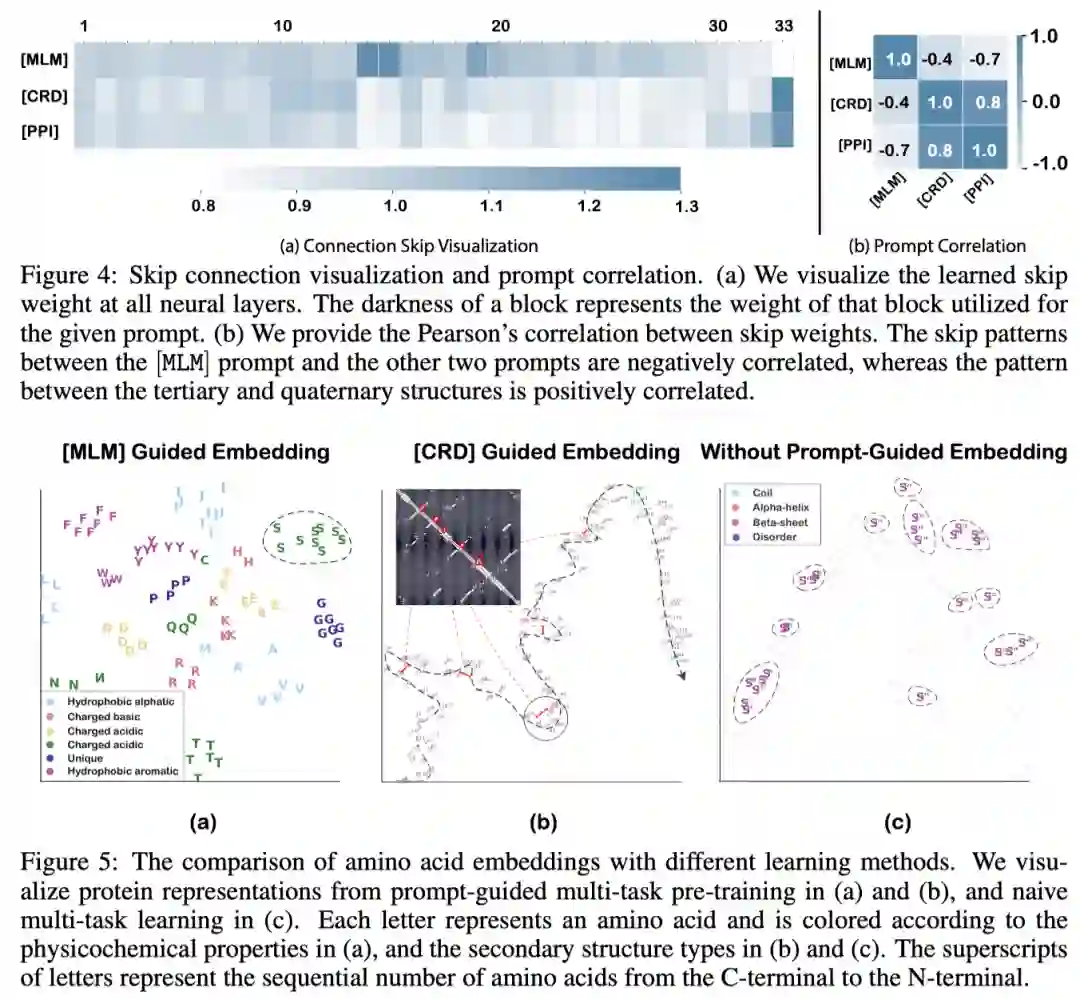
作者对不同提示符对应的跳跃连接和蛋白质表示进行了可视化,表明提示符对缓解任务间冲突的有效性。
结论
作者将提示学习的概念从NLP扩展到了蛋白质表示学习中,提出了提示符引导的多任务预训练和微调框架。利用这个框架,作者让模型通过多任务学习方法获得多级结构信息,在下游任务中可以灵活的利用这些信息。实验的结果表明,与传统的方法相比,该方法得到令人满意的改进。