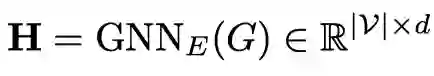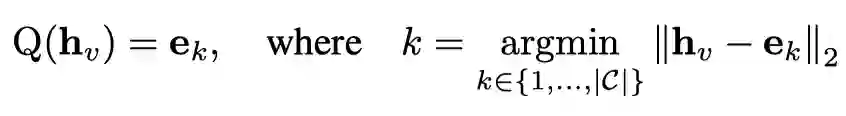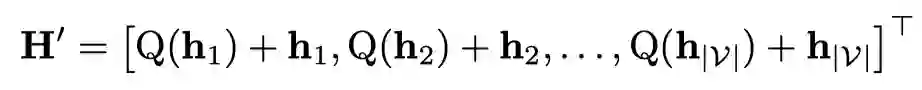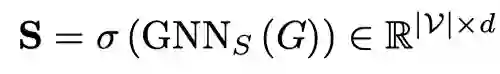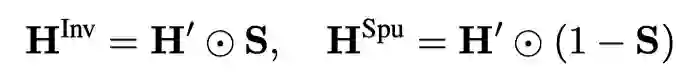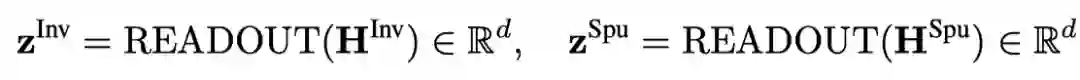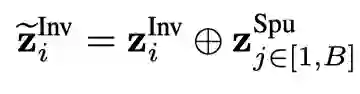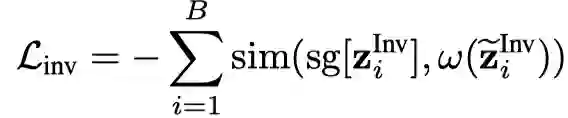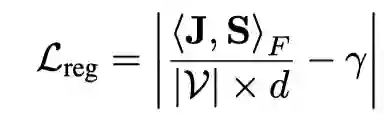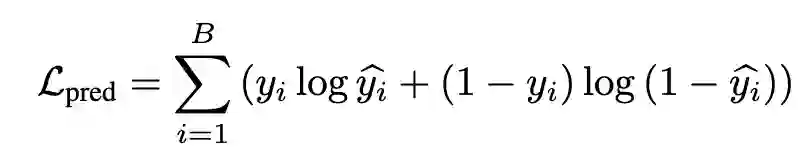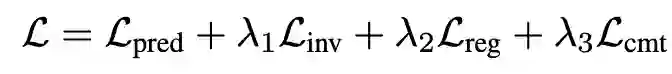论文题目:Learning Invariant Molecular Representation in Latent Discrete Space
本文作者:庄祥(浙江大学)、张强(浙江大学)、丁科炎(浙江大学)、卞亚涛(腾讯)、王啸(北京航空航天大学)、吕劲松(之江实验室)、陈红阳(之江实验室)、陈华钧(浙江大学)
**发表会议:**NeurIPS 2023 **论文链接:**https://arxiv.org/abs/2310.14170
****代码链接:https://github.com/HICAI-ZJU/iMoLD 欢迎转载,转载请注明出处
引言
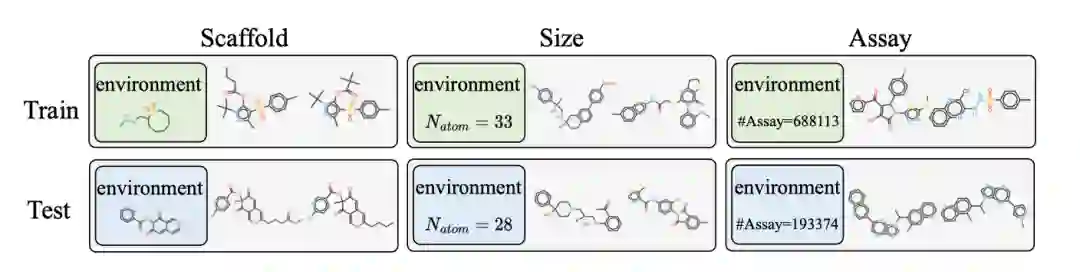
尽管分子表示学习取得了重大进展,但传统方法的一个普遍假设是,数据源是独立的,并且是从同一分布中采样的。然而,在实际药物开发过程中,分子表现出不同的特征,可能来自不同的分布,例如分子的大小(size)、骨架(scaffold)和试验(assay)等不同(图1)。这样的分布外(out-of-distribution, OOD)问题对分子表示学习的泛化性提出了挑战。目前的OOD泛化研究主要集中在欧氏数据上,他们采用了不变性原则(invariance principle),来关注对分布变化保持不变的关键因果因素而忽略虚假部分的重要性。但其在非欧几里得数据上的应用还需要进一步研究和探索。分子通常以图的形式表示,其中原子为节点,键为边,从而保留了丰富的结构信息。复杂的分子图结构使得准确区分不变的因果关系部分和各种虚假的相关关系具有挑战性。
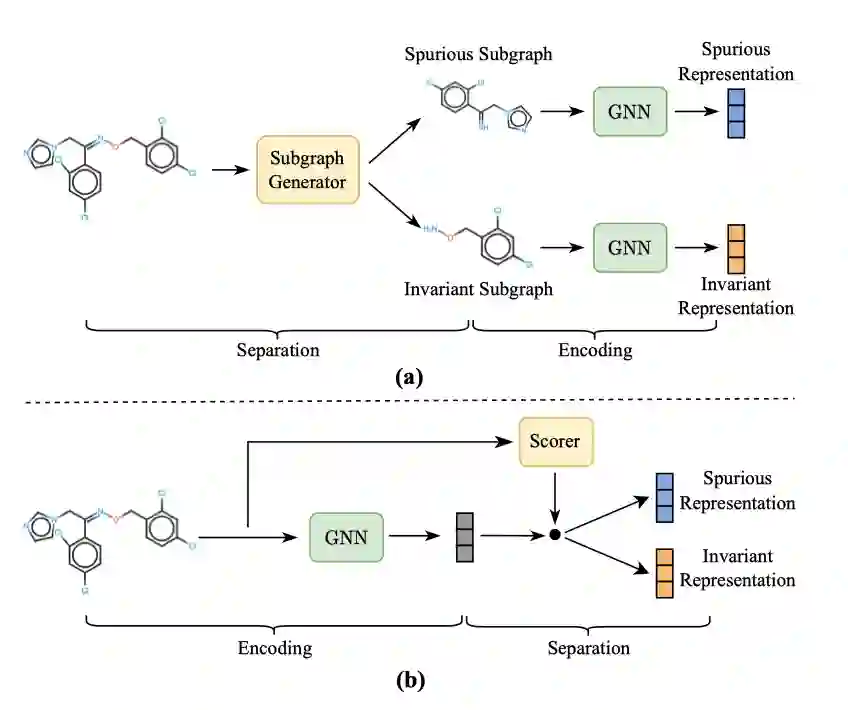
图1 一些研究工作尝试提升分子图表示学习的域外泛化能力。这些研究显式地在分子结构上对图进行划分,以提取不变的子结构,包括在节点、边或官能团的粒度上。这些可以概括为“先分离-后编码”的范式(图2,a)。即首先将图划分为不变部分和虚假部分,然后分别对每个部分进行编码。论文认为,这种做法对于极其复杂的图,例如现实世界中的分子,并不是最佳选择,因为有些复杂的特性无法通过分析分子结构的子集轻易确定。论文提出了“先编码-后分离”的策略(图2,b),论文首先使用图形神经网络(GNN)对分子进行编码(即编码 GNN),然后使用残差向量量化(Residual Vector Quantization)模块来减轻对训练数据分布的过度拟合,同时保持编码器的表现力。然后,利用另一个 GNN 对分子表征进行评分(即评分 GNN),衡量潜空间中每个维度对目标的贡献,从而明确区分不变表征和虚假表征。最后,设计了一个自我监督学习目标,旨在鼓励已识别的不变特征有效保留标签相关信息,同时摒弃环境相关信息。值得注意的是,该目标与任务无关,这意味着提出的方法可以应用于各种任务,包括回归、单标签或多标签分类。
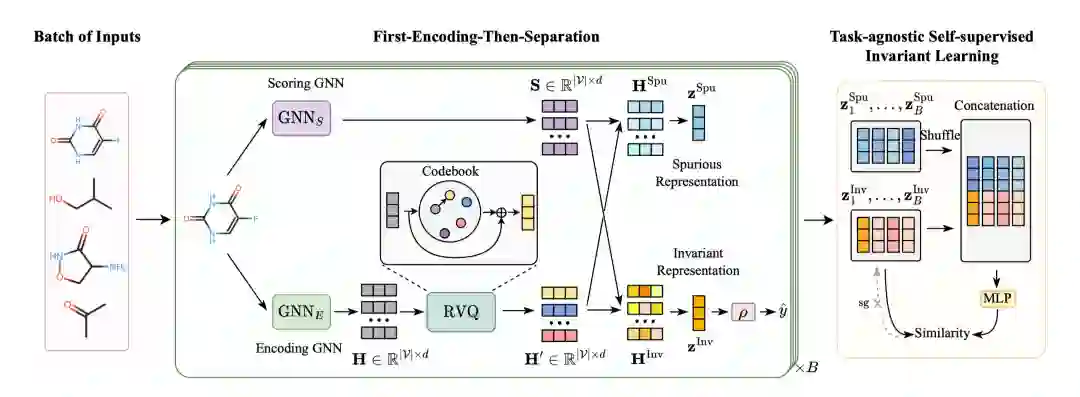
图2 方法
方法主要包括三个部分:(1)使用 编码GNN 和残差矢量量化模块获得潜在空间中的离散分子表示;(2) 通过评分 GNN将分子的表示分为不变部分和虚假部分;(3) 利用任务无关的自监督学习目标优化上述过程。
**使用 GNN 编码器和残差矢量量化模块获得分子表示
使用编码对输入的分子获得原子级别的表示:
定义一个可学习的codebook ,将得到的连续的原子级别的表示离散化:
离散化的操作可以增强泛化能力,缓解分布偏移造成的过拟合问题。然而,使用有限的离散编码替代连续输入也损害了模型的表现力,存在潜在的欠拟合问题。因此加入入残差连接,以在模型泛化和表现力之间取得平衡:
**在结构和特征维度进行区分
使用评分获得区分的得分:
然后,对节点特征应用,在潜在特征空间的结构和特征粒度上捕捉不变和互补的虚假特征:
**学习目标
论文设计了一个自监督的下游任务无关的不变学习目标。首先,对于同一个批次内的样本,对使用扰乱的进行增强:
然后设计一个自监督目标将与增强的作为正样本:
论文还设计了对评分GNN输出的正则项来控制分割过程:
类似于VQ-VAE,使用来控制codebook中的离散向量与GNN编码的连续向量之间的距离。使用获得预测标签,并且结合下游任务作为监督信号:
最终学习目标可表示为:
实验
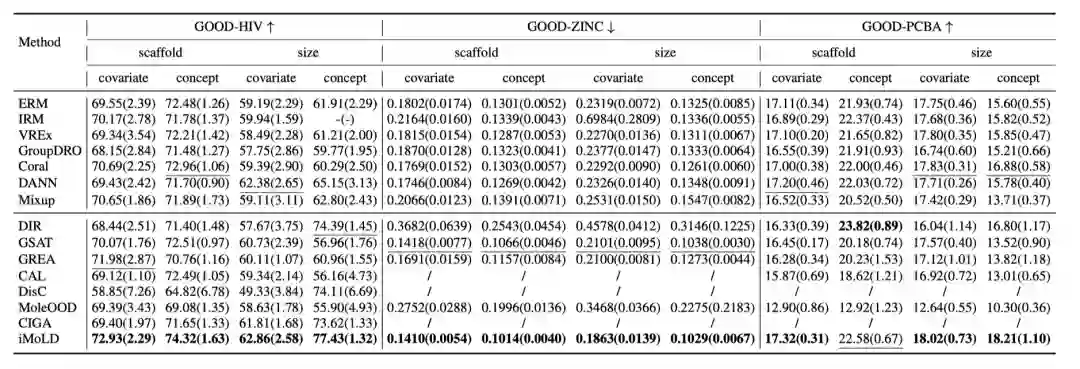
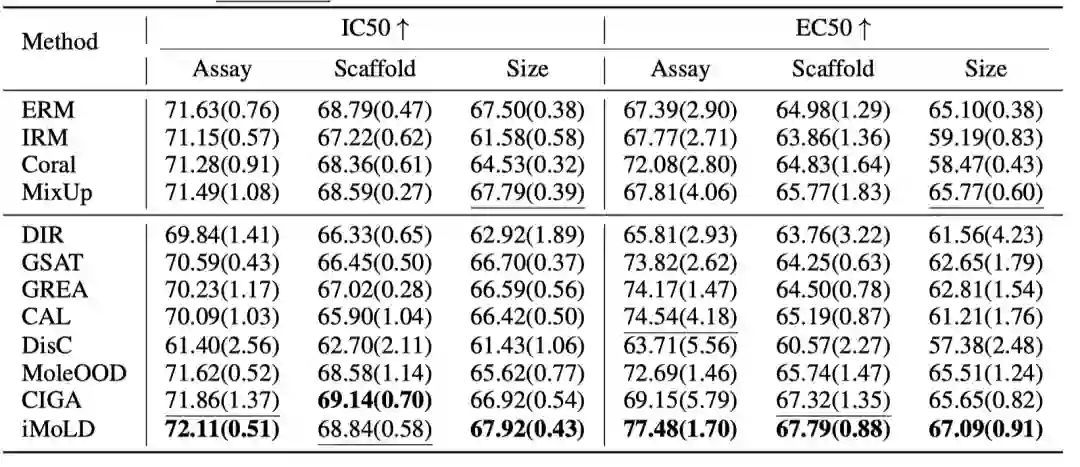
论文在GOOD和DrugOOD两个benchmark上进行了实验,实验结果表明,提出的方法可以应用在所有的下游任务上(单分类:GOODHIV;多分类:GOOD-PCBA;回归:GOOD-ZINC)。并且在 18 个数据集中的 16 个数据集上取得了最佳性能,在另外两个数据集上排名第二。
更多实验结果以及论文内容请阅读论文原文 总结
这项工作提出了一种新的框架,可以学习不变的分子表征,对抗分布偏移。采用了 "先编码后分离 "的策略,将编码 GNN 和残差向量量化相结合,在潜在离散空间中得出分子表征。然后通过学习评分 GNN,从该表示中识别不变特征。此外,论文还设计了一个与任务无关的自监督学习目标,以实现精确的不变性识别和对各种任务的通用性。在真实世界数据集上的广泛实验证明了提出的方法在分子 OOD 问题上的优越性。