**论文题目:**Mol-Instructions: A Large-Scale Biomolecular Instruction Dataset for Large Language Models **本文作者:**方尹、梁孝转、张宁豫、刘康为、黄睿、陈卓、范骁辉、陈华钧(浙江大学)
**发表会议:**ICLR 2024
**论文链接:**https://arxiv.org/pdf/2306.08018.pdf
代码链接:https://github.com/zjunlp/Mol-Instructions 欢迎转载,转载请注明出处
引言
在自然语言处理(NLP)的众多应用场景中,大型语言模型(Large Language Model, LLM)展现了其卓越的文本理解与生成能力,不仅在传统的文本任务上成绩斐然,更在生物学、计算化学、药物研发等跨学科领域证明了其广泛的应用潜力。尽管如此,生物分子研究领域的特殊性—比如专用数据集的缺乏、数据标注的高复杂度、知识的多元化以及表示方式的不统一—仍旧是当前面临的关键挑战。针对这些问题,本文提出Mol-Instructions,这是一个针对生物分子领域各项研究任务定制的指令数据集。
Mol-Instructions的构建
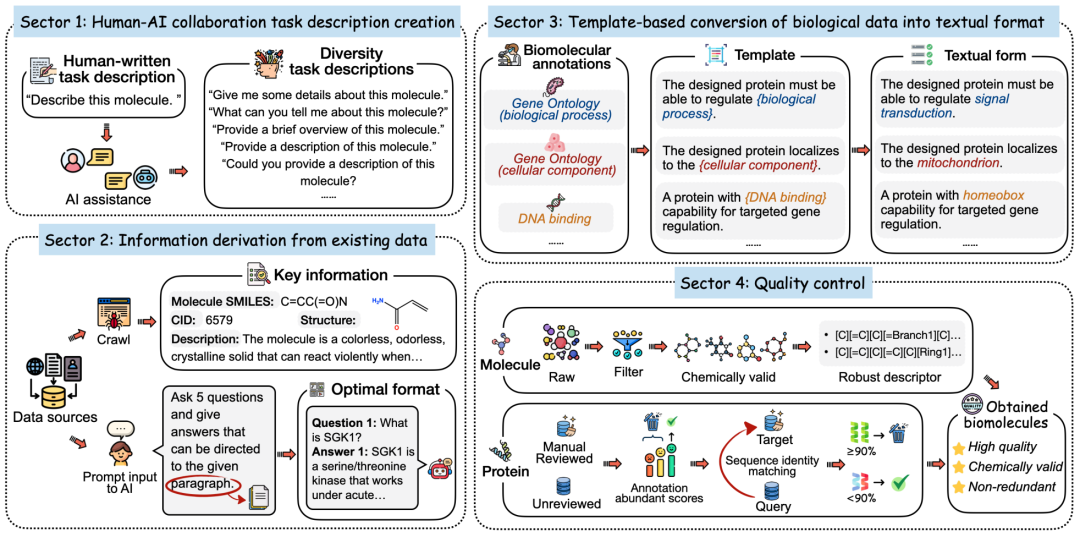
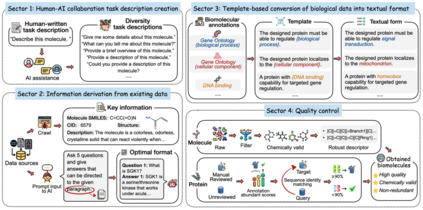
Mol-Instructions的构建流程如下:
- 借助LLM的能力,生成多样化的任务描述,模拟人类需求和表达的多样性。
- 采用多种预处理策略,将现有数据库中的数据转化为可用的指令数据。
- 利用模版将结构化的功能注释转换为易于理解的文本。
- 对小分子和蛋白质序列进行严格的质量控制,以排除化学无效和冗余的序列。
Mol-Instructions概览
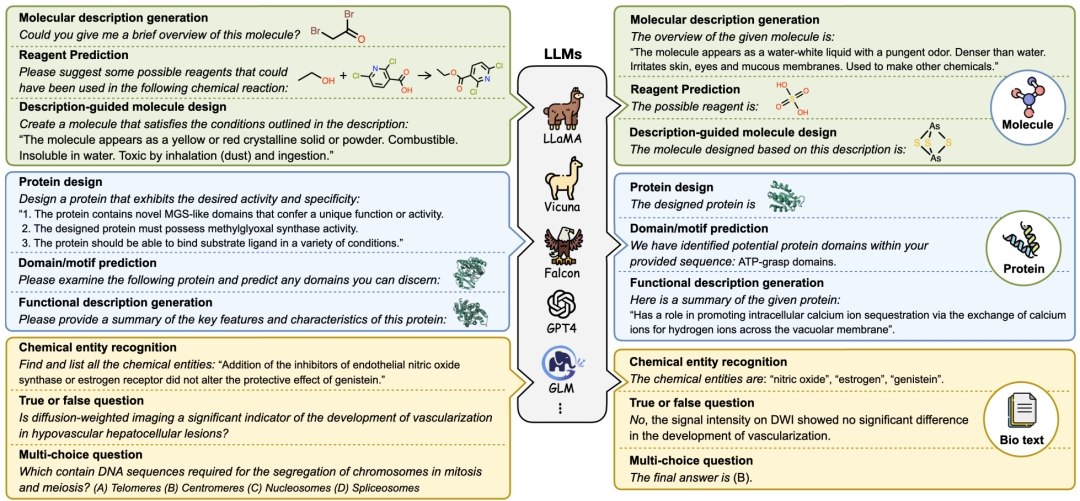
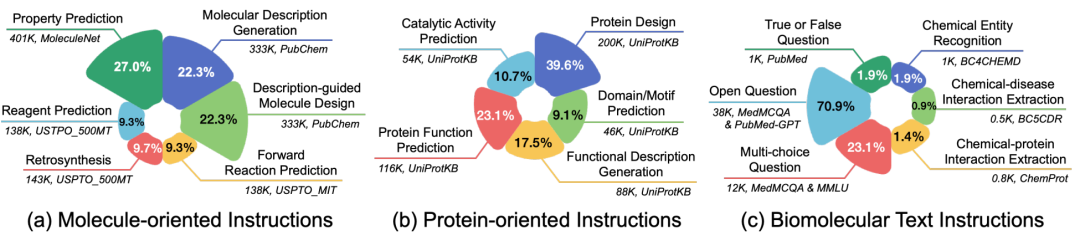
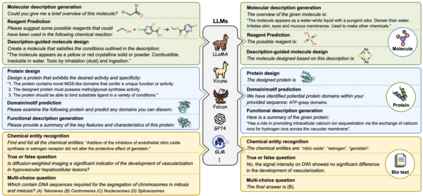
Mol-Instructions数据集共计含2043K条指令数据,覆盖了小分子、蛋白质以及生物分子文本三大领域的17个关键任务,包含了不同复杂度和结构的生物分子及丰富的文本描述。
小分子指令:深度探索小分子的固有属性与行为,研究化学反应和分子设计的核心挑战。理解和预测小分子的化学特性,优化分子设计,提高化学反应预测的准确性和效率。其目标是在化学和药物设计领域加速药物的研发进程,同时降低研发成本。 * 蛋白质指令:主要解决蛋白质设计和功能相关的问题。旨在预测蛋白质结构域、功能及活性,通过文本指令推动蛋白质设计。对于疾病的诊断、治疗以及新药的研发工作具有一定的价值。 * 生物文本指令:侧重于生物信息学和化学信息学领域的自然语言处理任务。旨在从生物医学文献中提取和解析关键信息,支持研究人员快速获取知识、便于进行查询。
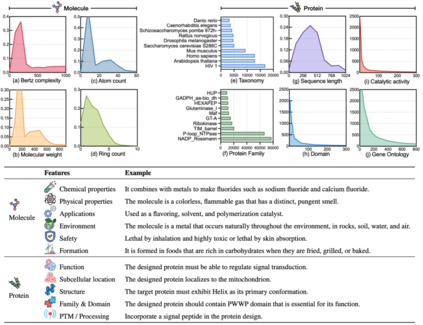
图(a-d)揭示了分子的多维特征。Bertz复杂度是评估分子复杂度的关键指标。分子量反映了分子的大小和复杂性,对众多化学反应具有决定性影响。原子计数揭示了分子的规模和复杂度,进而影响其稳定性和反应性。环计数则提供了结构复杂度和潜在稳定性的视角,对理解化学反应性和生物活性潜力至关重要。图(e-j)探究了蛋白质的特性。图(e-g)体现了蛋白质序列长度的不同分布。根据NCBI分类,这些蛋白质覆盖了丰富的物种和实验菌株,包括13,563个蛋白质家族和643个超家族。图(h-j)关注功能特征,如结构域、基因本体和催化活性的注释。这些数据表现出显著的长尾分布,凸显了推断特定蛋白质功能的挑战,尤其是那些罕见功能的蛋白质。 * 如表格所示,分子设计和蛋白质设计的文本描述提供了多维度的视角,涵盖从基本属性到特定应用场景的广泛特性。
实验分析
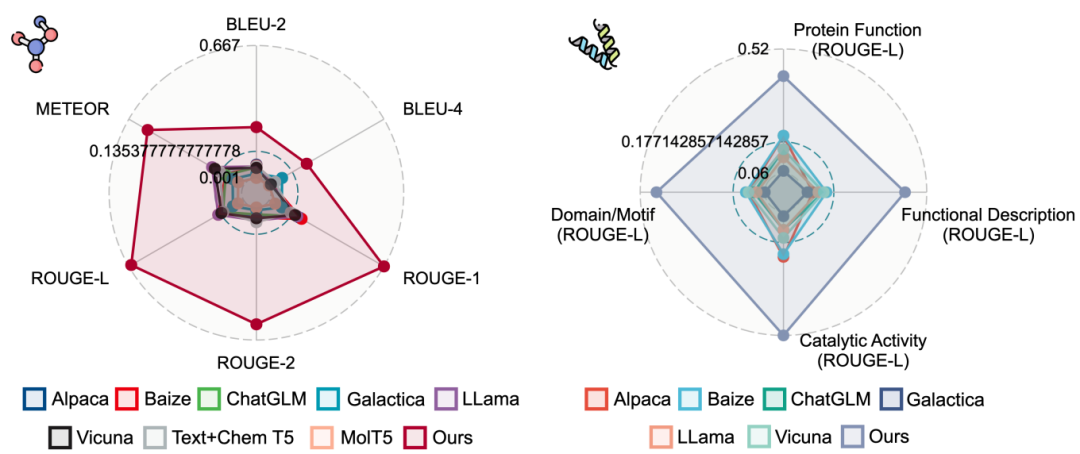
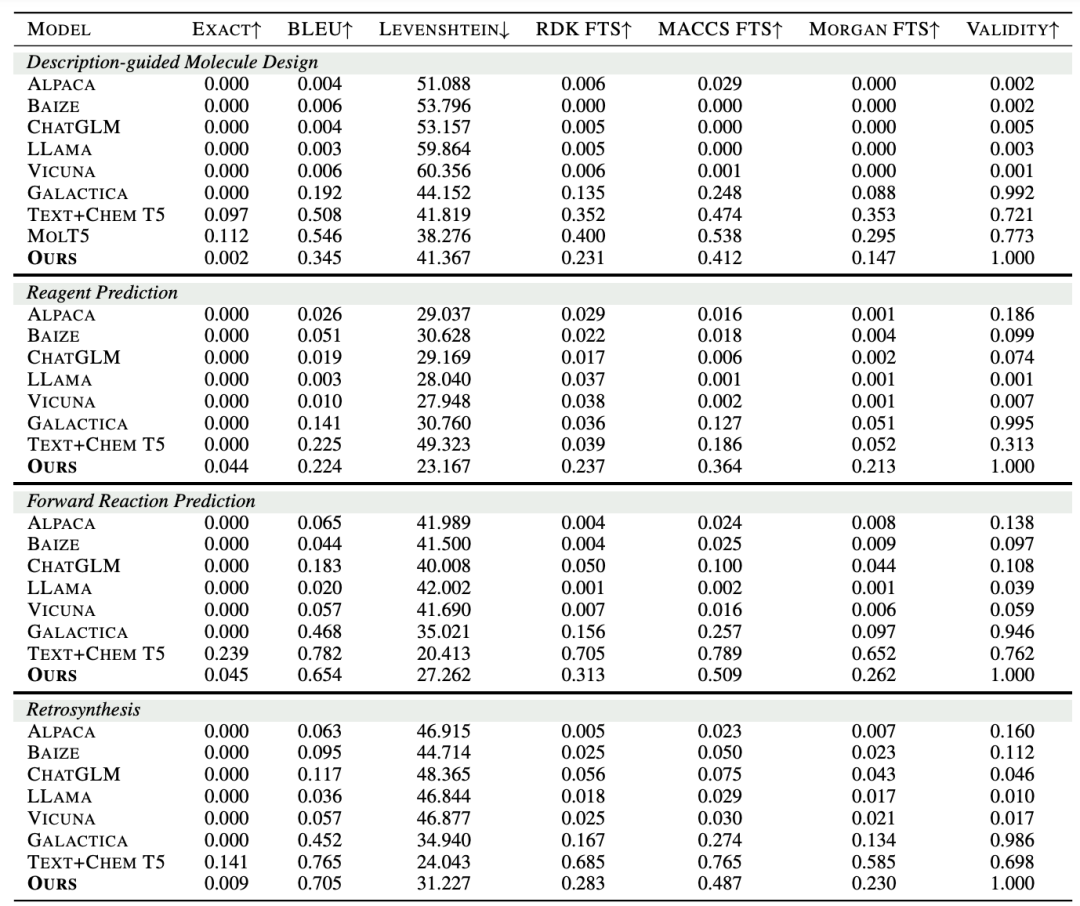
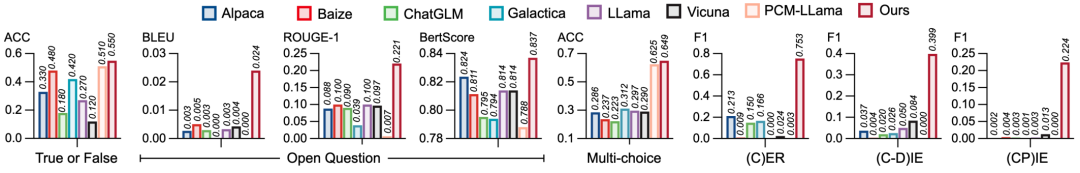
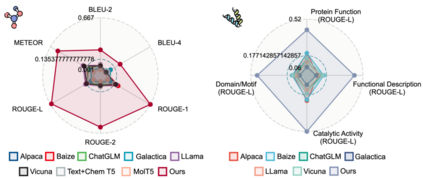
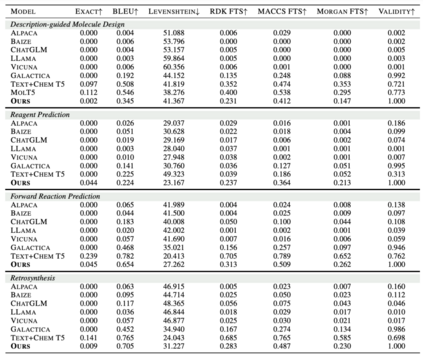
为评估Mol-Instructions对大型语言模型(LLMs)在理解和预测生物分子方面的助益,本文对LLaMA-7B模型进行了指令微调,并从多个角度进行了定量实验分析。实验结果显示,经Mol-Instructions微调的LLM在多种任务上的表现超越了其他大型模型,证明了Mol-Instructions在提升LLMs处理生物分子信息能力方面的关键作用。然而,由于轻量微调过程的局限性,经过微调的LLM在分子生成任务上的表现并未超越现有的专用小型模型。这反映了LLM在追求广泛任务处理能力时,可能会牺牲掉某些专用小模型的专业性。
总结
Mol-Instructions能够有效评估和提升通用LLM从人类语言到生命语言的跨模态理解能力,显著增强了LLM对生物分子的认知。为后续更深入地研究生物分子设计与解决复杂生物学问题提供了重要的数据来源。由于文本与生物分子表示空间的本质差异以及LoRA训练策略的局限性,当前LLM在理解生物分子语言方面的熟练度还未能与其掌握人类语言的能力相媲美。未来,通过扩展模型词表或将生物分子语言视为一种新的模态进行集成,可能是进一步提升LLM在生物分子领域的理解深度和性能表现的关键。