论文名称:MolTailor: Tailoring Chemical Molecular Representation to Specific Tasks via Text Prompts 论文作者:郭昊强,赵森栋,王昊淳,杜晏睿,秦兵 原创作者:郭昊强 论文链接:https://arxiv.org/pdf/2401.11403.pdf
摘要
如今深度学习技术已在药物发现领域得到广泛应用,加速了药物研发速度并降低了研发成本。分子表征学习是该应用的重要基石,对分子性质预测等下游应用具有重要意义。现有的大多数方法仅试图融入更多信息来学习更好的表征。然而,对于特定任务并非所有特征都是同等重要的。忽略这一点将潜在地损害分子表征在下游任务上的训练效率和预测准确性。为了解决这一问题,我们提出一种新颖的方法:该方法将语言模型视为智能体(Agent),将分子预训练模型视为知识库(KB)。语言模型通过理解任务描述,增强分子表征中任务相关特征的权重。因为该方法就像裁缝根据客户的要求定制衣服,所以我们将这种方法称为MolTailor。
引言
本文本提到的分子表征是指分子的向量嵌入,类似于自然语言处理中的词嵌入。 
- 我们提出了MolTailor,这是第一个通过文本提示生成任务相关分子表征的方法,为分子-文本多模态学习提供了新的视角。本方法不仅将文本中的知识注入分子表征,还利用了语言模型的推理能力。
- 我们构建了MT-MTR,一种新颖的分子-文本多模态预训练任务。该任务教会模型遵循文本指令调整分子表征。
- 我们在MoleculeNet[3]的八个子集上进行了全面评估,从而证明了本文方法的有效性。
相关工作
目前的分子-文本多模态预训练模型可以分为双塔和单塔结构:
- 单塔结构:使用语言模型作为进一步预训练的基础。KV-PLM[13]和MolXPT[14]通过定位分子名称将SMILES注入文献文本中,以获得混合语料,并分别使用MLM和LM任务进行预训练。MolT5[15]在分子和文本数据上使用replace corrupted spans任务进行预训练。同时,使用分子与文本描述之间的互译作为下游任务。Text+Chem T5[16]和 ChatMol[17]使用多任务学习进行预训练。GIMLET[18]将分子图和文本作为统一输入,并使用基于指令的有监督属性预测任务进行预训练。
- 双塔结构:MoMu[19]和 MoleculeSTM[20]使用分子-文本描述对进行对比学习。CLAMP[21]使用分子-生物测定对进行对比学习。
此外,ChemCrow[22]通过化学工具增强大型语言模型(LLMs),以完成现实世界中的化学任务。
方法
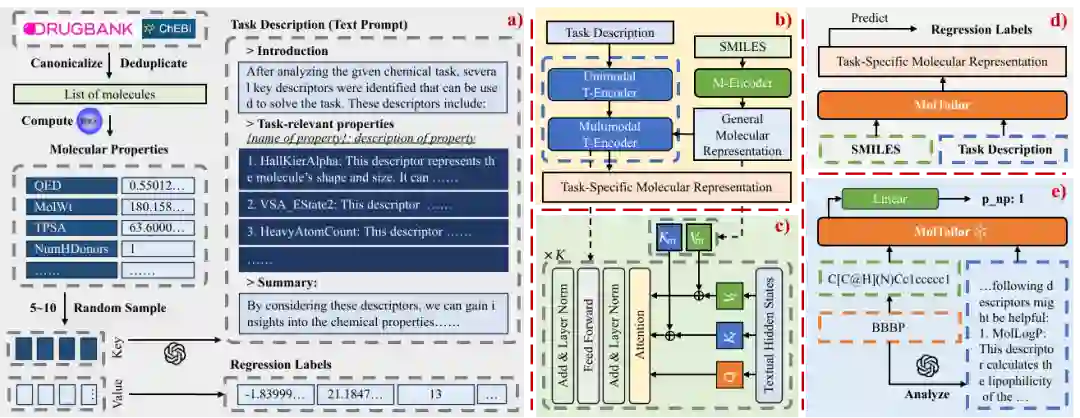
-
a) MT-MTR数据集的构建流程。我们从DrugBank[5]和ChEBI[6]中获取代表性分子,然后使用RDKit计算每个分子的209种属性。对于每个分子,我们从属性集中随机抽取5-10个属性,使用这些属性的名称通过GPT-3.5生成虚拟任务描述(Task Description),并将属性的值作为回归标签(Regression Labels)。
-
b) MolTailor的模型架构。MolTailor包括一个语言预训练模型(T-Encoder)和一个分子预训练模型(M-Encoder)。T-Encoder被分为单模态部分(Unimodal T-encoder, 用于理解任务描述)和多模态部分(Multimodal T-Encoder, 用于调整分子表征)。在实验中,我们使用PubMedBERT[7]与BioLinkBERT[8]分别作为T-Encoder,使用CHEM-BERT[9]与ChemBERTa-2[10]分别作为M-Encoder。
-
c) Multimodal T-Encoder的内部结构。该部分由K个经过修改的Transformer Encoder Block组成。修改后的Block可以同时执行自注意力和交叉注意力操作。形式化证明如下所示[11],其中,表示多头注意力,表示修改后的,表示文本隐状态(Hidden States),表示分子隐状态,表示自注意力操作,表示交叉注意力操作。
-
d) MolTailor的预训练任务。模型需要基于分子和文本提示预测任务描述中提到的属性。预训练使用MSE作为优化目标,计算公式如下,其中代表样本数量,是一个0-1向量,其中值1表示对应的回归标签存在,而0则表示其不存在,表示有效标签的数量,是属性的种类数量(这里), 是回归标签,而 是预测值。
-
e) MolTailor应用于下游任务。对于特定的下游任务,我们首先通过GPT-4分析该任务并生成与预训练相同形式的任务描述,然后将SMILES和任务描述作为输入来预测该任务的标签。预训练和下游任务与GPT交互所使用的模板如下所示:
图3 用于和GPT交互的Prompt模板
实验
实验设置
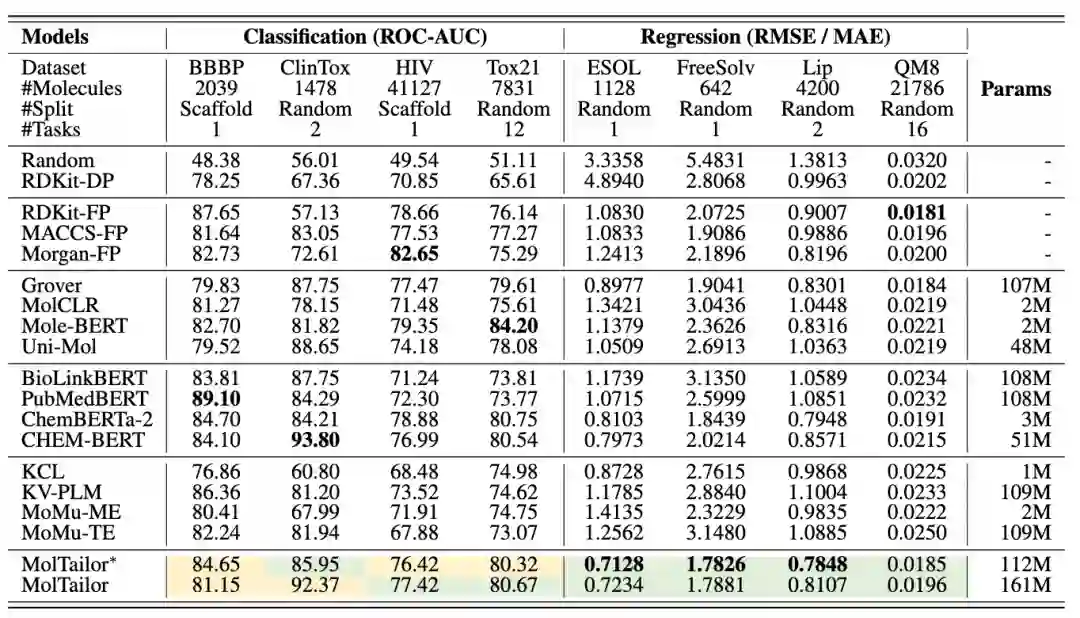
预训练语料。我们使用自行构建的MT-MTR语料进行预训练,该语料包含55,759个三元组。 下游数据集。我们从MoleculeNet[3]中选择了8个代表性任务进行实验,包括4个分类任务(BBBP, ClinTox, HIV, Tox21)和4个回归任务(ESOL, FreeSolv, Lipophilicity, QM8),涵盖生理学、生物物理学、物理化学和量子力学。每个任务使用推荐的分割方法(Random或Scaffold)[3]将数据分为训练/验证/测试集,比例为8:1:1。 评价指标。按照[3]的建议,我们使用ROC-AUC作为分类任务的评估指标。对于回归任务QM8,我们使用MAE,对于其他回归任务,我们使用RMSE。为了确保公平,我们使用Optuna[12]为每个模型在每个任务上搜索10次学习率(LR)。同时,我们重复每个任务3次,并报告平均值和标准差。由于篇幅限制,标准差包含在附录中。此外,为了更好地评估不同模型学习到的分子表征,我们使用Linear Probe设置(我们冻结模型参数后提取下游任务中分子的表征,然后通过一个可学习的线性层将分子表征映射为标签。)进行实验。因此,本文报告的基准实验结果可能与原始论文中的结果有所不同。
实验结果
图4 总实验结果 以上表格是MolTailor与基线模型在MoleculeNet上的实验结果。其中,MoMu的后缀中:ME代表分子编码器(Molecule Encoder),TE代表文本编码器(Text Encoder);MolTailor 表示使用ChemBERTa-2作为M-Encoder,而MolTailor表示使用CHEM-BERT;两种MolTailor均使用PubMedBERT作为T-Encoder,使用BioLinkBERT作为T-Encoder的实验结果在附录中。表格展示了3次实验结果的平均值。由于篇幅限制,标准偏差省略,但可以在附录中查看。表中以加粗字体表示最佳性能。绿色高亮表示MolTailor比作为M-Encoder的Backbone效果要好,而黄色则表示相反情况。 我们评估了由任务描述增强的分子表征是否能在8个任务上提高性能。实验结果表明MolTailor相比基准模型(Baseline)在4个回归任务上取得了性能提升,并在ESOL、FreeSolv和Lipophilicity数据集上取得了最先进(SOTA)的结果。在4个分类任务上,MolTailor的性能表现不一致,在使用CHEM-BERT作为Backbone时,相比于Backbone在HIV和Tox21上有所提升,但在其他两个数据集上表现下降。我们认为这可能是因为预训练任务更偏向于回归任务,这会抑制模型在分类任务上的性能。这也表明分类和回归任务可能需要不同的优化方法。此外,我们尝试将MT-MTR转换为MT-MTC进行预训练,但看到的效果提升并不明显,这表明这这种现象不是由于回归与分类的任务形式导致的。
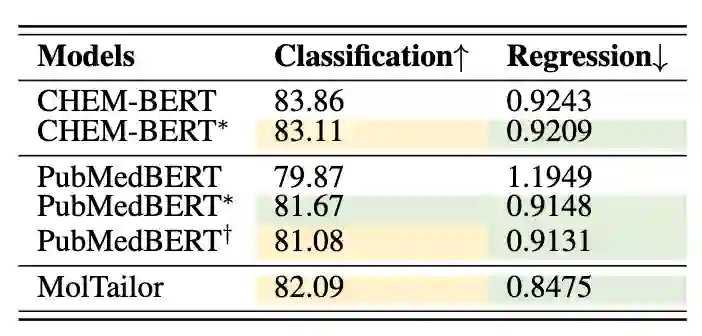
消融实验
为了评估任务描述对模型表现的影响,我们首先移除MT-MTR(这里使用的数据集去除了与下游任务重叠的分子)数据集中的任务描述,得到数据集MT-MTR。然后,使用MT-MTR训练CHEM-BERT和PubMedBERT,得到CHEM-BERT和PubMedBERT模型。接着,我们将MT-MTR数据集中的任务描述拼接在SMILES字符串后,构建数据集MT-MTR,并在此数据集上训练PubMedBERT,获得PubMedBERT。我们将以上模型在MoleculeNet上的性能与MolTailor进行比较,结果如下表所示所示,CHEM-BERT在回归任务上优于未经训练的CHEM-BERT,但在分类任务上表现下降,表明MTR作为预训练任务有利于下游回归任务,但对分类任务有负面影响。此外,PubMedBERT相比于PubMedBERT在回归性能上取得了进一步提升,但分类性能下降;MolTailor相比于CHEM-BERT同样。这表明引入任务描述有助于模型更好地从数据中学习。也就是说,如果构建新标签可以提高模型在分类任务上的性能,那么补充任务描述可以进一步放大这种收益。
图5 消融实验结果
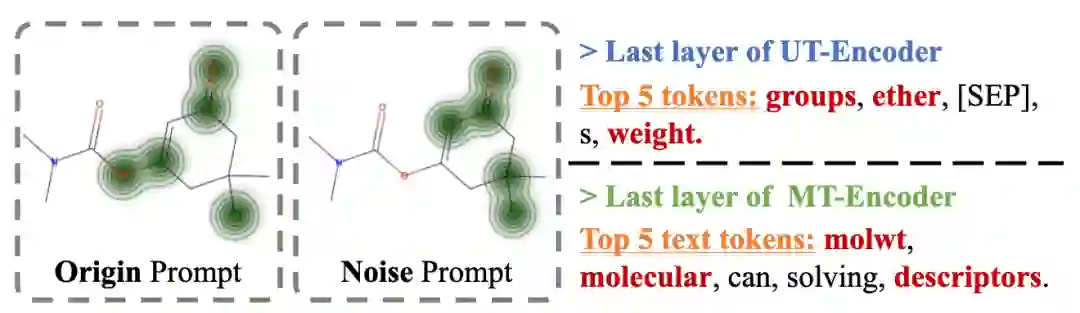
案例研究
图6 注意力可视化。左侧的两个分子图显示了MolTailor在不同文本提示下对分子的关注程度。右侧的文本显示了MolTailor最关注文本提示中的哪些信息 我们分析了UT-Encoder(Unimodal T-Encoder)和MT-Encoder(Multimodal T-Encoder)最后层的注意力矩阵,以探究MolTailor是否关注关键信息。分析结果如上图所示。具体来说,我们选择ESOL(预测分子溶解度)作为的代表性任务。如果模型关注到诸如分子量和极性官能团等决定溶解度的关键信息,则表明关键信息被捕捉到了。 将模型在ESOL上微调后,我们将测试集的SMILES和任务描述输入模型,以获得注意力矩阵 和 。因为我们使用“[CLS]”Token对应的向量预测下游任务标签,所以我们认为与 中“[CLS]”与其余Token之间的注意力值越大,表明模型就越关注那些信息。 在上图中,UT/MT-Encoder最关注的前5个Token中用红色高亮显示的部分与分子量和极性官能团有关,这表明模型确实关注关键文本信息。此外,左侧的两个分子图表示的是:MT-Encoder在不同文本描述(Origin表示上述实验所用的任务描述,Noise表示任务无关的文本信息)下最关注的分子Token。相比于Noise,Origin文本描述下MT-Encoder对像酯和酮这样的极性官能团的关注更多,意味着关键信息被更好地捕捉到了。
总结
总的来说,这项工作为分子表征学习提供了一个新的视角:不仅努力在表征中包含更全面的信息,而且结合上下文信息,通过定制获得更适合特定任务的分子表示。在这项工作中,我们不仅利用了文本模态中隐含的知识,更重要的是,我们尝试利用语言模型的出色推理能力,这在大模型时代将变得更加重要。
参考文献
[1] Rong Y, Bian Y, Xu T, et al. Self-supervised graph transformer on large-scale molecular data[J]. Advances in Neural Information Processing Systems, 2020, 33: 12559-12571. [2] Wang Y, Wang J, Cao Z, et al. Molecular contrastive learning of representations via graph neural networks[J]. Nature Machine Intelligence, 2022, 4(3): 279-287. [3] Wu Z, Ramsundar B, Feinberg E N, et al. MoleculeNet: a benchmark for molecular machine learning[J]. Chemical science, 2018, 9(2): 513-530. [4] Weininger D. SMILES, a chemical language and information system. 1. Introduction to methodology and encoding rules[J]. Journal of chemical information and computer sciences, 1988, 28(1): 31-36. [5] Wishart D S, Feunang Y D, Guo A C, et al. DrugBank 5.0: a major update to the DrugBank database for 2018[J]. Nucleic acids research, 2018, 46(D1): D1074-D1082. [6] Hastings J, Owen G, Dekker A, et al. ChEBI in 2016: Improved services and an expanding collection of metabolites[J]. Nucleic acids research, 2016, 44(D1): D1214-D1219. [7] Gu Y, Tinn R, Cheng H, et al. Domain-specific language model pretraining for biomedical natural language processing[J]. ACM Transactions on Computing for Healthcare (HEALTH), 2021, 3(1): 1-23. [8] Yasunaga M, Leskovec J, Liang P. Linkbert: Pretraining language models with document links[J]. arXiv preprint arXiv:2203.15827, 2022. [9] Kim H, Lee J, Ahn S, et al. A merged molecular representation learning for molecular properties prediction with a web-based service[J]. Scientific Reports, 2021, 11(1): 11028. [10] Ahmad W, Simon E, Chithrananda S, et al. Chemberta-2: Towards chemical foundation models[J]. arXiv preprint arXiv:2209.01712, 2022. [11] Chen X, Zhang N, Li L, et al. Hybrid transformer with multi-level fusion for multimodal knowledge graph completion[C]//Proceedings of the 45th International ACM SIGIR Conference on Research and Development in Information Retrieval. 2022: 904-915. [12] Akiba T, Sano S, Yanase T, et al. Optuna: A next-generation hyperparameter optimization framework[C]//Proceedings of the 25th ACM SIGKDD international conference on knowledge discovery & data mining. 2019: 2623-2631. [13] Zeng Z, Yao Y, Liu Z, et al. A deep-learning system bridging molecule structure and biomedical text with comprehension comparable to human professionals[J]. Nature communications, 2022, 13(1): 862. [14] Liu Z, Zhang W, Xia Y, et al. MolXPT: Wrapping Molecules with Text for Generative Pre-training[J]. arXiv preprint arXiv:2305.10688, 2023. [15] Edwards C, Lai T, Ros K, et al. Translation between molecules and natural language[J]. arXiv preprint arXiv:2204.11817, 2022. [16] Christofidellis D, Giannone G, Born J, et al. Unifying molecular and textual representations via multi-task language modelling[J]. arXiv preprint arXiv:2301.12586, 2023. [17] Zeng Z, Yin B, Wang S, et al. Interactive Molecular Discovery with Natural Language[J]. arXiv preprint arXiv:2306.11976, 2023. [18] Zhao H, Liu S, Ma C, et al. GIMLET: A Unified Graph-Text Model for Instruction-Based Molecule Zero-Shot Learning[J]. bioRxiv, 2023: 2023.05. 30.542904. [19] Su B, Du D, Yang Z, et al. A molecular multimodal foundation model associating molecule graphs with natural language[J]. arXiv preprint arXiv:2209.05481, 2022. [20] Liu S, Nie W, Wang C, et al. Multi-modal molecule structure–text model for text-based retrieval and editing[J]. Nature Machine Intelligence, 2023, 5(12): 1447-1457. [21] Seidl P, Vall A, Hochreiter S, et al. Enhancing activity prediction models in drug discovery with the ability to understand human language[J]. arXiv preprint arXiv:2303.03363, 2023. [22] Bran A M, Cox S, Schilter O, et al. Augmenting large language models with chemistry tools[C]//NeurIPS 2023 AI for Science Workshop. 2023.

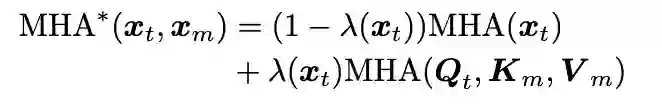
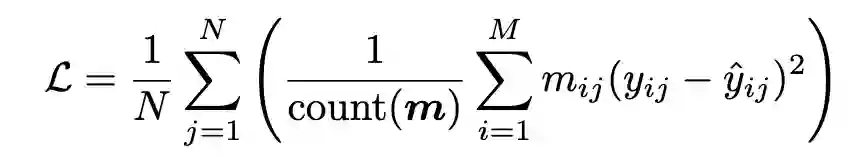
 图3 用于和GPT交互的Prompt模板
图3 用于和GPT交互的Prompt模板