作者 | 邹佳俊 审稿 | 杨崇周 这次为大家报道的是Nature Communiations 上一篇题为”Merging enzymatic and synthetic chemistry with computational synthesis planning” 的文章,来自美国马萨诸塞州麻省理工学院的Connor W. Coley团队。
根据化学反应数据训练的合成规划程序可以设计出感兴趣的新分子的有效路线,但它们利用少见的化学转化的能力有限。这一挑战对于酶促反应来说是严峻的,酶促反应由于其选择性和可持续性而很有价值,但数量很少。文章提出了一种逆合成搜索算法,该算法使用两种用于逆合成的神经网络模型平衡了酶促反应和合成反应的探索,以确定混合合成规划。这种方法通过数千种独特的酶促一步转化扩展了逆合成运动的空间,发现了合成或酶促搜索找不到的分子路线,并为其他路线设计了更短的路线。
介绍
酶促和非酶促合成有机(“合成”)反应可以协同组合以利用各自的独特优势。酶可用于在其他合成过程的关键点引入立体化学。酶还可以催化具有优异区域选择性的反应。结合酶促和合成步骤可以比单独使用一个更加有效。
然而,尽管出现了计算机辅助合成规划 (CASP) 工具,但确定同时使用酶促和合成有机反应步骤的合成路线仍然主要是手动的、直觉驱动的过程。逆合成是通过可能的化学物质空间进行搜索前体。起始位置是目标分子,目标是找到通往可行起始材料的途径。但是对所有前体的强力枚举很快在计算上变得难以处理。CASP 工具在许多方面都有所不同,包括如何提出单步逆合成移动、用于对单步逆合成建议进行排名的模型类别等
数据驱动的 CASP 工具在寻找混合合成路线方面受到限制,因为不同的 CASP 软件工具集被设计用于全合成有机合成规划和全酶合成规划。而酶促 CASP 工具,例如RetroPath使用仅包含酶促反应的反应数据库,将合成和酶促 CASP 搜索相同分子的结果拼接在一起是不够的,因为混合合成计规划的挑战之一是确定一组反应产生可被另一组使用的中间体的路线,从而产生如果一次只考虑一组反应,则可能仍未被发现的路线。
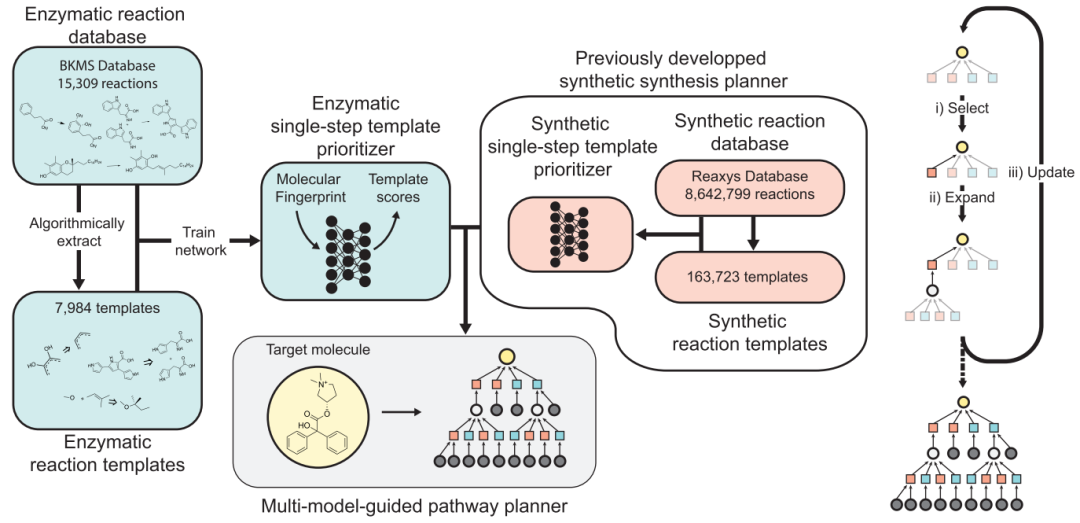
作者介绍了一种合成规划算法来生成多步合成规划,该规划利用了已知合成和酶化学的广度(图 1)。作者使用来自BKMS数据库的酶促反应数据训练了一个基于模板的酶促逆合成神经网络以对单个酶促逆合成步骤进行排序。作者表明,该模型捕获的化学在 ASKCOS的合成化学逆合成模型捕获的化学基础上进行了扩展,添加了4169个独特的模板。然后,设计了一种多步搜索算法,该算法同时使用酶逆合成模型和合成逆合成模型来优先考虑可能的逆合成步骤,从而平衡酶促和合成步骤的探索。作者发现这种混合搜索可以识别分子的路线,而仅使用酶促或合成有机化学无法找到这些分子的路线。此外,混合搜索确定了较短的途径,其中酶促步骤取代了多个合成步骤。最后,作者使用屈大麻酚(dronabinol)和阿福莫特罗(arformoterol)作为案例研究,展示了作者的搜索算法如何建议在其他情况下找不到的有前途的混合合成规划。
图 1.混合合成规划的机器学习方法
结果
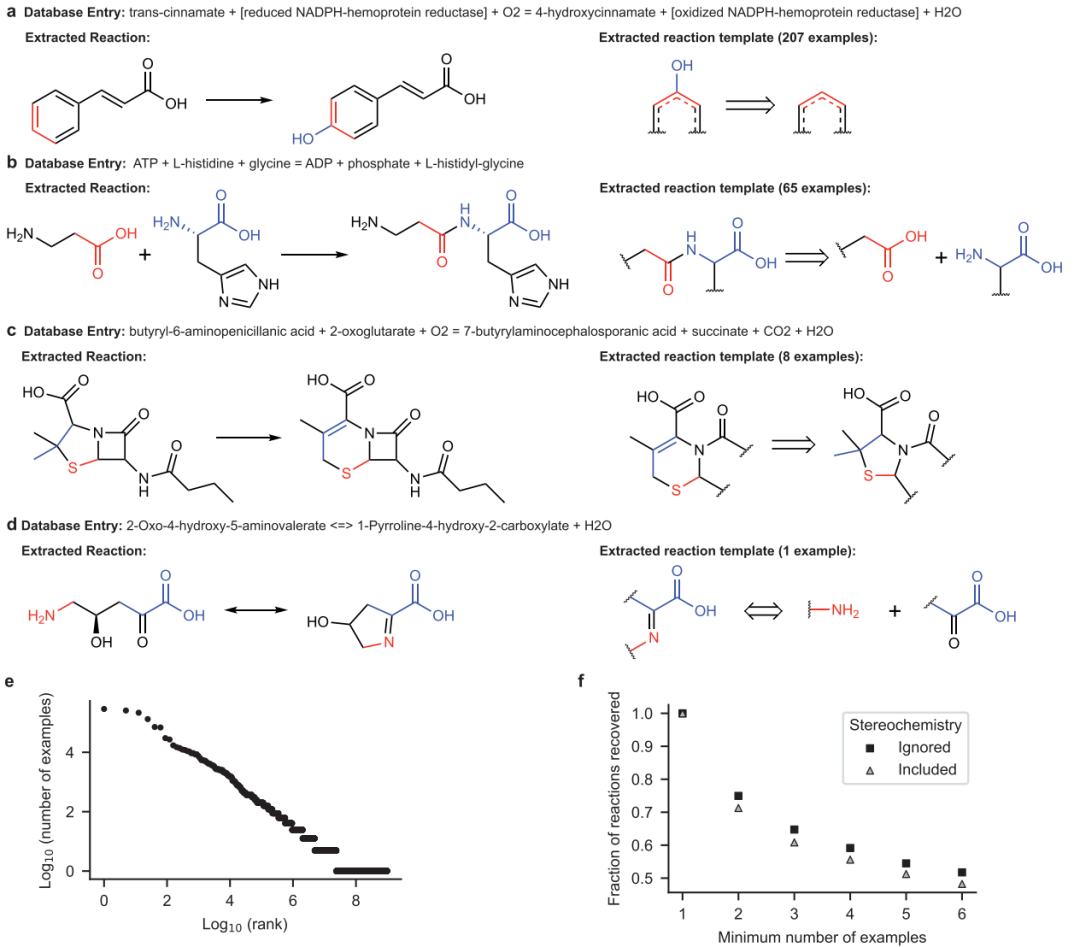
单步酶促逆合成扩增 酶促反应数据集来自BKMS数据库。BKMS包含从BRENDA、京都基因和基因组百科全书 (KEGG)、Metacyc和SABIO-RK聚合的大约37000个酶催化反应。作者通过去除生物辅助因子、将反应转换为标准化的SMILES字符串以及执行原子-原子映射来跟踪反应物中的哪些原子对应于每个反应产物中的哪些原子来处理反应数据。最终数据集包含15309个独特的单一产品原子映射反应SMILES字符串。
使用 RDChiral,总结每个反应化学的反应模板自动从这些原子映射反应中提取为广义 SMARTS字符串(示例如图 2a-d 所示)。总共7984个独特的反应模板足以描述15309个酶促反应。此方法为每个反应生成一个模板,其中包含手性信息和反应中心周围启发式确定的可变数量的上下文。选择这种基于模板的方法是为了在逆合成建议和数据库中的先例反应之间保持联系。
图 2. 从BKMS生化反应数据库中自动提取的反应模板
在作者的酶反应数据集中,近80%的反应模板只有一个先例(图2e)。在作者的酶促反应数据集中,近 80%的反应模板只有一个先例(图 2e)。即使稀有反应被分配了更广义的模板且立体化学信息被删除,如方法部分所述,要求提取的模板具有n>1个先例使过滤后的模板集无法描述近20%的数据库中的反应(图2f)。因此,作者没有根据先例的数量删除模板,以最大限度地提高捕获的酶促反应的多样性。
对于每个酶促反应,数据集中用于合成该分子的产物分子和提取的反应模板用作输入-标签对,以训练多层感知器(MLP)分类模型。训练MLP模板优先排序器模型,以根据产品的分子结构预测使用哪个模板合成训练集中的每个产品。在推论中,给定一个新的分子结构,模板优先排序器为7984个反应模板中的每一个输出一个softmax归一化分数(可以解释为最佳逆合成移动的概率)。
比较酶促转化和合成转化 合成有机化学比已知的酶促化学能够实现更广泛的转化。然而,酶是催化不同的反应还是仅仅催化具有提高的特异性和效率的反应并不明显。作者试图更好地了解Reaxys数据集中的反应模板捕获了BKMS数据集中的反应部分,以及在逆合成搜索中包括酶促反应是否有可能扩大可访问的化学空间。
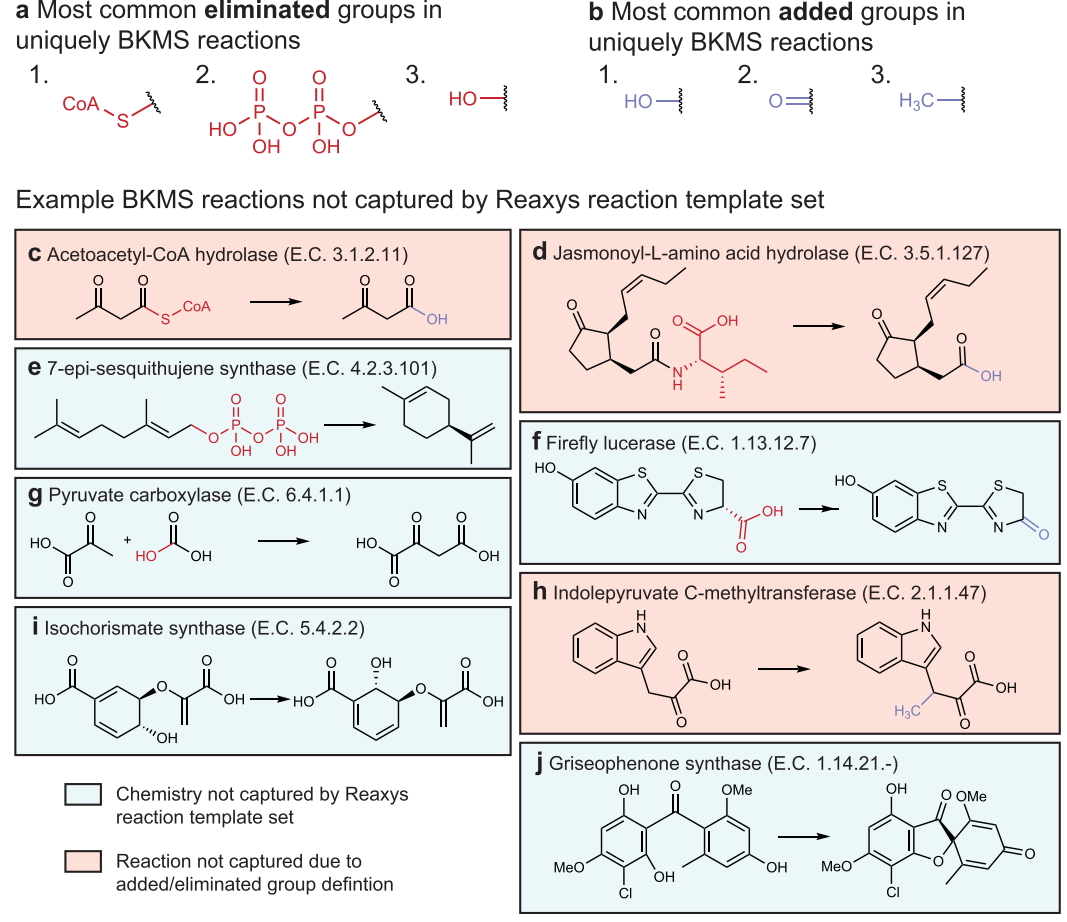
为了确定 BKMS 的哪些反应包含“独特”化学性质,作者评估了Reaxys的任何合成反应模板是否可以在给定产物分子的情况下再现相同的反应物。为此,所有合成反应模板都应用于 BKMS 数据集中的每个产物分子。如果任何反应模板复制了原始反应物分子,则酶促反应被标记为已恢复,因此不是唯一的。在酶促反应数据库中的14601个单步、非自发、非通用反应中,909个是用合成反应模板回收的(不考虑电荷或立体化学)。酶催化剂可以为这些过程提供增强的选择性,但化学转化可以在没有酶的情况下实现。
在剩余的5506个酶促反应中(对应于4169个独特的反应模板),其中一些可能可以通过合成有机化学来实现。反应定义中省略了某些试剂、辅助因子和离去基团,因此可以将反应建模为单一产物。这是执行迭代逆向合成时所必需的。
在Reaxys数据集中,在代表硫酯水解的反应规则中,酰基被视为被消除的基团,而保留的产物是硫醇,而在这项工作中,由于作者自动识别常见的生物辅助因子,硫醇被定义为被消除的基团并保留酰基(图3c)。这更符合CoA在生化反应中的作用。消除和添加组的存在混淆了跨数据集的化学转化的自动比较,因为它使得真正独特的化学与数据集准备期间所做选择的结果无法区分(图 3c-h)。
为了估计唯一酶变换的数量的较低界限,作者确定了在反应物和产品之间没有添加或丢失重原子的3466酶反应,968个反应无法由Reaxys反应模板来描述,对应于824个唯一的酶反应模板。部分由于放置这些限制,这些主要是单分子模板,包括氧化、还原、异构化等。作者举了两个例子,同极胆碱合成酶和格里索酮合成酶的反应在图3i,j。这表明这种酶扩大了合成化学家工具箱中可能的有机转换的范围,不仅仅为现有反应提供替代条件。
图 3. 合成反应组和酶促反应组的比较
由酶和合成模型引导的混合路线搜索 为了有效地探索逆合成搜索空间,作者扩展了ASKCOS中实现的树搜索算法。这项工作中开发的算法可以使用任意多个模型来指导搜索。通过迭代选择、更新和扩展构建以输入目标分子为根的搜索树(图 1b)。在扩展步骤中,作者的算法使用相应的模板优先排序模型对酶模板和合成化学模板进行评分。使用softmax函数对分数进行归一化。然后根据这些分数组合和排序不同的模板集。这意味着在逆合成扩展的每一步,都可以选择来自任一模板集的动作。
多模型搜索算法直接比较模板优先级模型的分数,以决定是否探索合成步骤或酶步骤。为了识别混合通路,该策略依赖于正确缩放的两个模型的分数(概率)。作者证明了他的模型是这样,通过比较两个外部测试集的模型第1推荐分数:来自MOSES数据集的48869个小有机分子和45035分子注释为生物(天然产品)的ZINC目录,这些分子在训练中没有被任何模型看见。酶反应模板是88%的小有机分子和96%的天然产品分子的前3个推荐中。相反,合成反应模板是99%的小有机分子和95%的天然产品分子的前三个推荐(图4a,b)。因此,对于这些集合中的大多数分子来说,合成和酶步骤都将在路径搜索中考虑至少三种可能的移动从分子中,允许算法在选择适当的搜索参数时找到混合计划。
当比较两个模型在不同化学空间上的分数时,会出现一种直观、轻微的偏差。对于有机小分子,顶级逆合成方案更可能是合成的而不是酶促的(图 4c,e)。相反,对于天然产物,顶级逆合成建议更可能是酶促的(图 4d,f)。这种趋势表明,模型显示出一种(合理的)偏差,导致酶化学优先考虑在结构上与天然产物更相似的分子。
图 4. 合成化学和酶促一步逆合成模型的输出分数分布
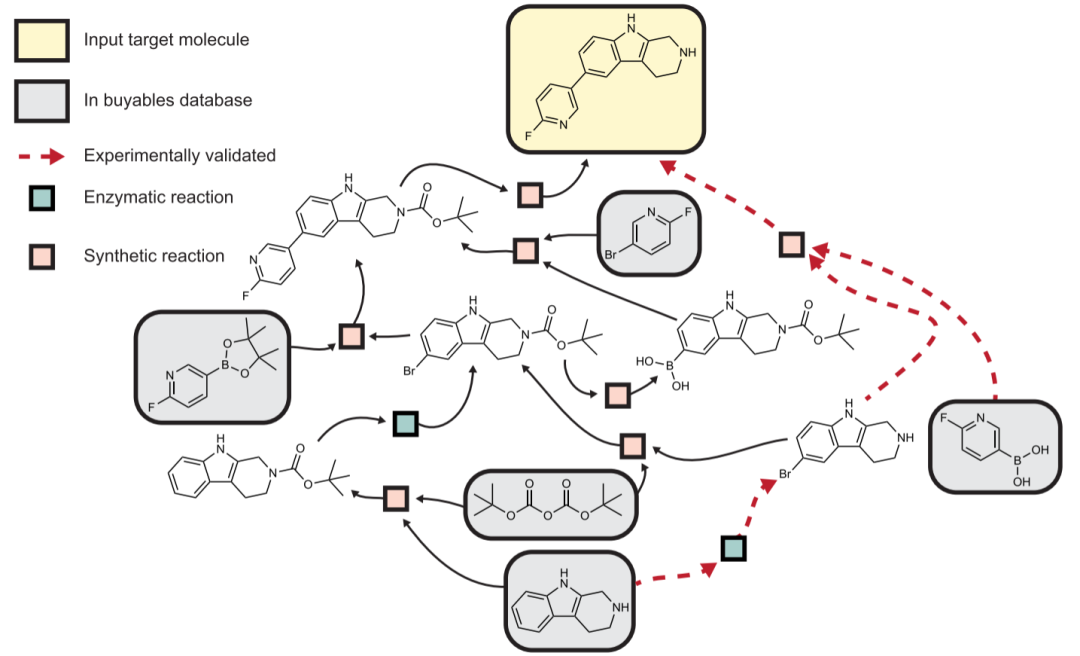
作者证明两种模型的相互作用如何回收如通过RebH合成的氟吡啶基色氨酸的杂交合成。到达可行起始材料的复合路径示于图5中,其中允许的起始材料是来自电子分子和Sigma-Aldrich的一组可购买的化合物。实验路径已经恢复,并在图中突出显示。示出的任何酶反应都不存在于模型的约束数据中,这意味着模型能够概括到未见产物和中间体。酶促芳基溴化模板可回溯至属于EC类1.14.19.55、1.14.19.58和1.97.1的反应数据库中的8种反应。数据库中最相似的反应是色氨酸的溴化。该反应的Brenda条目指向PyrH的Uniprot条目,所述PyrH是黄色素依赖性色氨酸卤素酶,与RebH使用的黄素依赖性色氨酸卤素酶共有机制、反应和38%序列同一性。
图 5. 氟吡啶基色氨酸的混合途径搜索结果示例
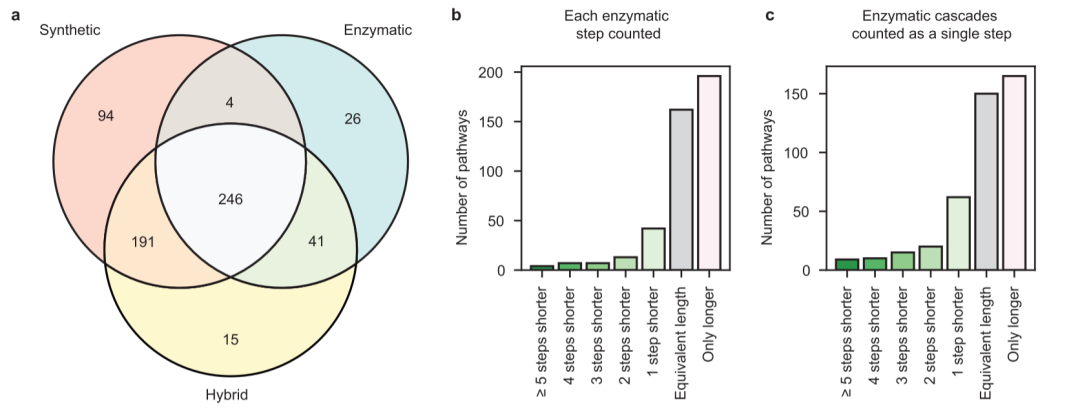
为了更好地了解作者的多模型算法的搜索空间与单模型算法的搜索空间相比,作者使用仅由合成的、只是酶促的或两种模板优先排序模型。他们比较了每次搜索能够识别合成途径的分子数量和每个目标分子发现的最短合成途径中的步骤数(图 6)。
在ZINC的1000个分子中,给出了相同的搜索参数,包括墙壁时间,酶合成规划器找到了317条通往路径,合成规划器找到了535条通道的途径,混合动力规划器找到了493条通往路径(图6a)。酶路径规划者寻找最小数量的分子路径的路径预计,因为酶模板集代表了最有限的转化空间。混合动力规划师找到的化合物比合成规划师少的路线,这并不完全令人惊讶;混合动力道规划师原则上可以找到其他两名规划师发现的超集路径,但在执行时间限制的搜索时不会如此。在搜索中包含更多的模板增加了搜索中考虑的可能移动的数量,并可以引领搜索算法在非生产的方向。然而,当将达到的目标与合成搜索和混合搜索进行比较时,混合搜索找到了56个分子的路线,合成搜索中没有找到其中的路线。其中,如果没有BKMS中的反应模板,11个分子的所有路线将不可能实现,六个分子需要在824个唯一酶反应的受限集合中存在的转换。找到了15个分子的路由,没有路由基于任何单独策略。这表明混合搜索引导与单个模型搜索不同的反合合成搜索空间,其方式有利于某些分子靶点。
图 6. 与混合动力和单模型搜索找到的路线比较 文章中作者对屈大麻酚(dronabinol)和阿福莫特罗(arformoterol)进行了案例研究,详细过程在论文原文章中查看。 总结与讨论
作者展示了一种逆合成计划的混合方法,该方法通过酶促和合成步骤生成有前途的合成计划,以达到复杂的分子目标。在单个逆向合成树搜索中部署多个优先排序模型被证明可以平衡酶促和合成反应建议,以发现具有两组反应的途径。返回的混合途径可以探索单独使用合成化学或酶化学无法获得的分子中间体。
通过比较 BKMS 反应数据集和先前从 Reaxys 中提取的反应模板集,作者发现酶促反应包括4196个独特的转化,而合成数据集则没有。虽然定义独特转化的含义并不简单,但酶催化剂似乎可以使化学转化在一个步骤中发生,而非酶促反应条件则不能。这表明酶在有机合成化学中的作用不仅在于以卓越的选择性进行反应,而且在于扩大可及分子的空间。
屈大麻酚和阿莫特罗的个案研究说明了独特的酶化学可如何解锁从新型构建基块或中间体到目标化合物的途径。这些案例研究还说明了基于模板的回溯合成模型如何可能暗示酶转化,这可能需要酶工程或筛选在实验室中实施,即酶对扩展生物催化工具箱的新型底物的创新应用。
这项工作中的模型是数据驱动的,因此用于平衡合成和酶促合成步骤的相同工作流程可以直接应用于新数据集。理论上不能保证新模型之间会观察到与本研究中相同的理想平衡,但是将softmax变换应用于每个模型的分数会限制模型的范围输出,以及与训练示例相似的输入的更高模型置信度的经验趋势似乎可能会持续存在。然而,如果存在已知的期望结果,可以引入平衡参数来手动调整模型的相对分数。另一个限制是,在搜索中组合多个模型会扩展搜索空间,因此在给定相同的时间限制的情况下,混合搜索可能无法找到由单个模型引导的搜索会找到的路径。这进一步激发了探索新技术的动力,例如强化学习以平衡模型建议。
作者相信像这样的混合CASP方法将加速新的高效合成路线的识别和开发。酶可以催化某些原本不可能发生的转化,并提高其他转化的选择性和效率,而合成化学提供了更广泛、互补的工具包。使用作者的算法确定应用酶化学来访问新目标的机会可以与高通量筛选和酶进化方面的实验努力协同工作,以发现新的生物催化剂。 方法
文章中有关的数学公式定义、推导在该部分中,感兴趣的朋友可以访问原文进一步学习。 参考资料 Levin, I., Liu, M., Voigt, C.A. et al. Merging enzymatic and synthetic chemistry with computational synthesis planning. Nat Commun 13, 7747 (2022). https://doi.org/10.1038/s41467-022-35422-y