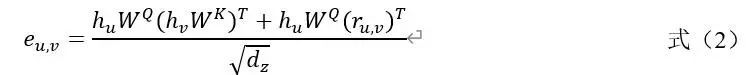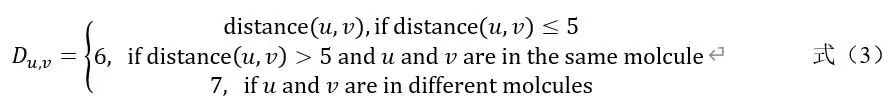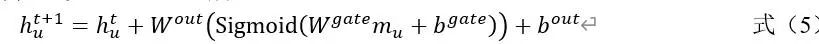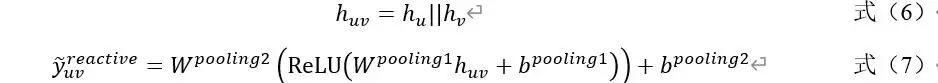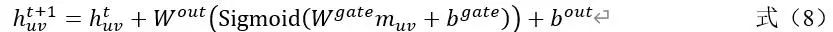编译 | 程宁 本文介绍由韩国科学技术院的Shuan Chen与Yousung Jung发表于Nature Machine Intelligence的研究工作。作者提出了一种基于原子映射的通用反应模板GRT用于描述有机反应,并在此基础上提出LocalTransform图注意力网络来预测分子之间的有机反应。由于模板仅描述反应前后原子结构变化,大大减少了反应规则的数量,实验结果表明LocalTransform准确率优于现有先进模型。除了通用反应模板内置的可解释性外,score–accuracy相关性也使得模型具有可解释性。
1 简介
预测有机反应结果是化学中的一个基本问题,利用计算机模型精准预测有机反应能够大大加快新分子的设计过程。目前已存在一些机器学习方法来预测有机反应,然而现有模型大多利用化学语言或者分子序列来对有机反应进行预测,这与资深化学家通过识别具体反应的子结构并通过已有化学知识判断有机反应有很大区别。
针对上述问题作者提出了一种新的描述有机反应的模板GRT(Generalized Reaction Template),并在此基础上提出预测有机反应的图神经网络模型LocalTransform。作者的贡献如下: (1)提出了一种描述有机反应的通用模板GRT。针对USPTO-480k数据集,GRT能描述99.7%的有机反应,对于top-100的有机反应覆盖率能够达到94.6%。
(2)基于GRT提出了LocalTransform图神经网络以预测分子之间的有机反应,实验结果表明该方法优于以往基于图的方法。 (3)由于模型是基于分类的方法,具有很强的score–accuracy相关性,因此具有一定可解释性。
2 方法
2.1 GRT通用反应模板 作者认为先前基于模板的方法性能较低的原因是模板中包含太多详细信息从而导致其低覆盖率和低扩展性。因此作者提出了仅描述反应前后原子结构变化的反应模板(GRT)用于描述有机反应。
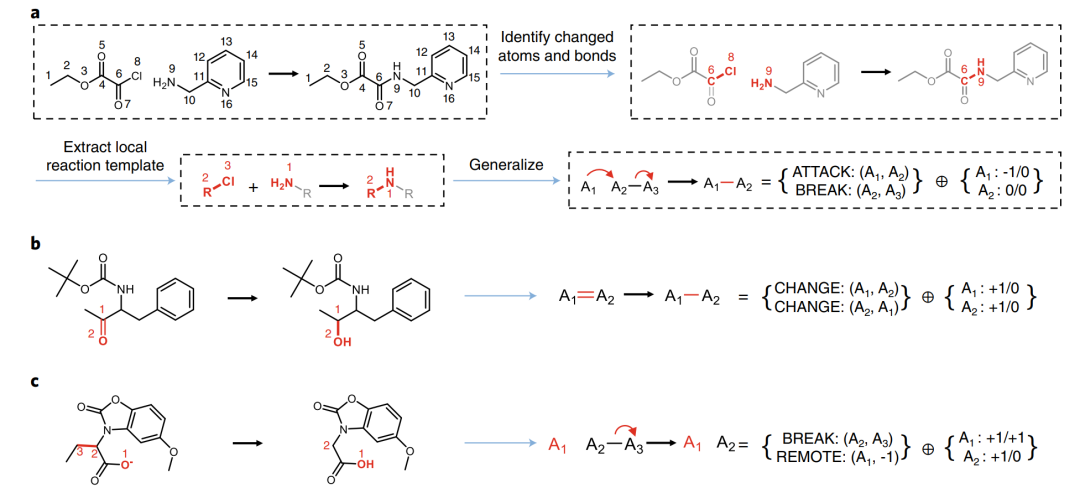
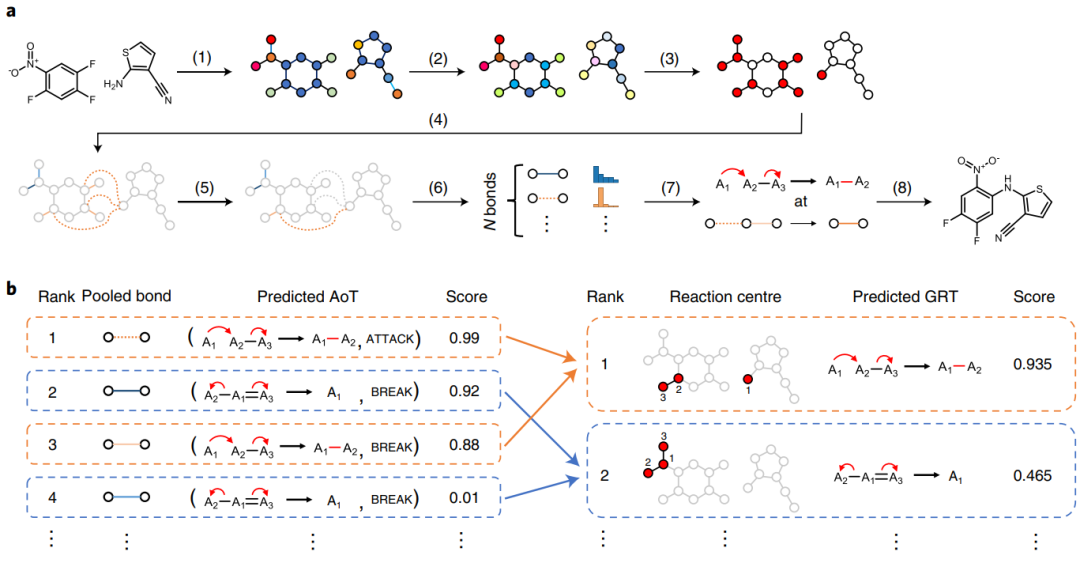
图1展示了GRT模板提取过程示例,首先根据反应前后分子变化抽象出具体反应位置,其中原子由通用原子符号“A”表示,然后根据分子具体变化类型分为四类:ATTACK、CHANGE、BREAK和REMOTE,并标注电子的移动情况。不同模板可能涉及不同数量的变化,在特定反应中心完成所选GRT所需的变化称为AoT。
图1 GRT提取过程
2.2 LocalTransform模型 ①分子图的构建。每组反应物通过两个分子图构成:特征图Gf和密度图Gd,分别记为Gf = (Vf, Ef)和Gd=(Vd, Ed),V表示原子,E表示化学键。Gf包含现有的原子特征和化学键特征,通过现有的化学键或虚键来连接可能的原子对,用于潜在的键形成、断裂和变化的预测。两个图都可通过DGL-LifeSci库来构建。
②提取分子图中每个原子的邻居特征。使用基于门控循环单元的MPNN对原子特征进行三次迭代更新,如公式(1),hu其中表示原子u的特征,hv表示相邻原子特征,huv表示相邻键的特征,最终得到原子在处更新后的特征。
③全局原子感知注意力机制。为了使反应物中的原子能够感知潜在的相互作用的电子,设计了一个基于Transformer的多头注意力机制。原子u和v之间注意力评分如公式(2)所示。
其中Wq和Wk分别是查询权重和键权重,T是转置运算符,dz为归一化因子,ru,v为相对位置embedding,可以通过公式(3)获得Du,v。
从原子到原子的消息通过公式(4)传递。
上述为非线性变化的权值和偏差,激活函数为ReLU。 最后通过连接门控单元转换原子特征。如公式(5)所示。
在MPNN学习到原子的局部环境后,反应物中的所有原子通过距离感知全局注意力网络进一步寻找与其他原子的关系信息,以进行潜在反应。
④组合特征构建和池化。将稠密图中有向图的已有键和虚键的特征拼接成一个原子特征,如公式(6)所示。并将其作为神经网络的输入,如公式(7)所示。
⑤全局注意力网络。键的特征被输入到另一个全局注意力神经网络,通过进一步学习聚合反应键之间的关系信息来对每个反应键的反应性进行排名。如公式(8)所示。
⑥AoT预测。使用双层神经网络对每个键的AoT进行预测,并通过SoftMax将最终预测结果转换为每个键的AoT得分,如公式(9)、(10)所示。训练LocalTransform的Loss为反应池化和AoT预测的交叉熵损失之和,如公式(11)所示。
⑦AoT排序和采集。预测出AoT的分数后,将所有键的预测AoT按分数排序,逐一收集预测AoT,直到收集到的AoT满足预测GRT的所需要的反应变化。最后,将预测的GRT应用于预测的反应中心。
图2 LocalTransform模型预测流程
3 实验
3.1 在USPTO-480k数据集的预测结果 表1展示了USPTO-480k数据集上本文模型以及其他模型的预测结果,实验表明除了top-1之外,LocalTransform模型均优于其他模型。虽然在top-1预测中Chenformer的效果比文中模型精度提高了0.5%,但在其他top-k的预测精度均小于LocalTransform。
表1混合预测场景下USPTO-480k数据集的top-k精确匹配精度
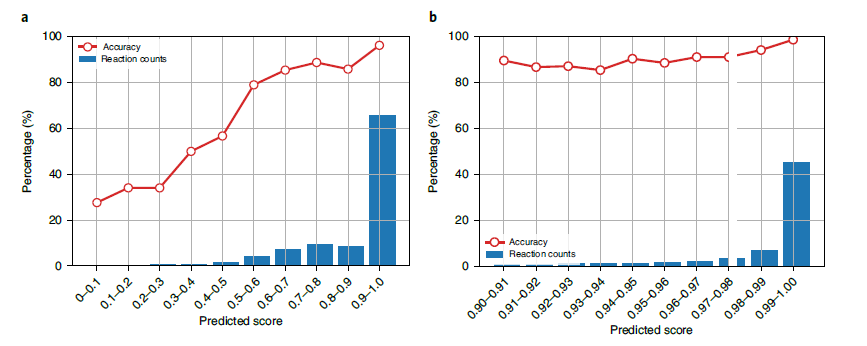
此外,模型会为每个预测对象生成分数作为其排名,预测分数可以解释为给定有机反应的预测结果的置信度。由图3可以看出模型预测分数越高,两种分子之间具有实际反应的概率越高。统计结果显示对于预测分数大于0.99的例子来说,其反应匹配正确率高达98.4%。
图3 top-1精确匹配准确度和作为预测分数函数的反应百分比
对于预测分数高于0.99但是匹配失败的例子,作者表示模型预测了一些更加合理或更有可能的反应,图5中有4个有机反应被Schwaller等人认为是由于数据集本身的错误。
图4 模型预测分数大于0.99但匹配失败的例子
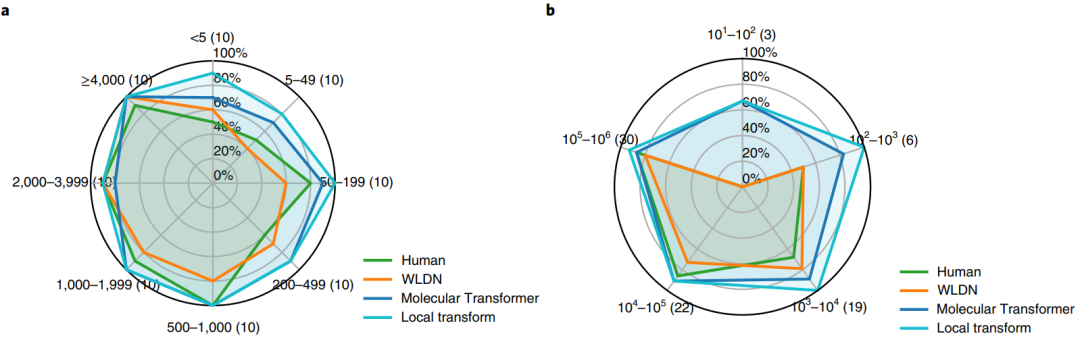
3.2 与专家对比结果 从测试集中获取80个稀有有机反应,人类专家和WLDN、Molecular Transformer、LocalTransform模型同时进行预测,参与实验的11位专家中选取得分最高的人,预测结果如图5a所示,可以看出LocalTranform取得了最好的结果,正确预测了 80 个反应中的 75 个。基于文中提出的GRT再次对数据进行分类,LocalTranform依然取得了最好的结果,如图5b所示。
图5 不同模型与专家的预测结果
4 总结
在这项工作中,作者首先提出了一种用于描述有机反应的通用反应模板GRT,该模板能覆盖大部分有机反应,具有极大的灵活性与简便性;然后基于此提出LocalTransform模型,通过识别反应中心并利用GRTs来预测有机反应;最后通过实验证明了该模型的有效性,并分析了识别错误的例子,指出模型很大程度上受限于反应映射的质量以及数据集中反应的多样性,预期未来通过使用具有更高质量原子映射方法的数据集以改进模型。
参考资料 论文链接: https://doi.org/10.1038/s42256-022-00526-z 项目源码链接: https://github.com/kaist-amsg/LocalTransform40 数据链接:USPTO-480k数据集: https://github.com/wengong-jin/nips17-rexgen13 图数据: https://github.com/kaist-amsg/LocalTransform/releases/tag/raw_data40