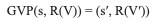Facebook:结合AF2数据,用几何向量感知机+图神经网络/Transformer新方法预测逆折叠
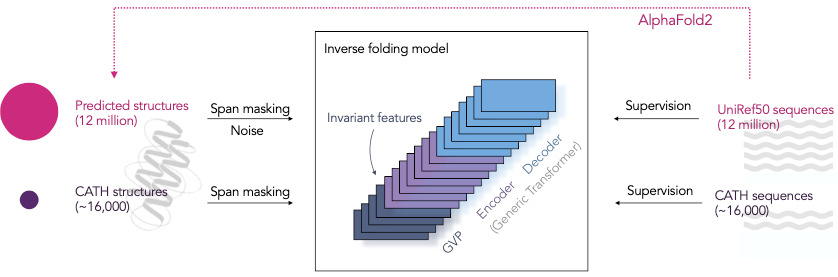
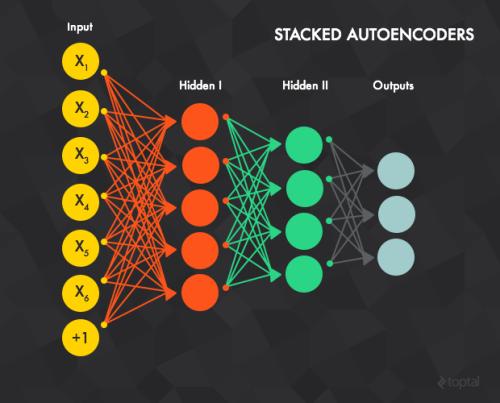
近日,Facebook利用AlphaFold2产生的数据,采用几何不变处理层的seq2seq的模型,在蛋白质骨架结构数据上实现了51%的序列复现,对于包埋残基的复现率达到72%,总体上比现有方法提高了近10%。
从蛋白质骨架结构预测序列的问题,也称为反向折叠或固定骨架设计。使用自回归encoder-decoder将逆折叠转为序列到序列的问题。其中模型的任务是从蛋白质骨架的坐标中恢复蛋白质天然序列原子。
Facebook团队究竟做了什么使得蛋白从头设计的准确率比现有方法提高了近10个百分点。其实从上述描述,可以看出做了以下两件重要的操作:
(1)额外增加训练数据
使用AlphaFold2预测12M蛋白质序列的结构,将训练数据增加了近3个数量级。
(2)使用了GVP(Geometric vector perceptrons,几何向量感知机)
使用GVP-GNN(几何向量感知机-图神经网络)与GVP-Transformer(几何向量感知机),GCP是主要思想是用几何输入处理不变layer替代dense layer。
AF2模型在结构预测的准确率也是众所周知,利用AF2产生的数据,增加训练集数据提高模型准确率应该挺正常的。
另外该团队使用的GVP(几何向量感知机)序列到序列的架构,在下面两篇文章中做了更多的细节描述,并在pytorch-geometric工具中已开源使用。
下面的文章将GVP在第一篇的基础上,用矢量门控vector gating把原始的矢量非线性部分做了替换,允许信息从标量通道传播到矢量通道。
几何向量感知机(Geometric Vector Perceceptrons,GVP)具体架构如下
几何向量感知机架构旨在通过提高生物分子结构的几何推理能力,结合CNN和GNN方法的优势,从生物分子结构中学习。
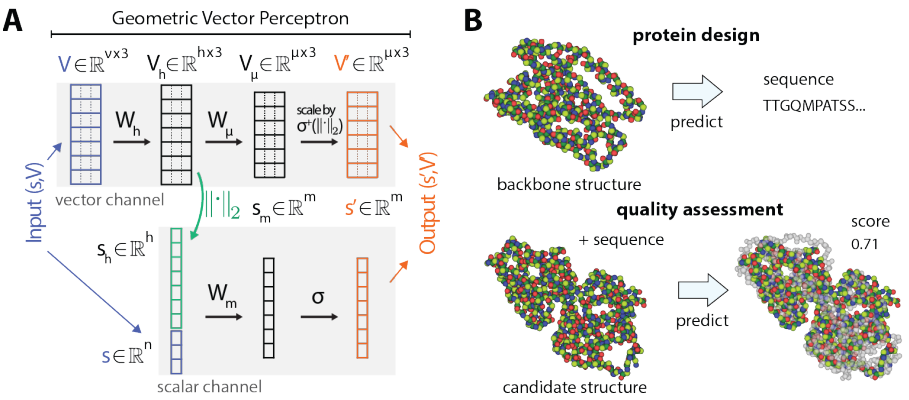
几何向量感知机是一个比较简单的模块,用于学习几何向量和标量上的向量值和标量值函数。也就是说,给定标量特征s和向量特征V,计算新特征(s', V'),该计算过程在图A,算法描述在Algorithm 1。
A. 几何矢量感知机示意图,Algorithm 1,给定标量和矢量输入特征的元组(s, V),感知机计算更新的元组(s', V'),s'是s和V的函数。B. 基于结构预测任务的说明。在计算蛋白设计(top)中,目标是预测一个氨基酸序列,该序列将折叠成给定的蛋白质骨架。单个原子被表示为彩色球体。在模型质量评估(bottom)中,目标是预测候选结构的质量分数,该分数衡量了候选结构与实验确定的结构(灰色)的相似性。
GVP的核心是由两个单独的线性变换Wm(用于标量特征),Wh(用于向量特征)组成,然后是非线性σ, σ+。然而,在转换标量特征之前,将转换向量特征Vh的L2范数连接起来;这允许从输入向量V中提取旋转不变信息。在向量非线性之前插入额外的线性变换Wu,用于控制输出维度,以独立于范数操作。
GVP虽然在概念上很简单,但可证明具有不变性/平等不变性(invariance/equivariance)和强的表征能力(expressiveness)的所需属性。首先,相对于三维欧式空间中旋转和映射的任意组合R,GVP的矢量和标量输出分别是等变和不变的-----即,如果GVP(s,V)=(s', V'),则
GNN通过根据旋转不变标量编码矢量特征(例如节点方向和边缘方向)来编码蛋白质的3D几何,通常通过在每个节点上定义局部坐标系来编码蛋白质的3D几何,作者建议将这些特征直接表示为几何向量 - 特征在空间坐标的变化下适当转换 - 在图传播的所有步骤中。
这种操作有两个好处。首先,不需要通过节点与其所有邻居的相对方向来编码节点的方向,而只需要为每个节点表示一个绝对方向。其次,它标准化了整个结构的全局坐标系,允许几何特征直接传播,而无需在局部坐标之间转换。
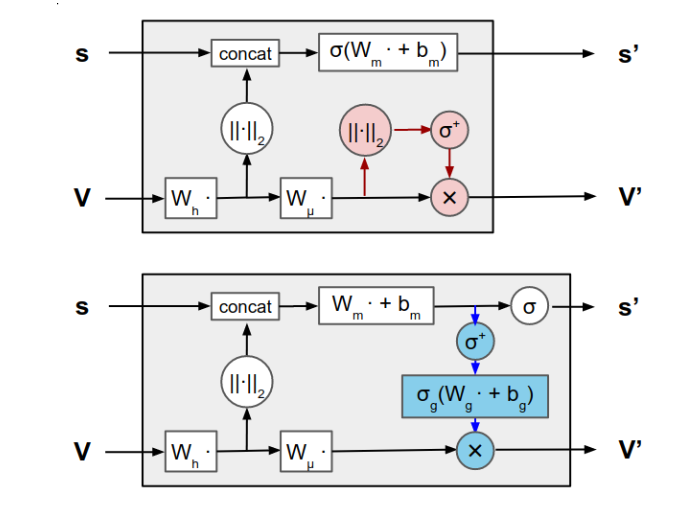
作者在下面的文章中将GVP结构做了轻微的改动。
原始的矢量非线性(红色)已被矢量门控vector gating(蓝色)所取代,允许信息从标量通道传播到矢量通道。圆圈表示行或元素操作。修改后的 GVP 是等变 GNN 中的核心模块。
参考文献
[1] Learning inverse folding from millions of predicted structures
[2] Learing from protein structure with geometric vector preceptrons
[3] Equivaiant Graph Neural Networks for 3D Macromelecular Strucuture
代码地址
https://github.com/drorlab/gvp-pytorch/blob/main/gvp/
https://github.com/facebookresearch/esm
细心的读者应该看到过Facebook团队在蛋白质表征上曾做过大量的工作,见本公众号其他文章。
Facebook 2.5亿个蛋白质序列的预训练模型:自监督语言模型学习生物学特性