抗原是侵入机体的外来物质,包括细菌、病毒等。抗体是机体在抗原刺激下,由B细胞分化成的浆细胞所产生的、可与抗原发生特异性结合反应的免疫球蛋白。抗体对抗原的高特异性和高亲和力使得抗体药具有靶向性强、副作用小和疗效显著等优点,因而在疾病的诊断和治疗中显示出其他类型药物无可比拟的优势,是治疗包括癌症及自身免疫性疾病在内的重要药物。人体在病毒刺激下可产生约10^20种抗体,加上人工合成的抗体,未知抗体空间巨大。然而,其中仅仅很小一部分抗体对特定抗原起作用。用湿实验来筛选能中和抗原的抗体耗时耗力,开发快速筛查中和抗体的计算方法十分必要。近日,以“Predicting unseen antibodies’ neutralizability via adaptive graph neural networks”为题的快速筛查抗体中和性的论文在Nature Machine Intelligence (IF:25.912)上发表。
研究人员提出了一种自适应图学习网络,该网络不需要抗原抗体的3D结构及领域知识,自动进化学习到影响抗原、抗体结合以及亲和力的结构特征;自适应学习得到的图网络具有丰富的生物学意义。以新冠为例,在抗原方面,算法量化了病毒变异的程度,加强了对病毒的生物学认识,有望用于指导对抗病毒的策略;在抗体方面,识别了新冠抗体的重要氨基酸序列及位置,对抗体人工合成有重要参考意义;算法推荐了广谱抗体,可以加强对抗病毒的变异。算法在HIV、新冠、流感、登革热四种病毒上得到验证,具有良好的泛化性能。
计算抗体对抗原的中和能力面临着多方面的挑战:(1)抗原抗体的结构未知,结合位点未知。抗体与抗原结合时,它仅与被称为抗原表位的蛋白区域结合,而不是与整个蛋白区域结合,例如新冠有超过1200氨基酸。通常,表位长度约为5或6个氨基酸。抗原又可以包含多个不同的表位。另外,病毒会不断地变异,人们对病毒的表位的特性认识要经历漫长的不断深入的过程。种种因素导致人们对抗原抗体的认知仍然十分有限,提取抗原抗体的有效特征十分困难;(2)样本量少是本研究遇到的另外一个挑战,例如流感病毒和登革热病毒都只有几百条中和反应数据,远远不够训练深度学习模型;(3)没有生物学意义的模型对人们改善对抗原抗体的理解是什么有限的,如何增加模型的可解释性以加强模型的生物学意义也是本课题要解决的难点。
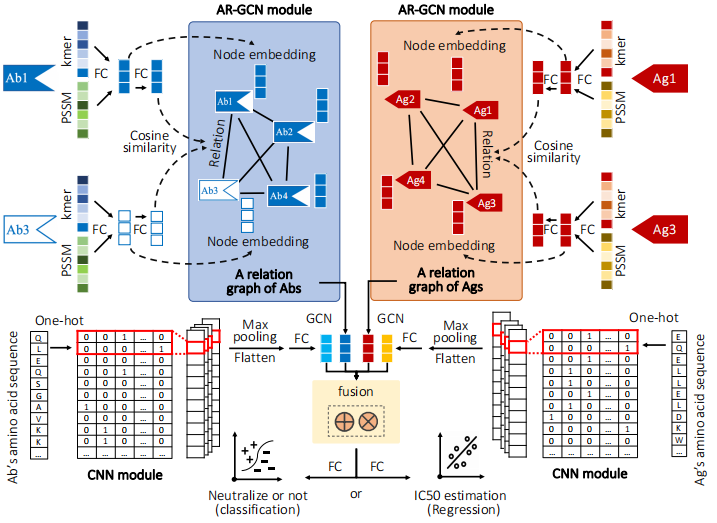
针对以上挑战,课题设计了动态自适应图神经网络,网络具有以下优点:(1)不同于现有的Graph Neural Networks(GCN),网络的边权重和节点在训练的过程中自动调整抗原抗体表征及其关系,最终形成抗原/抗体间的最有效关联。自适应图为模型提供了丰富的生物学解释;(2)算法通过一个神经网络经反向传播不断学习抗原/抗体的特征表达,在领域知识匮乏的前提下有效提取抗原抗体特征,学习到的特征为自适应网络的节点;(3)课题分别设计了局部和全局学习模块,学习抗原/抗体之间的反应和亲和性;(4)算法采用迁移学习克服数据量小的难题。模型性能显著超越其他基线方法。模型框架如图1所示:
图1:模型的整体框架
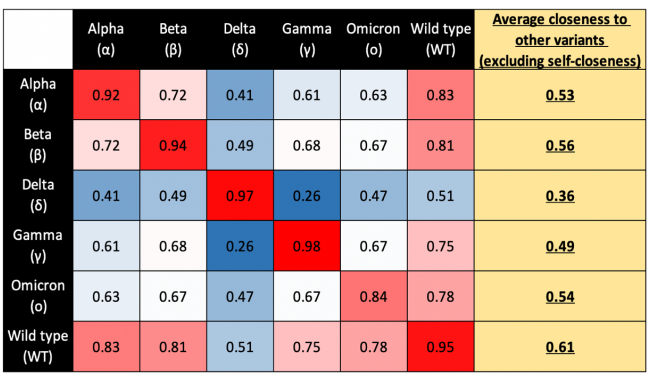
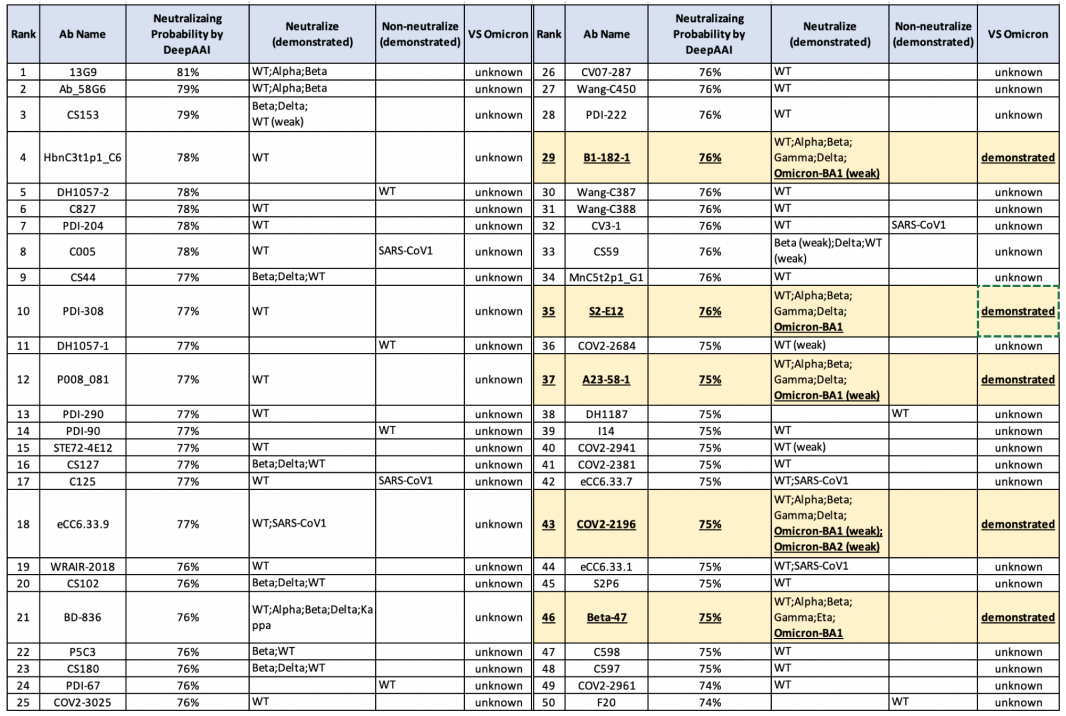
算法从以下几个层面加强对抗原和抗体的生物学理解:(1)自适应图量化抗原/抗体间的相似性,该结果可以用于指导对病毒变异的理解,见下表1;(2)识别了抗体的重要的氨基酸序列,该发现有望指导抗体人工设计,见图2;(3)新冠广谱抗体推荐,推荐的广谱抗体有望用于对抗病毒的变异,见表2。
表1:自适应关系图有效地量化了抗原间的不同:相比于其他病毒,Delta 显著不同于Alpha和Beta,这与Delta症状与野生毒株的症状表现不同相一致;Omicron与自身的相似性只有0.84,该值反映出Omicron亚型间的差异较大。
图(2):上面4行和下面3行分别是重链和轻链的重要3mer位置图
**图2:根据特征学习神经网络的权重,我们识别了抗体的具有重要性的k-mers。对于重链(上面4行),最重要的3-mer序列位于重链尾部,第2、3重要的3-mer位于连续的第44到 47个氨基酸;对于轻链(下面3行),最重要的3-mer位于轻链中间,而2、3重要的3-mer也比较靠近。 **
表2:推荐了50个潜在的中和新冠的抗体。其中的5个已经被验证,除去9个已有不同的结论之外,另外的36个抗体有待验证,详请参见原文。
另外,课题利用HIV的数据训练的模型对流感和登革热进行了迁移学习,模型性能表现良好。 参考资料 Zhang, J., Du, Y., Zhou, P. et al. Predicting unseen antibodies’ neutralizability via adaptive graph neural networks. Nat Mach Intell 4, 964–976 (2022). https://doi.org/10.1038/s42256-022-00553-w