今天给大家介绍一篇由厦门大学研究团队发表在Briefings in Bioinformatics的文章。作者在该论文中将DTI预测视为一个链路预测问题,并提出了一种基于具有注意机制的异构图的端到端模型(DTI-HETA)。
摘要药物-靶标相互作用(DTI)预测在药物重新定位、药物发现和药物设计中起着重要作用。然而,由于化学和基因组空间的庞大以及药物和靶标之间相互作用的复杂性,DTI的实验鉴定具有成本高且耗时长的问题。近年来,新兴的图神经网络(GNN)被应用于DTI预测,因为DTI可以有效地用图表示。然而,其中的部分方法仅基于齐次图,另外部分方法由两个不可联合训练的解耦步骤组成。为了通过整合异构图信息进一步探索基于GNN的DTI预测,本研究将DTI预测视为一个链路预测问题,并提出了一种基于具有注意力机制的异构图的端到端模型(DTI-HETA)。该模型在药物靶标信息构造的异构图上利用图卷积神经网络获得药物和靶标的嵌入表示,并在节点的信息聚合过程中引入了图注意机制。在得到药物和靶标的嵌入表示后,模型应用内积解码器来进行DTI预测。为了评估DTIHETA的性能,本研究在两个数据集上进行了实验。实验结果表明,本文的模型优于现有的方法。 1.****研究背景尽管在药物研究和开发方面取得了许多进展,但传统的药物发现过程仍然具有风险。风险主要体现在耗时和成本上,统计表明药物新分子实体研发的时间周期估计为12年,成本估计为18亿美元。目前,加速药物发现过程的关键是确定药物是否能与靶相互作用,因为识别药物和靶点之间的相互作用有助于候选新药的有效筛选工作。尽管有成千上万的化合物存储在各种数据库中,但大多数化合物没有相应的目标信息。随着药物和靶点可用数据的增长,越来越多的学者试图研究有效的计算方法来识别新的药物-靶点相互作用(DTI)。目前,DTI预测的计算方法主要有三类:基于配体的方法、对接模拟和化学基因组学方法。虽然在生物学上被广泛接受,但基于配体的方法和对接模拟面临许多限制,例如已知配体的数量不足、蛋白质的3D结构未知等等。化学基因组学方法可进一步分为几类,如基于机器学习的方法和基于相似性的方法。在这些化学基因组学方法中,基于机器学习和深度学习的模型因其可靠的预测结果而备受关注。在这些方法中,关于药物和靶点的知识被编码为特征以训练模型。然后将训练后的模型用于预测新的DTI。这些方法通常涉及特征提取和DTI预测两方面,但在构建模型时很少考虑药物-靶点对的潜在有效相互作用,并且不能利用药物-药物和靶点-靶点相似关系。此外,这种方法仅使用DTI矩阵作为用于训练的标记矩阵,忽略了异构生物数据中包含的信息。最近,图形神经网络(GNN)的快速发展将深度学习的应用扩展到了图结构领域,相关方法也应用于药物发现。然而,现有的一些基于图的方法一方面是为齐次图设计的,缺少异构数据源的各种信息。另外,这些方法在聚合中心节点信息时没有考虑不同相邻节点的贡献,导致预测性能较差。本文基于对相关工作中不足的分析,提出了一种应用注意力机制和图卷积方法基于异构图的端到端DTI预测模型。 2.模型方法
图1.DTI-HETA模型架构图在本模型中,首先基于药物-药物和靶标-靶标相似度矩阵和DTI矩阵构建异构图。然后,利用图卷积神经网络获得药物和目标的嵌入表示。为了突出不同邻域节点在聚集图卷积信息中对中心节点的贡献,在节点嵌入过程中引入了图注意机制(GAT)。最后,应用内积解码器来预测DTI。模型的整体架构如图1所示。模型的图卷积模块使用图G中中心节点的相邻节点来定义信息传播框架,在这里称为节点的局部计算图。参数和权重在所有局部计算图之间共享,在同一局部计算图中应使用相同的信息传播方法。如图1的图嵌入部分所示,有四种不同的局部计算图:(a)、(b)、(c)和(d)。在(a)中,中心节点是药物d1,并且其所有相邻节点是药物;在(b)中,中心节点d3是药物,并且存在两种类型的相邻节点:药物d1和d5以及靶标t4。(c)和(d)是另两种情况,其中目标节点位于中心。在GCN的每一层中,根据原始图中的边类型计算四个局部计算图。局部计算图隐藏状态的计算先对中心节点的相邻节点进行线性变换,相同边类型共享权重系数,然后求和并使用ReLu函数进行激活,从而得到该局部计算图的隐藏状态。药物和靶标节点的隐藏状态由节点在各自局部计算图上的隐藏状态相加得到。另外,模型考虑到不同相邻节点的贡献不同,引入了图注意机制(GAT)。GAT在中心节点信息聚合过程中学习得到一个注意力系数e,为相邻节点分配不同的权重。以节点i和j为例,GAT分别对两个节点执行线性变换,然后使用一个映射函数f将注意力系数eij分配给图中的节点。映射函数f通过一个单层前向传播神经网络学习得到。为了比较不同节点之间的注意力系数,再使用softmax函数进行归一化。在计算节点i和j之间的注意力系数之后,GAT可以将不同的权重分配给中心节点的相邻节点。 作者认为本模型的优点有: 1.基于异构图设计预测模型,可以充分利用数据所携带的信息。2.为异构图设计相应的图卷积策略,并引入GAT以突出相邻节点的不同贡献。3.以端到端的方式训练模型,模型参数可以更有效地更新。 **3.**验证实验
模型验证实验包括三个方面:1.作者基于自己构建的数据集,分析模型中涉及的参数,包括节点嵌入维数和卷积层数,获得一组最优模型参数。
图2.模型参数实验结果图
实验结果如图2所示。其中,随着节点嵌入维数的增大,模型性能先提高,然后逐渐降低。作者分析当d太小时,节点嵌入携带的信息过少,导致拟合不足并影响最终的预测性能。而当d太大时,提取的节点嵌入可能包含噪声,这也会影响预测性能。而卷积层数的增加则会一直导致模型性能的逐渐下降。这是因为图卷积会使相邻节点的表示更加接近,层数过度增加将导致节点表示收敛到一个点,从而导致过度平滑。同时,层数的增加也导致模型参数数量急剧增加,这可能导致过度拟合,影响预测性能。 2. 对比分析了不同解码器对预测性能的影响。
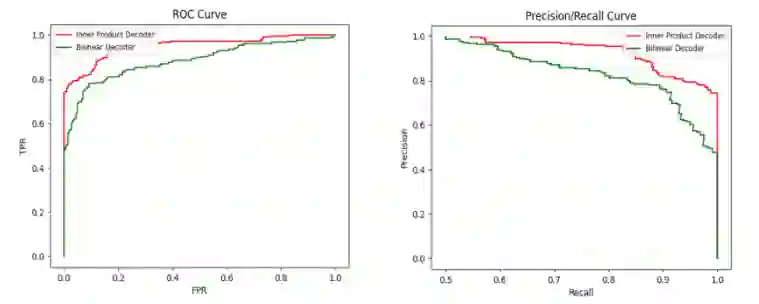
图3.解码器实验结果图
实验结果如图3所示,结果表明内积解码器比双线性解码器有更好的预测结果。这可能是因为双线性解码器引入了可训练权重矩阵,增加了模型中的参数并导致过拟合。此外,DTI-HETA模型使用简单解码器,可以获得更好的预测结果,表明该模型不依赖于特定解码器。 3. 将本文提出的模型在两个数据集上与其他方法进行比较,以验证其优越性
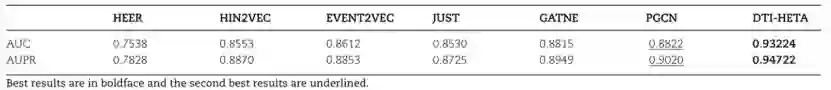
表1.作者数据集上不同模型的AUC和AUPR比较表1显示了DTI-HETA在作者自己构建数据集上与其他六个其他模型之间的比较结果。
表2 .Yamanishi-Enzyme数据集上不同模型的AUC和AUPR比较
表3 Yamanishi-Ion数据集上不同模型的AUC和AUPR比较
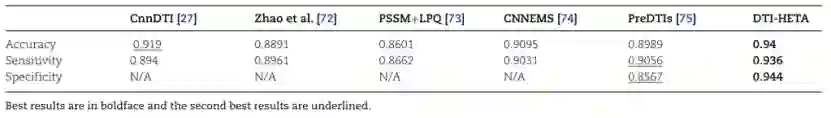
** **表4 Yamanishi-Enzyme数据集上不同模型的Accuracy , Sensitivity和Specificity比较
** **表5 Yamanishi-Ion数据集上不同模型的Accuracy , Sensitivity和Specificity比较
另外,由于Yamanishi数据集广泛用于其他研究,因此选择了一些最先进的模型与DTI-HETA进行比较,具体比较结果见表2-表5。实验结果表明,本文提出的模型在性能上比其他模型均具有一定的优势。 **4.**总结
目前,药物和靶蛋白的异构图已成为DTI预测的有力工具。但是基于药物-靶点异构图的GNN模型的巨大潜力尚未得到充分利用。面临的问题比如正负数据的类别不平衡可能会影响GNN方法的性能。此外,GNN可以容易地捕获数据集中可能存在的偏差模式。 在本研究中,本文提出了DTI-HETA,一种用于预测异构数据源DTI的端到端GCN模型。DTI-HETA通过定义图卷积获得药物和目标的节点嵌入表示,该图卷积将注意力机制引入异构图中,然后使用解码器预测潜在的DTI。实验结果表明,DTI-HETA优于最先进的端到端模型和非端到端模式。 此外,作者认为尽管DTI-HETA显示了良好的预测性能,但该研究仍面临一些挑战。 首先,数据处理时随机选择未知药物-靶点对作为阴性样本,这可能限制模型的预测精度,未来希望可以将实验测量的阴性样品作为阴性样本。另一个挑战是,下一步的工作可以考虑合并和探索更多与药物和靶点相关的异质网络,如代谢网络和药物-疾病网络。这些不同的异构网络将提供有助于DTI预测的丰富语义信息。 总的来说,DTI-HETA模型可以通过缩小DTI的搜索空间,推动新型DTI的发现工作,并且可能为理解药物作用的潜在机制提供重要提示。