我们很高兴地邀请到来自哈佛大学的Marinka Zitnik教授来给我们做一个关于计算生物学中的图神经网络的讲座。在本讲座中,Marinka教授概述了为什么图学习技术可以极大地帮助计算生物学研究。具体来说,本演讲涵盖了3个范例用例: (1) 通过异构知识图谱的多关系链接预测发现安全的药物-药物组合;(2)通过学习子图嵌入对患者结局和疾病进行分类;(3)通过图的少样本学习,学习有效的疾病治疗方法。
机器学习的成功很大程度上依赖于数据特征的选择,而这些数据特征是应用于这些方法的。因此,部署算法的大部分实际工作都是在设计支持有效机器学习的特征。我们已经在开发表示学习方法方面取得了重大进展,该方法扩大了机器学习在科学和医学中的范围,并简化了其适用性。
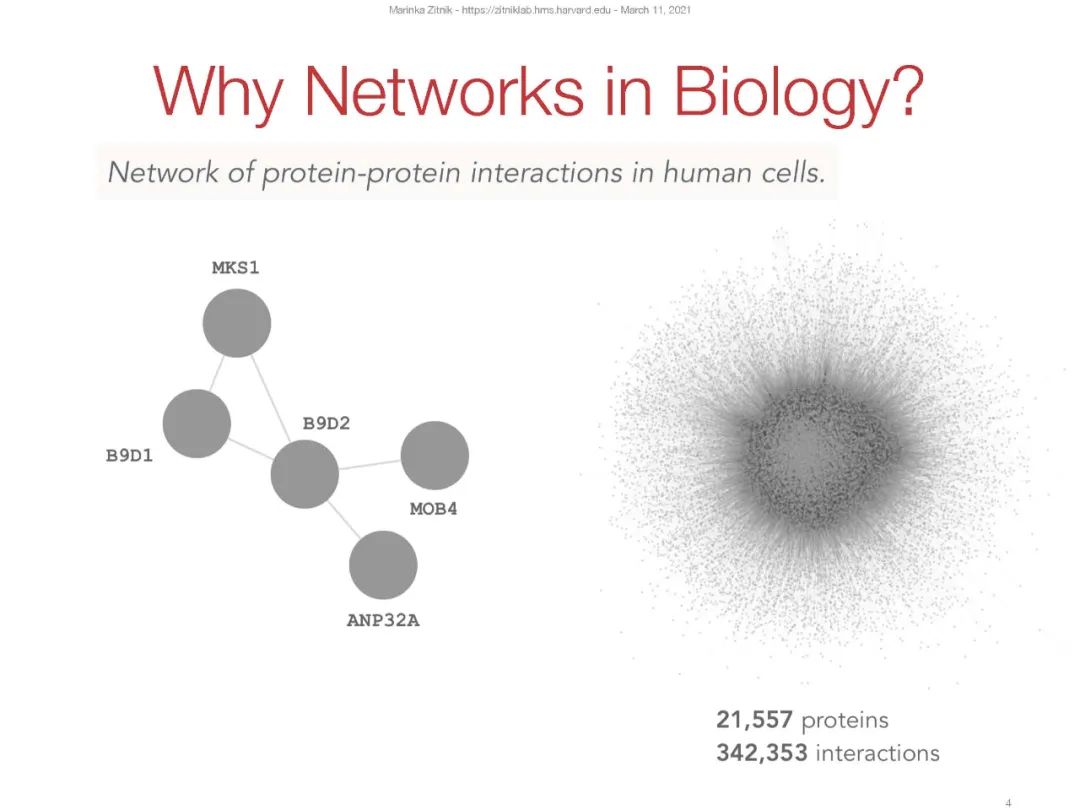
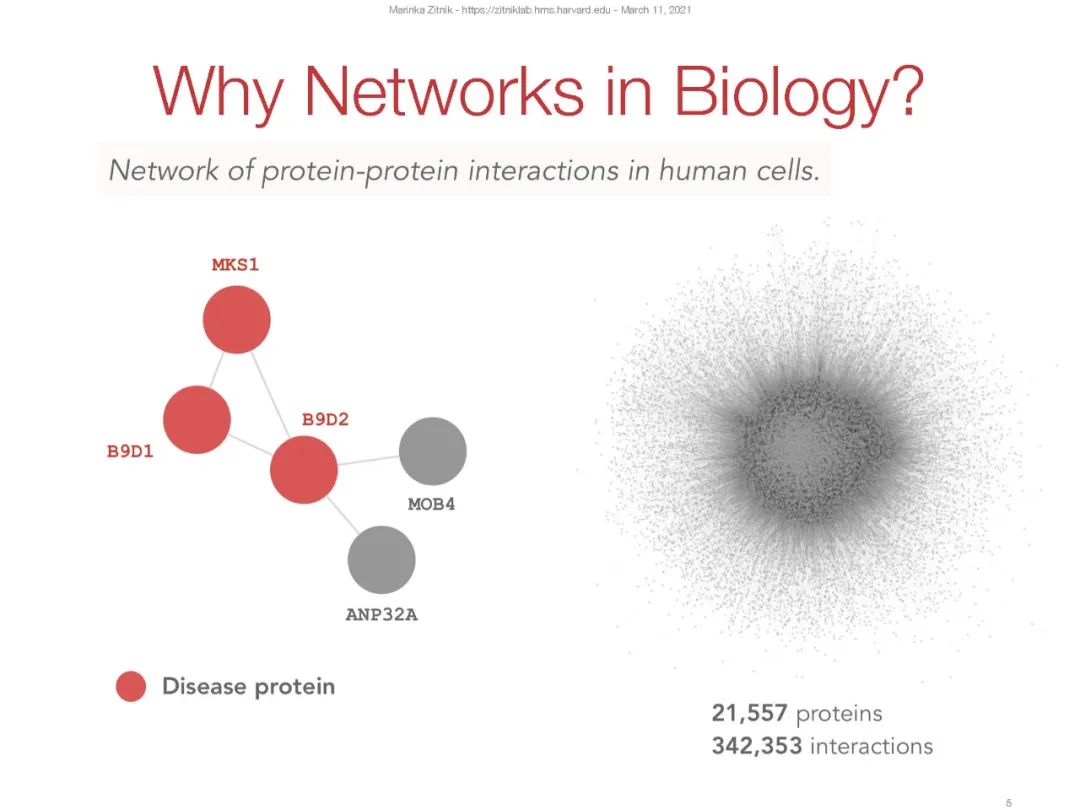
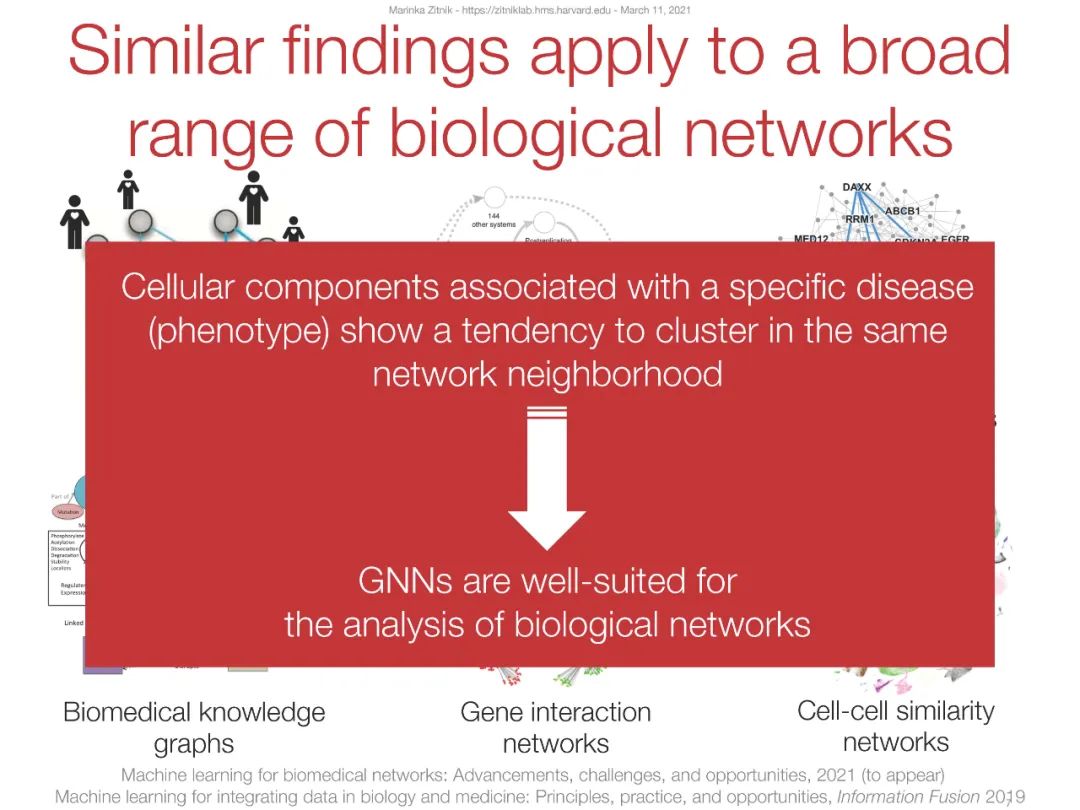
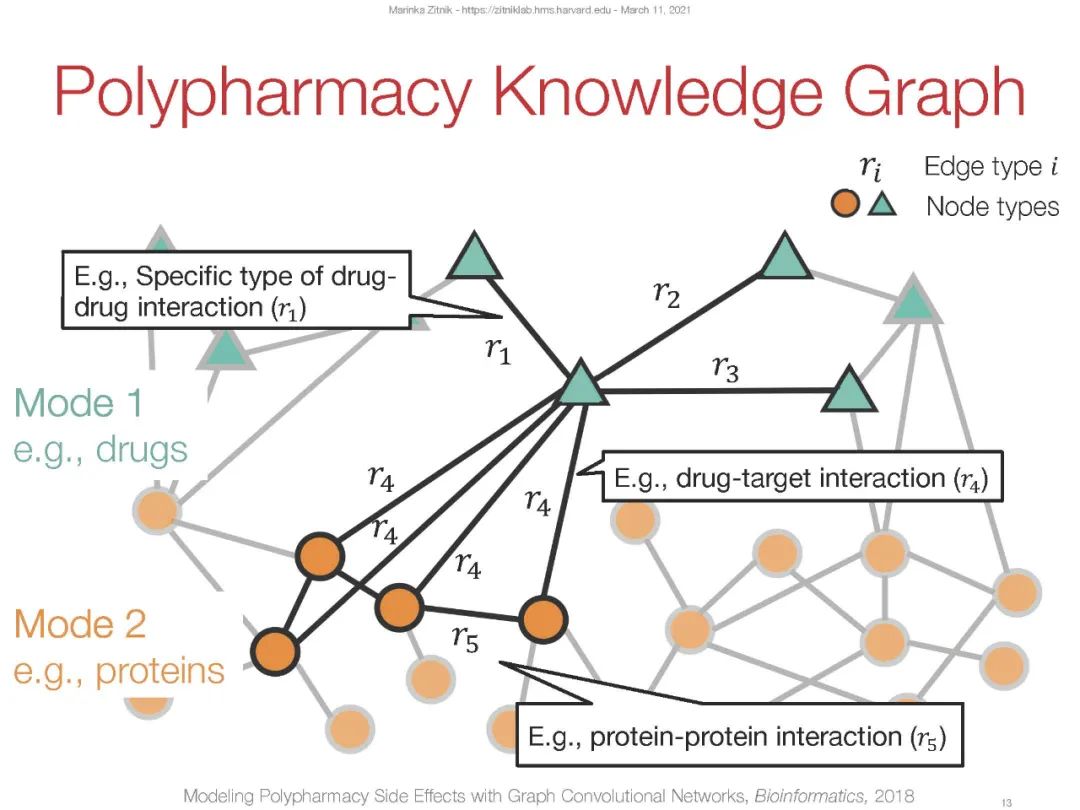
然而,目前流行的深度和表示学习算法是为具有规则的网格状结构的数据设计的(例如,图像具有二维网格结构,序列具有线性的一维结构)。这些算法无法真正利用实体之间不规则交互的复杂、互连的数据,即图的本质——边缘。我们正在研究解决这些挑战的方法。我们方法的技术核心是向量空间嵌入的概念。我们通过指定深度转换函数或图神经网络,将节点或更大的图结构映射到低维空间中的点(称为嵌入),来形式化这一思想。重要的是,这些函数被优化以嵌入输入网络,以便在这个学习的空间中执行代数运算,反映网络的拓扑结构。
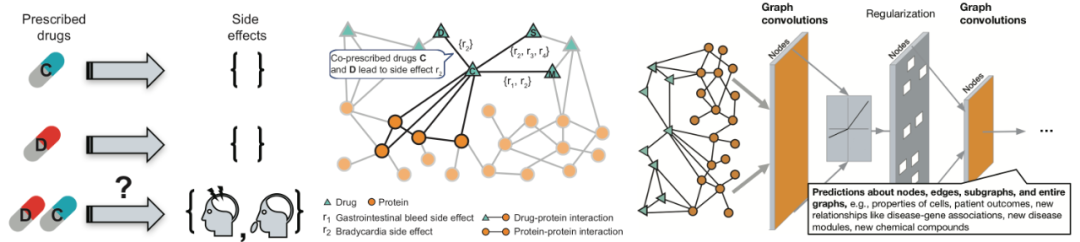
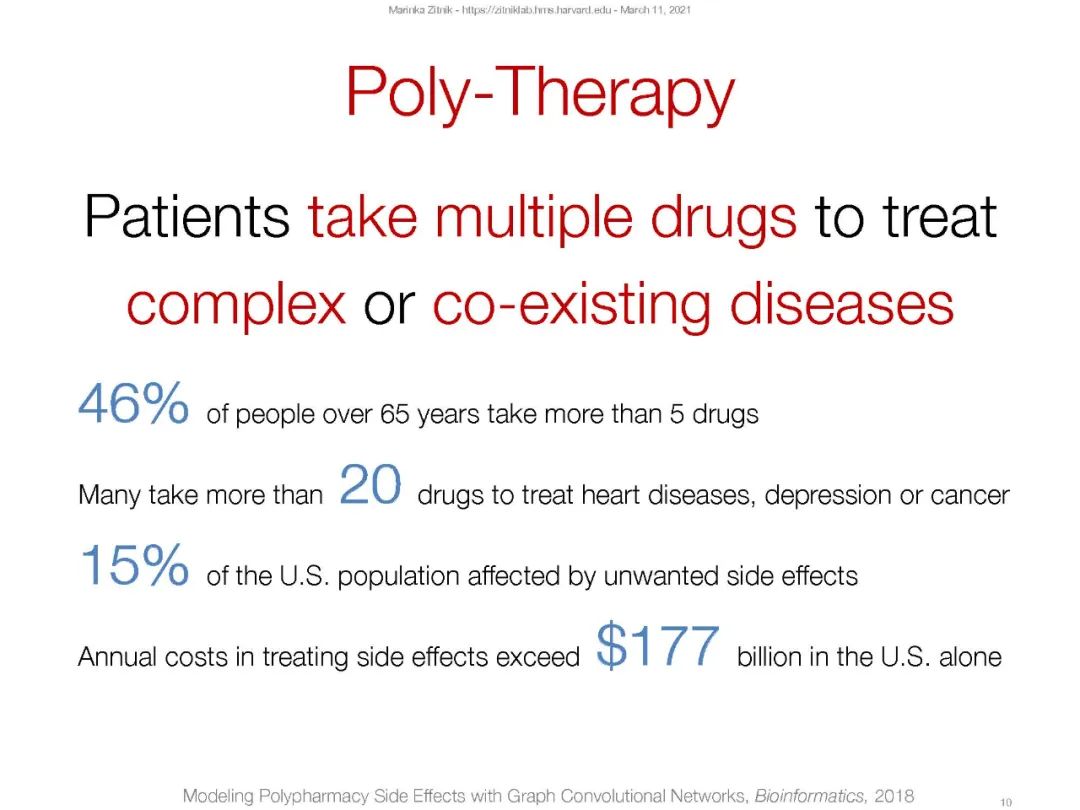
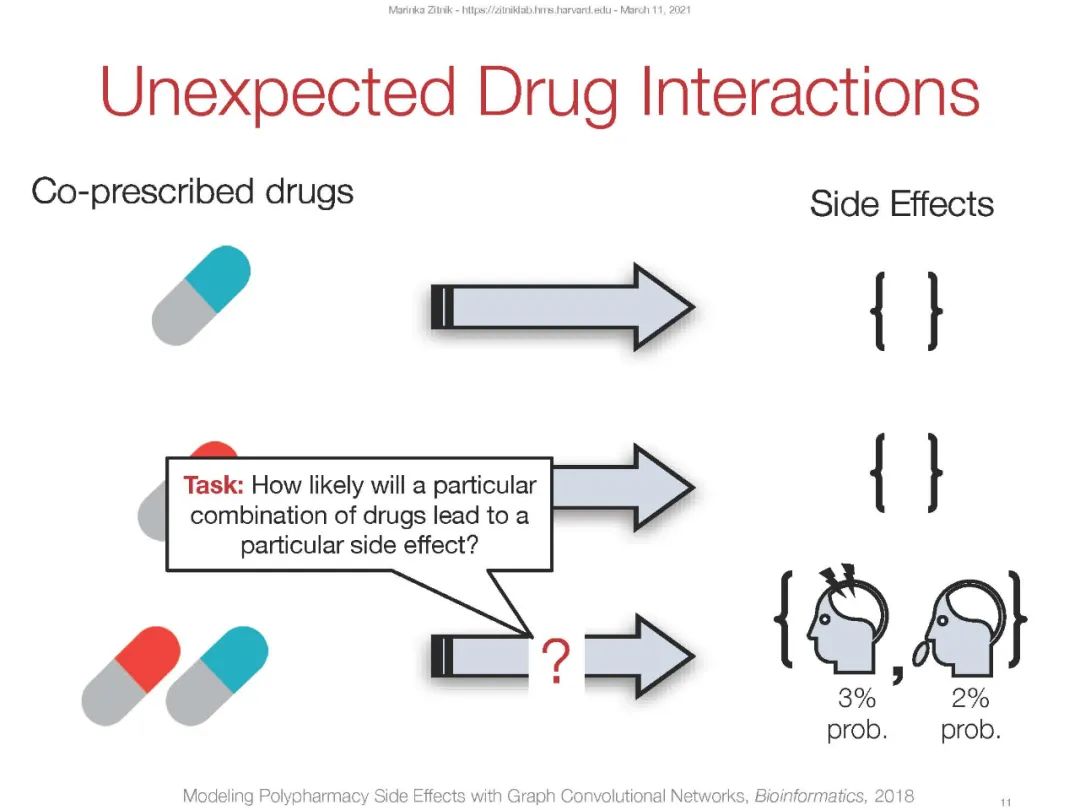
我们的研究在生物信息学和网络生物学和医学的深度学习领域开创了图神经网络的先河。这使我们能够更广泛地应用神经网络,并将目光投向神经网络学习图像和序列的经典应用之外的新领域。例如,我们展示了嵌入式技术如何使药物用于新的适应症,以及发现了数十种药物组合,这些组合对患者来说是安全的,而且比现在的治疗方法有更少的副作用。此外,我们还发现嵌入可以通过识别药物靶标、疾病蛋白、分子功能和其他表型来实现精确的分子表型,这比更复杂的算法要好得多。
我们也在积极开发机器学习的方法,以学习可操作的表示形式——使其能够支持可操作的假设——并允许我们模型的用户提出“如果……”的问题,并获得准确、精确、稳健和有意义的预测。
在生物医学数据的背景下,我们希望改进算法,用更少的数据训练出更多的人,利用模型的能力,将从一种数据类型获得的预测能力应用到另一种类型,设计情境自适应的人工智能,使其能够在遇到新任务和新情况(例如,新的病人、疾病或细胞类型)时,学习和推理从未见过的系统。
https://zitniklab.hms.harvard.edu/research/
相关参考文献:
Modeling Polypharmacy Side Effects with Graph Convolutional Networks To Embed or Not: Network Embedding as a Paradigm in Computational Biology Predicting Multicellular Function Through Multi-Layer Tissue Networks GNN Explainer: Generating Explanations for Graph Neural Networks Embedding Logical Queries on Knowledge Graphs Learning Structural Node Embeddings via Diffusion Wavelets