编译 | 程昭龙
审稿 | 王静
本文介绍由哈佛医学院的Martin Hemberg和韩国延世大学生命科学与生物技术学院生物技术系的Insuk Lee共同通讯发表在Nucleic Acids Research的研究成果:单细胞生物学面临的一个主要挑战是识别细胞类型特异性基因功能,这可能会大大提高精准医学的水平。基因的差异表达分析是一种流行但不充分的研究方法,需要补充与细胞类型相关的功能。因此,作者提出了单细胞网络分析平台scHumanNet,用于解决人类不同基因功能的细胞异质性。scHumanNet是基于HumanNet参考相互作用组构建细胞类型特异性基因网络(CGN), 它在单细胞转录组数据上构建的CGN比其他方法显示出更高的细胞环境功能相关性。此外,基于跨细胞类型网络紧致性的基因信号的细胞反褶积揭示了与T细胞相关的乳腺癌预后标志物。scHumanNet还可以利用CGN的中心性对与特定细胞类型相关的基因进行优先排序,并确定CGN在疾病和健康状况之间的差异中心。作者通过揭示乳腺癌预后基因GITR的T细胞特异性功能效应,以及抑制神经元特异性自闭症谱系障碍基因的功能缺陷,证明了scHumanNet的有效性。
简介
基因不是孤立地起作用的,因为它们编码的蛋白质彼此相互作用,并与其他分子相互作用。从网络生物学的角度来看,分子间的相互作用决定了每种细胞类型的功能。然而,由于环境依赖性,细胞类型特异性的分子相互作用难以识别和解释。单细胞RNA测序(scRNA-seq)的出现使得能够表征复杂组织中的不同细胞类型,以及确定它们在混合细胞群中的相互作用。
从单细胞转录组数据推断细胞类型特异性基因网络(CGN)的主要困难是缺乏细胞类型特异性基因相互作用的金标准。因此,研究人员经常使用模拟的合成网络。使用参考蛋白质之间的相互作用进行的评估表明,大多数网络推断方法,包括为bulk RNA-seq数据和scRNA-seq开发的方法,都无法从scRNA-seq数据重建准确的基因相互作用网络,这可能是因为scRNA-seq数据的稀疏性和虚假的技术差异。为了克服这一问题,需要开发一种使用scRNA-seq数据来研究细胞类型特异性基因功能的精确网络建模方法。
目前使用单细胞转录组数据构建网络的方法有两种:无参考推断和参考引导推断。前者更受欢迎,可以直接从单细胞转录组数据中发现基因相互作用,但其假阳率普遍较高。相比之下,参考引导的方法通过过滤背景相关单细胞的给定转录组的参考相互作用组来构建网络,因为过滤的相互作用很可能存在于给定的细胞类型中。
在本文中,作者提出了使用单细胞转录组数据构建CGN的计算平台scHumanNet。作者使用HumanNet作为参考相互作用组,因为它是在疾病基因预测方面表现最好的人类基因网络之一。同时,作者使用了SCINET算法的修改版本。除了构建CGN外,scHumanNet还提供了几种分析工具来帮助研究疾病基因的细胞类型特异性效应。通过网络中心性分析表明, scHumanNet在检索细胞类型特异性基因方面优于其他单细胞网络推断方法,适用于基因细胞类型特异性的研究。作者还证明了与相同细胞类型相关的基因在网络中表现出更高的组内连接度(即紧凑性)。利用CGN之间的网络紧凑性,作者将乳腺癌预后特征去卷积为细胞类型,并识别出与免疫细胞(而非癌细胞)相关的细胞类型。作者还发现,已知标记基因GITR的预后价值由于其T细胞特异性中心性而与T细胞相关。此外,作者开发了一种用于差异中心性分析的统计框架,该框架揭示了疾病基因中的细胞类型特异性功能缺陷。将该分析框架应用于自闭症研究中的大脑scRNA-seq数据,实验结果发现,在疾病状态下的抑制性和兴奋性神经元中,相互作用网络的失调升高。
结果
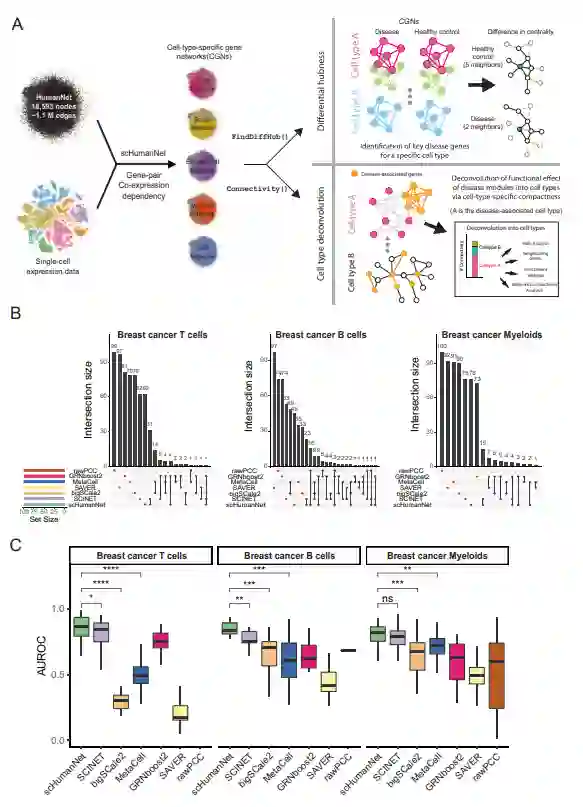
scHumanNet可有效地检索每种肿瘤细胞类型的特异性基因 为了评估通过scHumanNet获得的CGN (图1A)是否比其他推断方法生成的CGN更适合研究细胞类型特异性基因功能,作者比较了各种无参考和有参考指导的方法。在已发表的乳腺癌scRNA-seq数据上,作者使用rawPCC、MetaCell、SAVER、GRNboost2和bigSCale2五种无参考方法以及基于PCNet的SCINET参考指导方法构建了T细胞、B细胞、骨髓细胞、ECs、CAFs和癌细胞网络。虽然GRNBoost2是一种仅限于TF-靶标调控相互作用的网络推断方法,但作者之所以将其纳入比较中,是因为它是SCENIC的基础算法,而SCENIC又是在单细胞生物学中广泛应用的网络建模方法。不同的网络构建方法所考虑的相互作用类型不同,而对于scHumanNet而言,广泛的相互作用定义可以获得最佳结果。
网络节点的功能重要性通过其中心性来衡量,而细胞类型特异性基因可能在相应的细胞类型中发挥重要作用。因此,作者期望每个CGN中具有高中心性的基因被富集为细胞类型特异性基因。使用基于每个网络边得分的加权度中心性,作者比较了每个网络中的前100个基因,从而忽略网络大小的差异。实验结果表明,在评估每种细胞类型的前100个hub基因时,六种网络构建方法都没有观察到太多重叠(图1B), 并且这种模式与前50和200个hub基因相同。为了确定hub基因是否优先于细胞类型特异性功能,作者评估了每个细胞类型特异性输入的AUROC。使用Azimuth celltype数据库,其中包含从大型scRNA-seq数据集中提取的特征标记基因,可知在参考引导的CGN中,通过中心性对细胞类型标记基因的检索率高于无参考的CGN (图1C)。在参考引导的CGN中,scHumanNet比SCINET更优先考虑细胞类型特异性基因,特别是在B细胞和T细胞中。这些结果表明,scHumanNet在检索人类基因细胞类型特异性功能方面优于其他CGN构建方法。
图1 scHumanNet和其他方法生成的细胞类型特异性网络比较
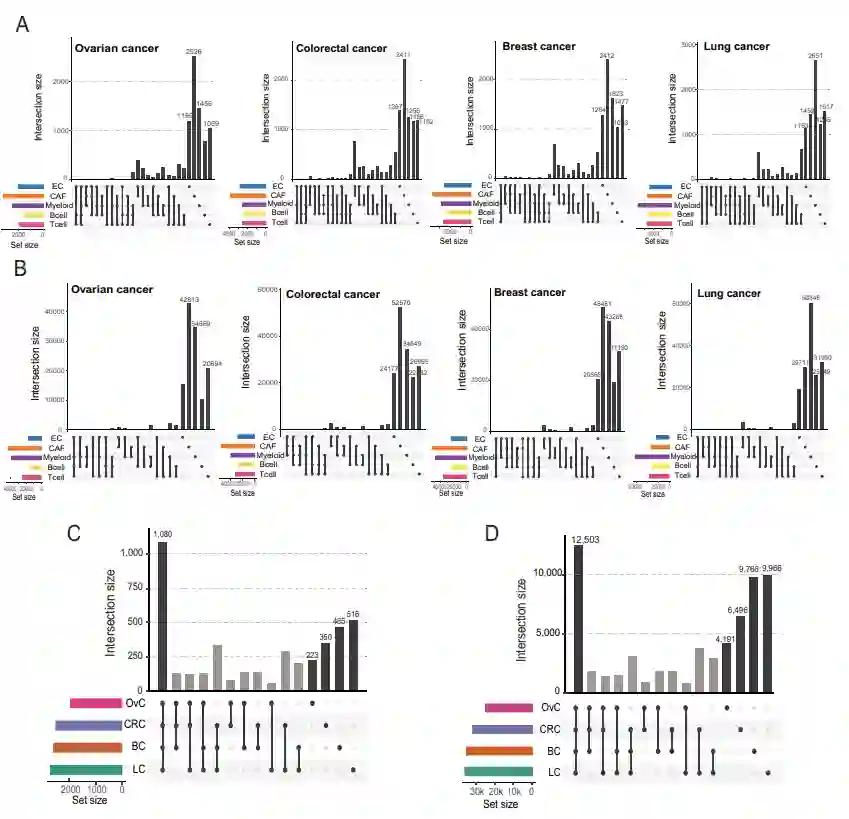
scHumanNet揭示了CGN在不同癌症类型间的共性和差异性 肿瘤浸润细胞在癌症中的功能通常使用细胞类型特异性基因表达进行研究。本文表明,网络生物学可以补充基于表达的功能研究。为此,作者使用scHumanNet构建了T细胞、B细胞、骨髓细胞、ECs、CAFs以及来自乳腺癌、结直肠癌、肺癌和卵巢癌的癌细胞的CGN。接下来,作者研究了这些CGN是否可以提供与细胞类型或疾病状态相关的功能见解。不同类型非癌细胞之间的网络比较显示,在所有癌症中,只有一小部分节点和边在细胞类型之间共享(图2A, B),而很大一部分是在癌症类型中共享(图2C, D)。这些结果表明,scHumanNet生成的CGN主要是由细胞环境而不是疾病或组织环境影响的。
图2 scHumanNet生成的CGN在不同癌症类型中的共性和差异性
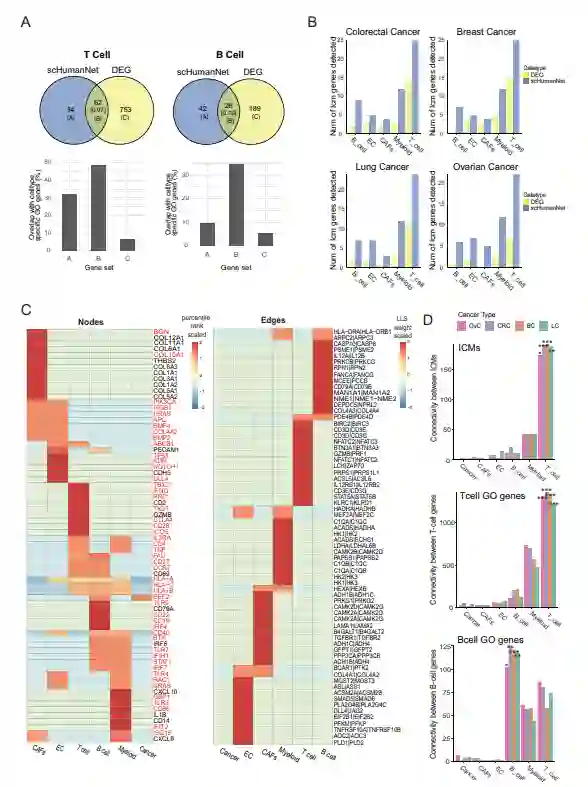
scHumanNet的中心性和紧凑性预测基因功能的细胞类型特异性 在不同细胞类型中重新连接基因相互作用可能会改变不同细胞环境中具有不同功能重要性的基因的网络中心性。因为具有高度中心性的hub基因在给定的细胞环境中与许多其他基因相互作用,所以它们更有可能在维持特定细胞类型的功能方面发挥重要作用。因此,作者研究了每种肿瘤浸润细胞的scHumanNet hub基因是否可以反映不同癌症类型的细胞环境依赖性的功能重要性。为了评估细胞类型的特异性,作者利用GO数据库来整理与B细胞或T细胞相关的可靠基因。接下来,作者评估了scHumanNet的能力,根据与B细胞或T细胞中已知功能基因的重叠来预测每种细胞类型的特异性基因。值得注意的是,针对每种细胞类型的基于网络和基于表达的候选基因表现出较低的一致性,这表明两种预测具有互补性(图3A)。此外,两种预测之间的交集显示出与已知细胞类型特异性基因的强烈重叠。在大多数情况下,基于网络的预测与已知的细胞类型特异性基因更相似,这进一步证实了scHumanNet hub基因可以有效地识别细胞类型特异性基因。
作者预计ICM将在肿瘤浸润细胞的特异性基因中富集。因此,作者汇编了43个先前确定的ICM,并比较了它们与不同细胞类型的scHumanNet hub和DEG的重叠。对于所有细胞类型,实验观察到通过scHumanNet hub基因对ICM的检索率高于DEG (图3B)。此外,在所有的癌症类型和细胞类型中,通过DEG检索到的ICM是scHumanNet检索到的ICM的子集。
作者使用scHumanNet构建的CGN中的加权中心性对基因进行优先级排序,发现它对细胞类型特异性标志基因具有高度的预测能力。基于这一观察结果,作者选择更密切地研究TF,因为TF是特定细胞分化和维持特性的关键决定因素。细胞类型特异性差异表达分析通常不足以检测特定细胞类型的TF,因为TF的基础表达水平普遍较低。相反,基于网络的方法已被广泛用于推断TF -靶标的相互作用。假设CGN中的网络中心性可以有效地对特定细胞类型的TF进行优先排序。为了评估DEG和scHumanNet中心性对细胞类型特异性TF的预测,作者从TF- Marker(细胞类型特异性TF数据库)中检索了细胞类型特异性TF。由于输入数量有限,作者只分析了B细胞和T细胞的特异性TF。实验结果表明,在不同癌症类型的B细胞和T细胞中的TF优先排序方面,基于网络的方法表现较好。
作者还发现scHumanNet中心性可以预测细胞类型特异性疾病相关基因。例如,在所有类型的癌症T细胞中,前15个hub基因包括参与细胞介导免疫和免疫检查点信号通路的基因(图3C)。经实验证明,使用scHumanNet的网络中心性可以比差异表达分析更有效地识别在给定细胞环境中发挥重要作用的基因。此外,scHumanNet中的FindAllHubs()可以在健康和疾病环境中识别具有细胞类型特异性功能的hub基因。
不同细胞类型之间的分子网络重组可能导致组内连接(或紧凑性)的差异,这也可以用于估计功能相关性。作为概念验证,作者使用了ICM基因以及B细胞和T细胞特异性基因。scHumanNet中的Connectivity()函数使用不需要识别最优参数的限制性随机抽样,针对非参数零模型检验组内连接的重要性。正如预期所示, ICM基因以及B细胞和T细胞特异性基因分别与所有癌症类型的T细胞、B细胞和T细胞类型相关(图3D)。这表明基于网络的方法提供了一种互补且直观的方法,可以根据紧凑性将基因集分配给功能相关的细胞类型。
图3 通过scHumanNet的差异表达分析和网络中心性预测细胞类型相关基因
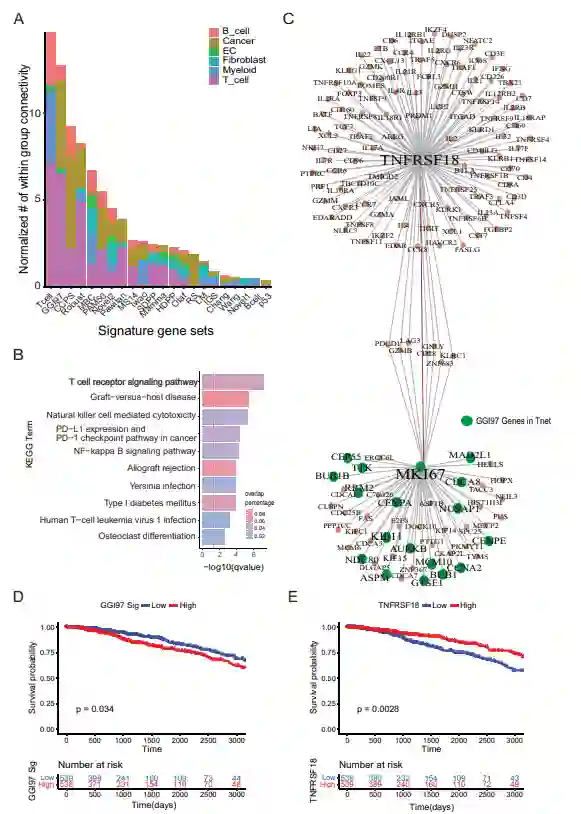
使用scHumanNet对癌症预后特征进行细胞类型反卷积 ICM在T细胞网络中表现出最高的致密性,这与它们的细胞作用一致。作者假设可以利用scHumanNet在CGN上的网络紧凑性,将从整体组织中获得的疾病相关基因特征反卷积到单个细胞类型。例如,癌症预后特征可能与癌细胞相关,因为它们通常在肿瘤组织中被识别。然而,肿瘤组织中也常含有非癌细胞,如基质细胞和免疫细胞,并且一些预后相关基因可能在肿瘤微环境的非癌细胞中发挥作用。为了验证这一假设,作者研究了在乳腺癌中报告的33个预后特征,并使用scHumanNet测量了CGN中每个预后特征的标准化组内连接(图4A)。实验结果表明,来自非癌细胞的许多预后特征具有很强的网络紧凑性,特别是来自T细胞的Tcell、GGI97、Robust、Pawitan。这也表明,T细胞功能可能在一定程度上解释了乳腺癌的临床结果(图4B)。
T细胞增殖在免疫治疗反应中很重要。在T细胞网络中的24个GGI97特征基因中,有18个是Ki67的直接邻居(图4C),Ki67是细胞增殖的已知标志物。GGI97特征与生存率低有关,TCGA-BRCA样本中GGI97基因的中位表达证实了这一点(图4D)。为了了解GGI97基因在T细胞中的作用,作者检测了T细胞网络中与GGI97基因直接相关的前10个hub基因。此外,与Ki67直接相连的hub基因GITR(TNSFR18)预后为阳性临床结果 (图4E)。综上所述,研究结果表明,scHumanNet可以将癌症预后特征反卷积到细胞类型中,并确定特定细胞类型治疗方法的关键靶点。
图4 使用scHumanNet将乳腺癌特征反卷积到细胞类型
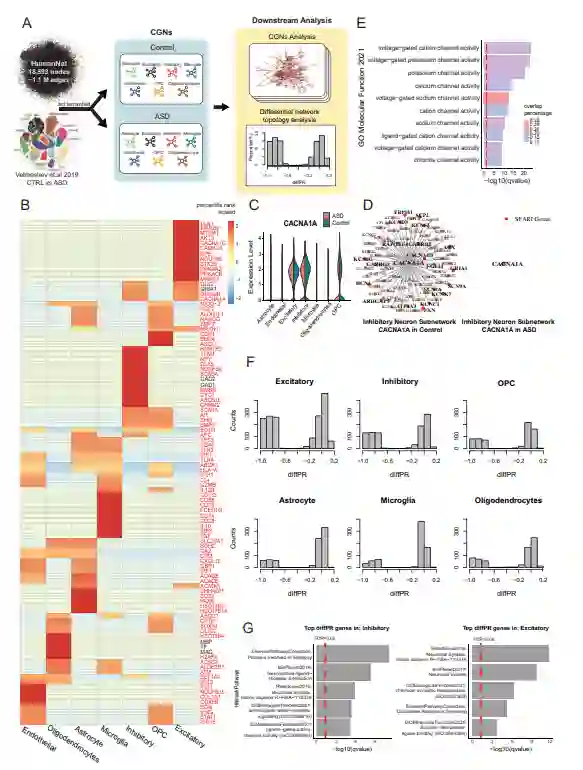
scHumanNet中使用差异中心性分析鉴定疾病相关细胞类型 scHumanNet的另一个应用是识别差异hub,即在两种生物学环境(如疾病和健康状况)之间中心性发生显著变化的基因。scHumanNet中的FindDiffHub()函数根据每个上下文特定网络的中心性程度对基因进行排序,然后识别那些与零模型相比其百分位数排名发生显著变化的基因。此外,TopDiffHub()函数允许用户提取排名前n的基因。使用scHumanNet的差异中心性分析,作者研究了具有很强遗传性的神经发育障碍ASD。作者预先假设,在疾病状态下,SFARI基因的扰动可能导致野生型分子相互作用的细胞类型特异性丧失。因此,网络中心性的降低可能指向与疾病相关的细胞类型。使用已发表的数据集(15名诊断为ASD的供者和16名匹配对照的104 559个细胞),作者构建了7个用于健康和疾病状况的CGN (图5A)。实验发现,每种细胞类型的scHumanNet hub基因与细胞类型特异性功能相关(图5B)。
作者的研究分析还揭示了许多基因在对照和疾病条件之间的网络中心度方面存在显著差异。通过具有默认参数的FindDiffHub()评估中心性排名差异最大的基因,可发现来自兴奋性和抑制性神经元的差异中枢显著富含SFARI基因,这与主要在EC和星形胶质细胞中发现的DEG形成鲜明对比。经实验验证,其中抑制性神经元富集了过表达的SFARI基因。同样,对于CACNA1A,虽然它在抑制性神经元中没有差异表达(图 5C),但在网络中心性方面存在显著差异(图 5D),并且在ASD抑制性神经元网络中丢失了许多功能相互作用。相互作用的基因大多与离子通道相关(图5E),这表明神经调节功能,特别是在抑制性神经元中,可能因CACNA1A功能缺失突变而受损。这些结果表明,使用scHumanNet进行差异中心性分析可以揭示与疾病相关的细胞类型。
最后,作者还研究了在健康状态下具有高中心性但在疾病状态下具有低中心性的基因是否可能对有关细胞类型特异性疾病机制提供帮助。实验证明,与其他细胞类型相比,兴奋性神经元、抑制性神经元和少突胶质细胞祖细胞的功能缺失基因频率最高(图5F)。在所有细胞类型中,疾病中中心性高但健康对照中中心性低的基因的发生率较低。对神经元中丢失的hub进行基因集富集分析表明,它们的功能主要与神经元活动相关(图5G)。上述实验结果表明,在疾病状态下,这些hub基因失去了与其他基因的大部分相互作用,从而导致ASD的神经元功能失调。相比之下,在ASD网络中变得更加核心的基因并未在与神经元功能相关的通路中富集。
图5 scHumanNet对CGN的ASD和健康对照样本进行差异中心性分析
总结
单细胞生物学的一个重要目标是解决人类疾病的细胞异质性。单细胞基因表达分析可以根据疾病相关基因在特定细胞类型中的差异表达来识别疾病相关细胞类型。在本研究中,作者提出了一个基于网络分析细胞类型特异性的计算平台scHumanNet,它可以补充基于表达的方法。该平台的核心部分是构建不同细胞类型的基因网络CGN。单细胞转录组数据已被用于使用参考引导或无参考网络推断方法构建CGN。由于缺乏针对特定细胞类型的高质量和经过实验验证的基因间相互作用,因此对推断的CGN进行评估并非易事。事实上,由于从单细胞转录组数据推断基因间相互作用的假阳率很高,来自这些网络的功能假设通常是基于一组边而不是单个边。为此,作者通过在hub基因中检索细胞类型特异性基因以及相应细胞类型中功能基因的网络紧密性来验证CGN的质量。在本研究中,作者比较了从单细胞转录组数据推断CGN的各种方法,发现参考引导的方法优于无参考方法。这些结果可以通过单细胞转录组数据的噪声和稀疏特性来解释,这些特性导致了许多假阳性的基因间相互作用。此外,在两个参考引导的CGN分析平台中,scHumanNet优于SCINET。虽然他们使用了相同的网络推理算法,但他们使用了不同的参考交互作用。之前,作者已证明了scHumanNet的参考相互作用组HumanNet在预测疾病基因方面的性能明显优于其他人类基因网络,包括SCINET的参考相互作用组。这表明参考相互作用组的质量是参考引导CGN性能的关键,未来对参考相互作用组的改进将进一步改善CGN。
在这项研究中,作者展示了CGN在人类疾病基因的细胞类型特异性研究中的两种应用。首先,基于一组疾病基因在CGN上的网络紧凑性,将疾病基因的作用反卷积到细胞类型中。并且scHumanNet平台在今后有望扩展到对每种组织的所有细胞类型的疾病基因集进行系统性的细胞类型去卷积,从而生成人类细胞图谱数据的CGN。其次,利用CGN根据不同细胞类型疾病和健康状况之间的差异中心性来识别与疾病相关的细胞类型。因此,scHumanNet平台可以分析差异hub基因。
scHumanNet有一些局限性。尽管研究结果表明,参考引导方法产生了更多生物相关的CGN,但它的代价是无法发现特定于细胞类型的新相互作用。此外,细胞类型反卷积对于一小组基因(例如一组三个基因)可能不可靠,因为网络紧凑性的统计检验需要相对大量的基因来确保足够的置信度。
总之,作者提出的单细胞网络生物学计算平台scHumanNet,能够解决疾病相关基因功能的细胞异质性。scHumanNet可以将疾病基因集的功能作用反卷积到细胞类型中,并通过CGN的拓扑分析识别疾病相关的细胞类型。这些结果表明,scHumanNet将促进人类疾病基因细胞类型特异性的研究,从而推进精准医疗的发展。 参考资料 Junha Cha, Jiwon Yu, Jae-Won Cho, Martin Hemberg, Insuk Lee, scHumanNet: a single-cell network analysis platform for the study of cell-type specificity of disease genes, Nucleic Acids Research, 2022;, gkac1042. https://doi.org/10.1093/nar/gkac1042.
数据和代码 https://github.com/netbiolab/scHumanNet