编译|程昭龙
审稿|林荣鑫,王静 本文介绍由美国斯坦福大学医学院干细胞生物学与再生医学研究所的Sean M. Wu通讯发表在 Nature Communications 的研究成果:在单细胞RNA测序分析中,由于细胞表现出复杂的多层身份或过渡状态,导致对数据集的精确注释成为主要挑战。因此,作者提出了一个高度精确的机器学习工具devCellPy,它能自动预测跨复杂注释层次结构的细胞类型。为了证明devCellPy的强大功能,作者从已发布细胞的数据集中构建了小鼠心脏发育图谱,并训练devCellPy生成心脏预测算法。该算法可以在多层注释和小鼠发育数据中达到高预测精度。最终研究表明,devCellPy是一个可跨复杂细胞层次结构、物种和实验系统进行自动细胞预测的工具。
1 简介 在过去的十年中,单细胞RNA测序(scRNA-seq)技术为调控胚胎发育、细胞特性和疾病状态的转录环境提供了前所未有的见解。但随着实验中获得的细胞数量持续增长,生物信息学家通常需通过无监督聚类和手动细胞类型分配的费力过程来识别数千个细胞。此外,人工细胞类型指定可能导致研究组间细胞注释的高可变性,以及实验间细胞识别的低再现性。
为了应对这些挑战,研究人员利用参考数据集将注释映射到新收集的数据上,开发了用于识别细胞类型的信息学工具。虽然这些工具提供了强大的注释算法,但依然缺乏全自动化的过程对跨复杂注释层次的细胞进行分类,其中这些细胞表现出多个身份子类或时间限制的细胞类型。例如,在从发育中的胚胎收集的scRNA-seq数据中,细胞表现出动态和短暂的细胞状态以及更精细的细胞特征。在训练预测模型时,使用不考虑时间变量的算法自动分配细胞标识时,某些细胞标识可能只在已定义的发展时间点出现,这就造成了额外的挑战。此外,如果没有对数据进行大量的子聚类和重新计算新的降维特征空间,这些更精细的特征通常是无法检测到的。自动细胞预测算法通常要求用户构建单独的参考模型,以实现对高粒度细胞亚型的注释。这一挑战在发育数据集中尤为突出,其中细胞类型存在于发育的有限时期内,因此,使生成统一预测模型复杂化。
为了解决这些挑战,作者提出了基于Python的发育细胞预测(devCellPy)工具,用于从任何组织或物种获得的高度复杂的注释层次结构中自动预测细胞身份。devCellPy预测模型的基础是极端梯度提升(XGBoost),这是一种有监督的机器学习方法,通过使用一系列梯度增强的决策树集成来学习创建精确预测所需的输入特征集。DevCellPy通过学习特定参考数据集的注释层次结构和创建预测模型以全自动化的方式跨所有注释层对细胞进行分类,其在细胞标识的自动化分配方面取得了重大进展。重要的是,该算法允许将时间点变量合并到注释层次结构中,因此,可以对限定时间段内出现的细胞标识进行分类。
为了证明devCellPy在多层细胞预测方面的能力,作者从多个公开的scRNA-seq数据集中构建了一个单一的大规模心脏发育细胞图谱。因为在心脏发育过程中存在非常复杂的不同细胞类型注释,并且心脏内的细胞类型表现出随时间变化的基因表达模式,所以作者在心脏发育数据集上测试了devCellPy的预测能力。实验结果表明,该算法在多个层面(包括时间受限的细胞群)都具有高度准确性的预测能力,这进一步证明了devCellPy算法在跨物种预测心肌细胞类型方面的广泛适用性。
2 结果 devCellPy 能够生成用于细胞类型和亚型分类的多层预测算法
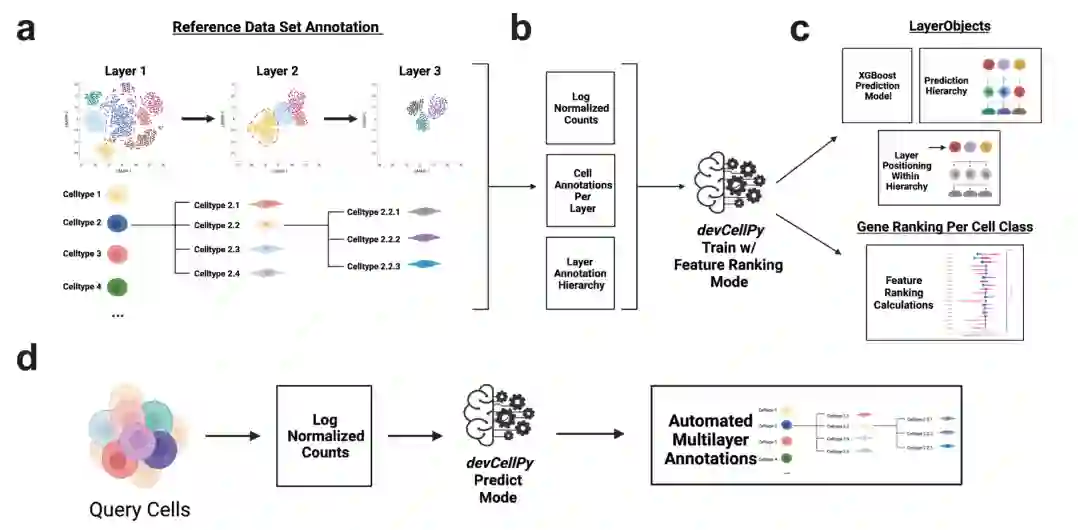
为了解决以分层方式自动分类细胞类型和亚型的挑战,作者构建了一种基于Python的软件包devCellPy,它可用于生成包含时间元素的细胞身份预测算法。devCellPy由训练和预测步骤组成。在训练期间,一个包含多层注释的参考数据集被用于训练算法(图1a)。用户为devCellPy提供一个注释层次结构,其指定了数据集中的多个层和细胞类别,包括用于构造细胞类型和时间相关的注释层次结构的时间点变量。此外,用户为参考数据中的所有细胞提供一个对数规范化表达式矩阵,以及包含跨层次结构所有层的单细胞注释的元数据表(图1b)。作者在devCellPy中引入LayerObject类来创建一个有组织的数据结构,其中算法学习数据集的注释层次结构,并包含层次结构中每个层的位置信息(图1c)。该系统允许跨层次结构的正确分支自动分类细胞亚型,并为层次结构的每一层训练一个XGBoost预测模型,并将其存储在该层各自的LayerObject中。
除了LayerObjects,作者还在devCellPy中实现了最近开发的Shapley Additive explained (SHAP)算法。SHAP有助于devCellPy输出在进行细胞类型分类的训练过程中自动识别的基因标记,从而突出显示用于对感兴趣数据集中的细胞类型进行分类的主要阳性和阴性基因标记(图1c)。在使用参考数据集训练devCellPy之后,用户可以使用devCellPy生成的预测算法,通过导出一个直接加载到算法中的对数归一化计数矩阵来对查询数据集进行分类(图1d)。devCellPy将自动读取矩阵文件,并使用存储在LayerObjects中的预测模型来输出查询细胞的多层细胞类型和子类型预测。除了提供输出注释之外,devCellPy还将为每个分类的细胞输出概率度量,以向用户提供有关算法在进行细胞预测时的可信度信息。总体而言,该算法的结构允许用户从任何模型生物的任何组织获得的任何参考scRNA-seq数据集来创建训练模型,并可以轻松导出这些模型,以对类似细胞类型和亚型的新数据集进行预测。
图1 devCellPy结构图
大规模心脏发育图谱的构建
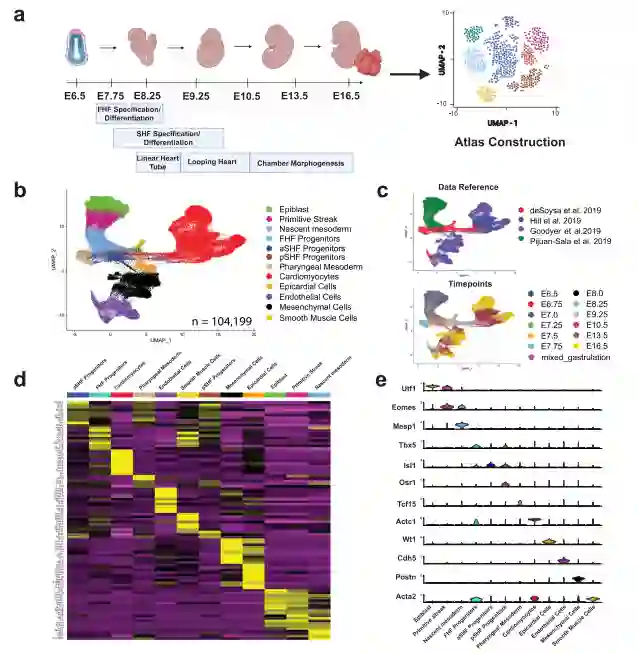
为了测试devCellPy在生成用于进行多层细胞注释的高精度预测算法方面的性能,作者首先从四个公开的数据集中集成了一个中胚层来源的心脏发育细胞类型的大规模scRNA-seq图谱,经过整合的数据集可观察到从早期原肠胚形成到主要心脏细胞类型的清晰发育轨迹(图2a)。
通过关注中胚层衍生的细胞类型,作者观察到从多能外胚层细胞开始的分化树,通过原始条纹过渡到早期新生的中胚层祖细胞,并进展为心脏祖细胞(图2b)。尽管这些数据集来自四个不同的来源,但经过成功地整合所有数据集,可以观察到12种主要细胞类型在整个发育时期的聚类(图2b, c)。UMAP图显示了发育一致的结构,心脏祖细胞组成树干,随后分裂成不同的细胞类型。作者还通过对所有主要标注的细胞类型进行差异基因表达分析,进一步验证了构建图谱中分配的注释,并证实了已报道的12个主要细胞群的主要细胞标记的独特表达(图2d,e)。
图2 中胚层来源心脏发育细胞图谱的构建
devcellPy生成的算法可以精确预测注释层复杂层次结构中的细胞类型
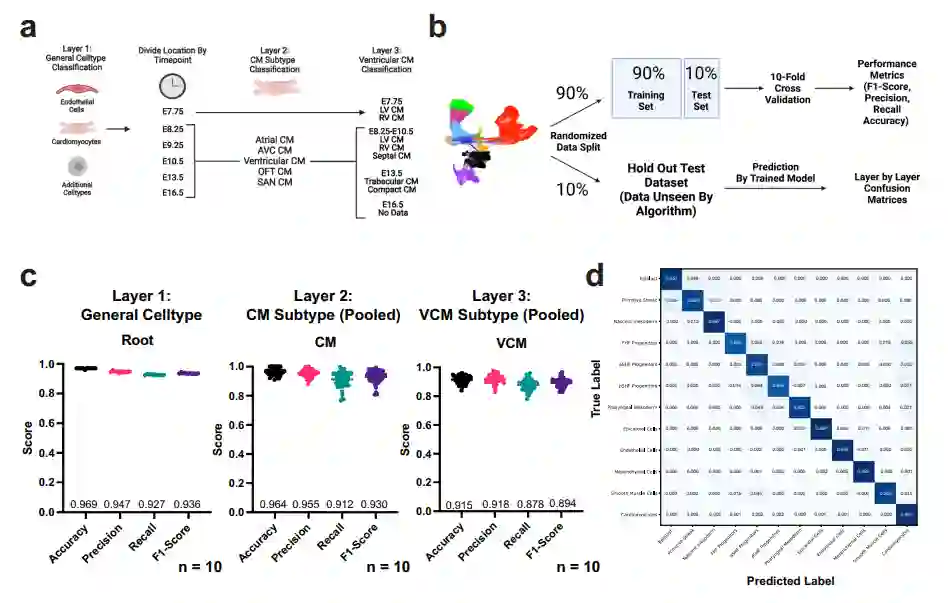
在建立了包含多层注释的大型心脏发育图谱后,作者继续测试了devCellPy在该数据集上生成高度精确的细胞识别预测算法的能力。通过对心肌细胞图谱进行多层分析,其中顶层代表广义细胞注释,其次是心肌细胞亚型,以及在发育过程中按时间点划分的心室心肌细胞亚型(图3a)。为了测试算法的分类性能,作者将数据随机划分为90%和10%的分区,分别用于交叉验证和保留数据集测试(图3b)。实验结果显示,对于第一层注释,该模型在 10 轮独立训练中显示出高整体准确度(图 3c)。同时,devCellPy 对10% 保留数据进行了分类(这些分类在训练后的模型中未发现),这证实了devCellPy高度准确的预测(图 3d)。
对心室心肌细胞亚群的心肌细胞预测算法性能的进一步分析表明(图 3c),实验观察到的性能指标在第一层注释上得分最高,而在注释层次结构较低级别中发现的密切相关的细胞类型中略有下降。为了进一步评估devCellPy的性能精度,作者还将该算法的性能与先前发布的单细胞预测算法(CaSTLE、SeuratV3、scmap、SingleCellNet)进行了比较,结果表明,devCellPy在所有评估的细胞类型中显示了最高的整体精度。
图3 devCellPy在各层之间的性能评估
devCellPy识别生物相关的细胞标记
为验证devCellPy生成的心脏预测算法是否可以识别与发育相关的基因,以便跨细胞层进行预测。作者通过使用devCellPy中的SHAP算法,确定了测试的每一层注释中细胞类型的主要阳性和阴性预测基因。对于第一层,算法自动识别出先前验证过的细胞类型预测因子。在实验中,一般的细胞类型预测因子可以揭示已知细胞类型的主要阳性预测因子,而除了识别一般细胞类型的典型标记外,SHAP还可以识别其他细胞类型的标记。
除了在第一层中识别一般细胞类型的主要标记外,作者还确定了用于在多个分化时间点识别心肌细胞和心室心肌细胞亚型的主要基因。并且,devCellPy还跨时间点识别了多个先前发表的心肌细胞亚型标记,同时也以特定的时间点方式识别了独特的标记。
devcellPy生成的算法从de novo数据集中准确预测细胞类型
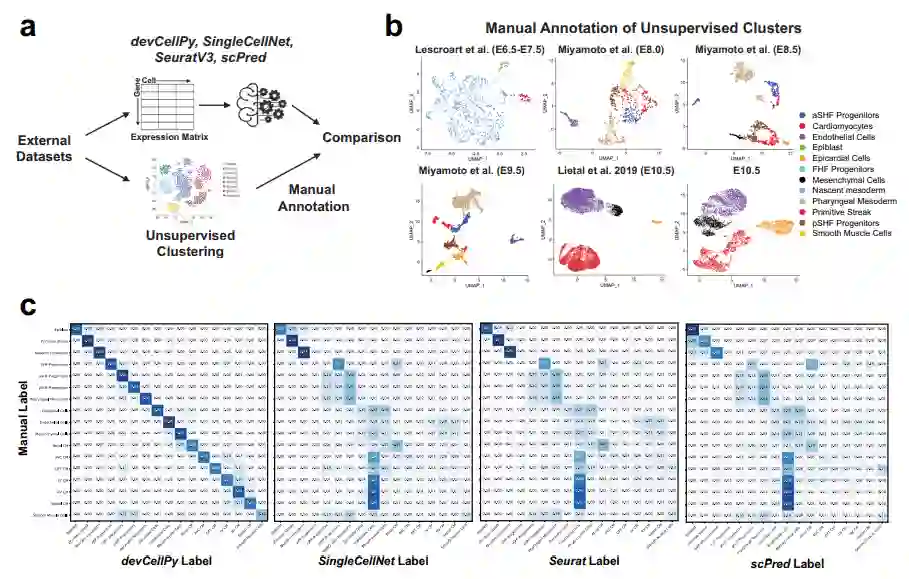
为了进一步评估devCellPy的性能,验证devCellPy生成的预测算法是否能够成功预测以前数据集中没有遇到过的细胞类型。作者分析了来自三个新来源的scRNA-seq数据,并从E10.5小鼠心脏中生成了新的数据,并测试了devCellPy对心脏图谱中存在的所有细胞类型进行全自动预测的能力。同时,作者进一步评估了其他细胞预测算法对心脏图谱中所有细胞类型进行完整分类的能力(图4a、b)。作者将机器学习分类与查询数据集的无监督聚类注释期间分配的手动注释进行了比较(图4b),手动注释和devCellPy预测的比较显示,两种注释方法在17个细胞类别中的11个中具有高度的一致性,准确率达80%(图4c)。
与之前发表的其他机器学习方法相比,devCellPy在心脏图谱中所有细胞类型的分类方面优于SingleCellNet、Seurat和scPred。所有其他方法对外胚层、原始条纹和新生中胚层的人工方法显示出高度的一致性,但在其他细胞类型中观察到的预测准确性较低。并且,在密切相关的细胞类型中,如心脏祖细胞、心外膜和间充质细胞以及心肌细胞亚型,其他方法在人工方法和机器学习预测之间显示出低一致性(图4c)。总体而言,实验结果证明了devCellPy生成的预测算法可以准确预测de novo数据集中的细胞类型,并在对高粒度细胞类型进行预测时优于其他方法。
图4 devCellPy预测新的scRNA-seq数据
devCellPy生成的心脏预测算法揭示了hiPSC衍生心肌细胞的发育不成熟和心室特异性心肌细胞分化
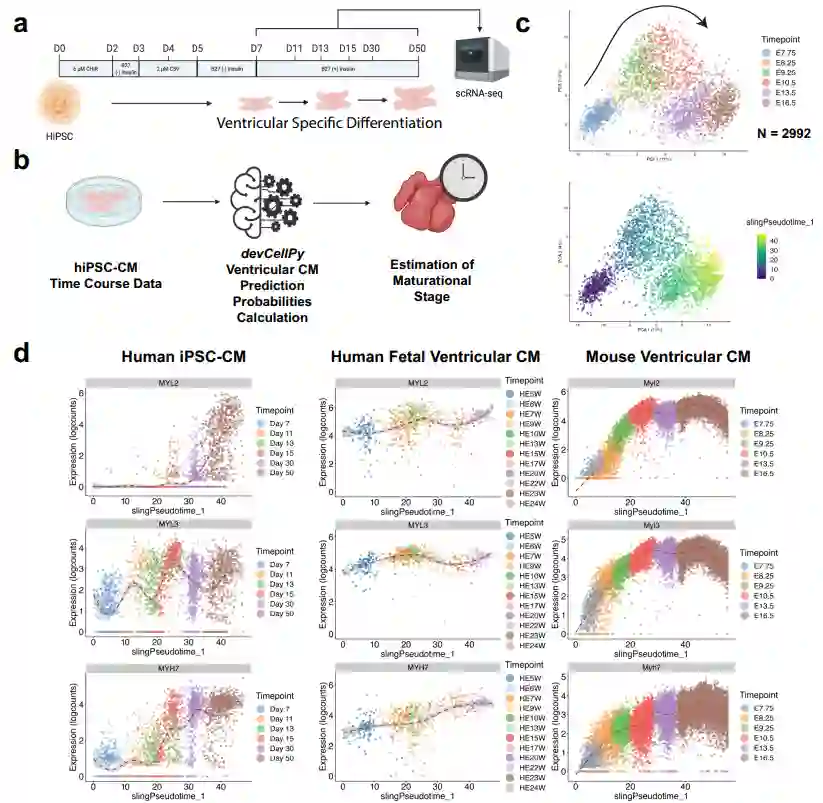
在验证了devCellPy对胚胎小鼠心肌细胞分类的准确性后,作者研究验证了是否可以使用小鼠心脏预测算法来准确预测人诱导多能干细胞(hiPSC)衍生的心肌细胞的类别(图5a, b)。鉴于已知的体外衍生心肌细胞不成熟,作者验证了早期胚胎小鼠模型是否能更好地预测 hiPSC 系统中的心肌细胞亚型。通过对六个时间点的心肌细胞进行分析,并绘制了前两个主成分的单细胞图。正如预期的那样,细胞以时间依赖性方式沿着第一个主成分前进(图5c)。此前有报道称,小分子双相WNT协议在缺乏后向视黄酸信号的情况下主要产生心室特异性心肌细胞。为了证实这一点,作者绘制了在hiPSC、人类胎儿和小鼠胚胎发育过程中经过验证的心室标志物Myl2、Myl3、Myh7的表达图,并证实了所有这些确定的心室标志物的表达逐渐增加(图5d)。为继续验证基于小鼠数据训练的devCellPy模型是否能够准确识别hiPSC-CMs心室特性,作者基于E7.75和E13.5之间的早期小鼠胚胎时间点训练了多个devCellPy预测模型,用于识别心肌细胞亚型。然后使用基于时间点的模型计算devCellPy的预测置信度,以确定hiPSC-CMs心室特性。实验结果表明,hiPSC-CMs表现出发育不成熟,并且hiPSC-CMs的胚胎表型和小鼠心肌细胞成熟基因表达程序具有密切保守性。
图5 devCellPy小鼠预测模型确定了心室优势HiPSC-CMs的发育不成熟
3 总结 在这项研究中,作者开发了一个基于Python的生物信息学管道devCellPy,它可以使用精确的高分辨率参考数据集,自动预测多个发育时间点和级联注释层的细胞类型。分层注释结构存在于多个器官、物种和模型系统之间。正如此处介绍的心脏发育图谱所示,细胞表现出多个身份子类。此外,对数据集进行精确的手动标注通常需要有对阳性和阴性表达标记的专业知识,这可能导致经验不足的用户,在数据集标注过程中的可再现性较差。以前的研究也表明,包含多个子类的高度相关细胞类型的深度注释数据集,可能会对自动细胞分类器的性能产生负面影响。作者通过创建分层组织的预测模型来解决这一问题,该模型在 “LayerObject”对象类的注释层次结构中编码它们的位置。通过将密切相关的细胞类型的预测分解为不同的注释层,从而在注释层次结构中的高粒度细胞子类之间实现更高分辨率的预测结果。重要的是,用户可以在层次结构中指定时间点,从而允许在跨注释的多个子层中构建时间点相关预测。随着细胞在预测层次结构中移动,devCellPy将只对用户设置的概率阈值的细胞进行下一层分类,从而允许对跨每个分类层的细胞类型进行高置信度注释。此外,devCellPy中的LayerObjects具有高度可移植性,允许用户共享训练过的预测模型,或导出单个LayerObjects进行单层预测。devCellPy的可移植性允许科学界广泛使用专家策划的参考图谱,以在复杂场景中进行细胞预测。
除了可移植性之外,devCellPy还需要对数据集进行处理以进行训练和预测,devCellPy可以识别超过35000个输入基因中的生物学相关标记基因。该算法的高预测性能表明,除了归一化计数表达式矩阵外,不需要额外的预处理或特征选择来从精确的高分辨率参考数据集中训练算法和参考标签。当将devCellPy的性能与其他之前发表的细胞分类算法进行比较时,devCellPy的自动化分层方法在分类心脏图谱中存在的所有细胞类型方面优于其他分类训练方法。同时,devCellPy完全自动化了跨多层细胞标识的训练,从而自动化了跨复杂注释层次结构预测细胞的过程。此外,虽然其他方法允许为一组独特的标签生成细胞预测模型,但devCellPy允许跨时间点相关注释自动分类,从而为跨发育数据集的细胞分类提供了显著的改进。
devCellPy为scRNA-seq分析提供了一个重要工具,它为生成细胞类型/亚型预测算法提供了一个全自动化的管道,且该算法非常适用于分层注释的数据集。作者的工作表明,devCellPy生成的算法具有高度通用性,可推广到任何scRNA-seq数据集,并提供了一个完全开源的Python包。随着大规模发育细胞图谱的发展,devCellPy将提供资源来帮助识别跨平台和物种的细胞类型,特别是在注释良好的参考数据集中显示复杂的多层注释方案。
参考资料 Galdos, F.X., Xu, S., Goodyer, W.R. et al. devCellPy is a machine learning-enabled pipeline for automated annotation of complex multilayered single-cell transcriptomic data. Nat Commun 13, 5271 (2022). https://doi.org/10.1038/s41467-022-33045-x
数据链接:
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE184943 https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE165300 https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE126128 https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE131181
代码链接: