编译|王娜
审稿|王海云本文介绍由美国耶鲁大学计算机科学系的Smita Krishnaswamy通讯发表在 Patterns 的研究成果:为了同时分析多个组学数据中的信息,**作者提出了一个叫做单细胞多模态生成对抗网络(scMMGAN)的框架,该框架将来自多种模态的数据整合到环境数据空间的统一表示中,并结合对抗学习和数据几何技术进行下游分析。该框架的关键改进是一个额外的扩散几何损失,它使用一个新的内核来约束原本过度参数化的GAN。**作者证明了scMMGAN有能力在各种数据模式上产生比其他方法更有意义的结果,并且其输出可用于从现实世界的生物实验数据得出结论。
1 简介 整合不同来源的数据是计算基因组学的一个关键挑战。目前已经出现了几种单细胞技术,包括RNA-seq、ATAC-seq、Hi-C、ChIP-seq和CITE-seq等。在这些模式中,只有一部分可以作为同步测量——往往有质量下降的因素,如基因规模减少、吞吐量降低和噪音的增加。其余的测量必须在同一群体中不同的细胞子样本上完成。这就是本文中要解决的关键问题:预测缺失的或不同时的模态,以产生一套更完整的特征。
作者提出的方法基于循环一致的生成对抗网络(CycleGANs)框架。在基于GAN的领域适应框架中,生成器网络被训练来将一种模式的数据点映射到另一种模式的数据点中。在训练过程中,一个判别器被用来确保生成的点是从代表第二种模式的高维分布中采样的。在CycleGANs中,有两个背对背的生成器,一个从第一模态到第二模态,另一个从第二模态到第一模态。重构误差使两个背对背领域适应的结果再次成为原始分布,也就是说,生成器在数据空间中存在训练点的区域上彼此相反。
作者在广泛的数据类型上展示了用scMMGAN对齐数据模式的能力。首先在有同步测量的数据集上对其进行验证,并在评估中使用这些数据作为基本事实。然后作何使用scMMGAN对一个新的三阴性乳腺癌数据集进行了彻底的调查,作者将乳腺癌培养物HCC38的细胞异种到小鼠体内,并允许其从原发肿瘤位置转移到次发肿瘤位置。结果表明,scMMGAN可以推断出细胞结构的空间位置。
2 结果 scMMGAN模型结果
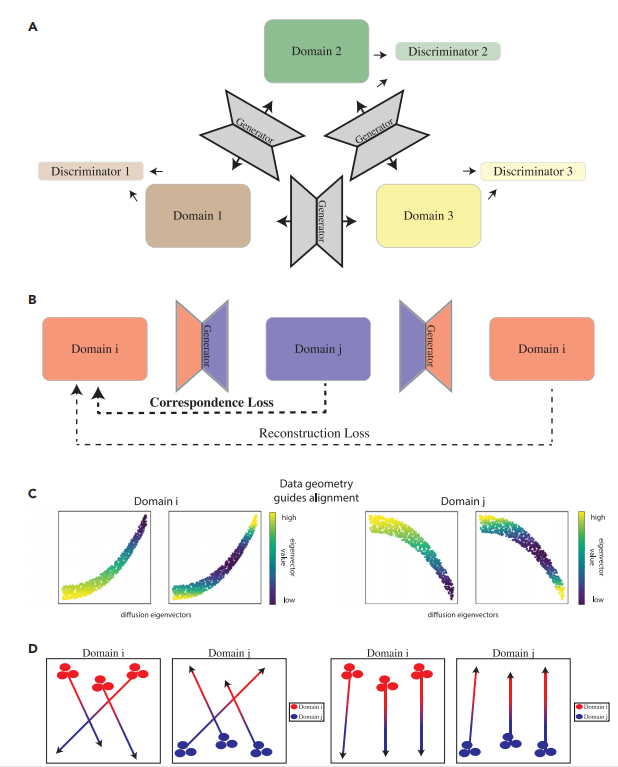
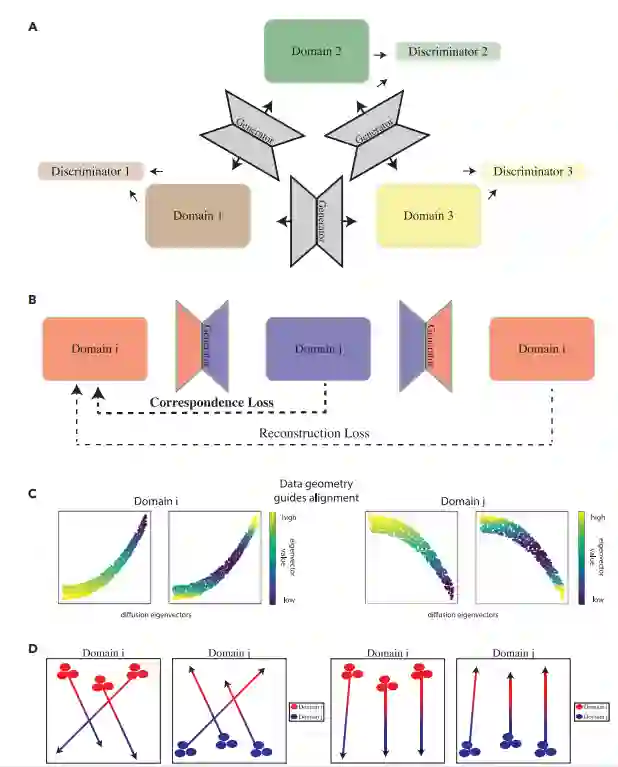
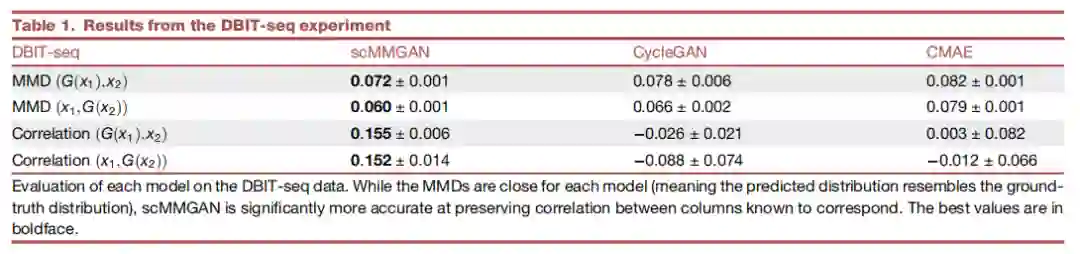
图1A描述了scMMGAN的框架。每一对数据域或模态都有一对生成器网络,在它们之间进行双向映射,形成一个多样化的多模态映射。重建损失是原始数据与域i和j之间的两个配对生成器组成的平均平方误差(MSE)。除了判别器指导生成器将其输入模态转化为输出模态的这一损失外,损失中还有两项,以确保学到的映射是有信息和意义的。这些在图1B中被描述出来。
然而,简单地匹配概率分布会导致细胞状态的不连贯性(图1D)。在这里,作者给出的一个关键见解是,分布必须只在对应的约束条件下进行匹配。这些对应关系本质上是反映在每一种模式中的基础系统的不变性。在以前的工作中,作者为每个数据集使用定制的对应关系损失。然而,在这里需要注意到,在匹配单细胞数据时,可以使用一个几乎通用的约束,那就是流形几何保存。
图1 scMMGAN架构和对应的损失
实验结果,空间、scRNA-seq和蛋白质组学数据之间的映射
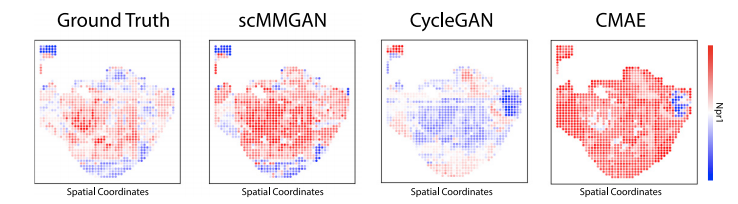
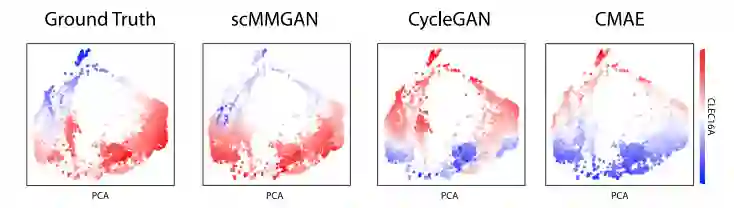
图2 DBIT-seq实验的结果比较
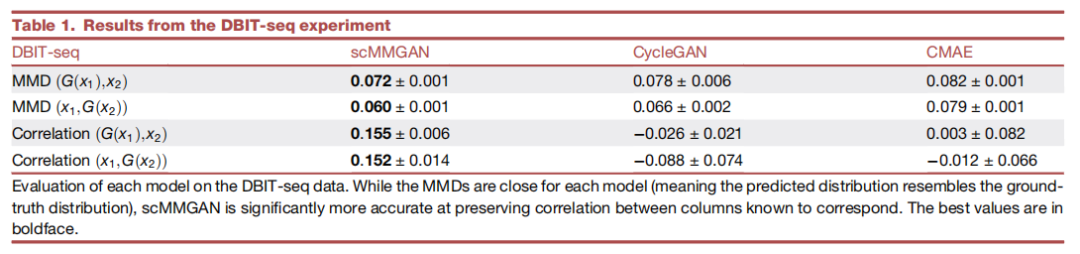
作为scMMGAN的初步验证,作者利用新开发的组织测序DBIT-seq中同时测量的多模态数据作为基本事实。作者将空间定位的scRNA-seq数据和空间定位的蛋白质数据视为两个独立的测量,并学习在它们之间进行映射。然后,利用它们是共同测量的这一事实,以及每个数据集中的一些列是相关的(对应的基因和蛋白质)来评估所学映射的准确性。图2显示了scMMGAN和基线模型在这些数据上的示例结果。结果表明scMMGAN对真实数据的建模效果最好。为了量化通过排列组合保留个体观察信息,作者使用了已知对应于同一基因的转录组空间和蛋白质组空间的列之间的相关性。由于这些值对应于同一个基因,作者期望在映射前的点对应的值和映射后的值之间有一个相关性。这些分数从数量上证实了作者在这些实验中看到的结果(表1)。所有的模型都能够准确地匹配目标分布(低MMD),每个模型的一个或两个SD区间重叠组成的性能非常相似。然而,当观察保留的相关性时,可以看到scMMGAN实现了最好的对齐,已知对应的列之间的平均相关性为r=0.154。
表1 DBIT-seq实验的结果
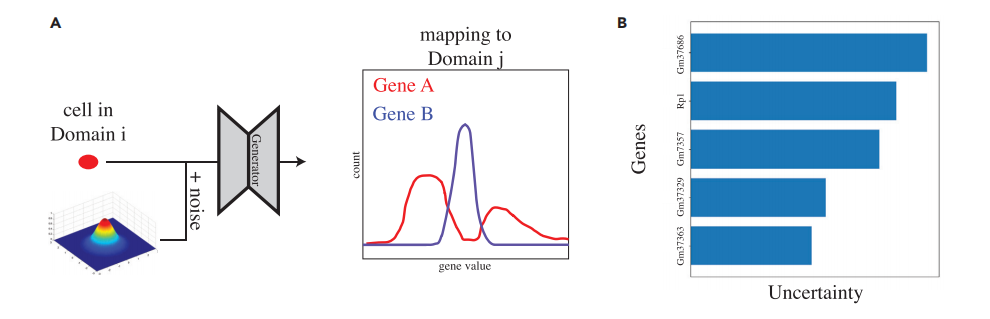
作者设计了如图3所示的实验以测试DBIT-seq数据中的转录组和蛋白质组测量结果。正如预期的那样,作者发现有类似蛋白质组学测量的基因的平均方差是0.026,而没有类似蛋白质组学测量的基因的平均方差是1.419。这是一个合乎逻辑的结果,因为转录本计数与相应的蛋白质组测量之间的关系更容易建模,因此可以更有把握地进行。这证实了作者对这种多模态环境下的工作过程的理解。
图3 不确定量化实验的设计
scRNA-seq和ATAC-seq数据之间的映射关系
接下来作者对由成对的 ATAC-seq和RNA-seq在同一细胞的测量设计了实验。作者使用的数据集来自于一个公开的人类血液数据集,该数据集是通过细胞分选从健康捐赠者的外周血单核细胞中取出的粒细胞。图4显示了通过输出图对结果进行的定性评估,第一栏是真实的ATAC值,后面几栏是生成的相应RNA-seq值。和以前一样,scMMGAN的输出与真实结果最匹配。对于其他模型,虽然它们在分布水平上对每个基因有适当的激活量,但在点的水平上,它们的排列是不准确的。从表2中可以看出,虽然所有的模型在分布水平上都能充分匹配地面实况(从它们的低MMD中可以看出),但在对它们进行逐点评估时,可以看到明显的差异。scMMGAN的预测与地面实况的平均相关性为r=0:336,而其他模型基本上都是不相关的。
图4 结果比较:从 ATAC-seq/RNA-seq实验
表2 ATAC-seq/RNA-seq实验的结果
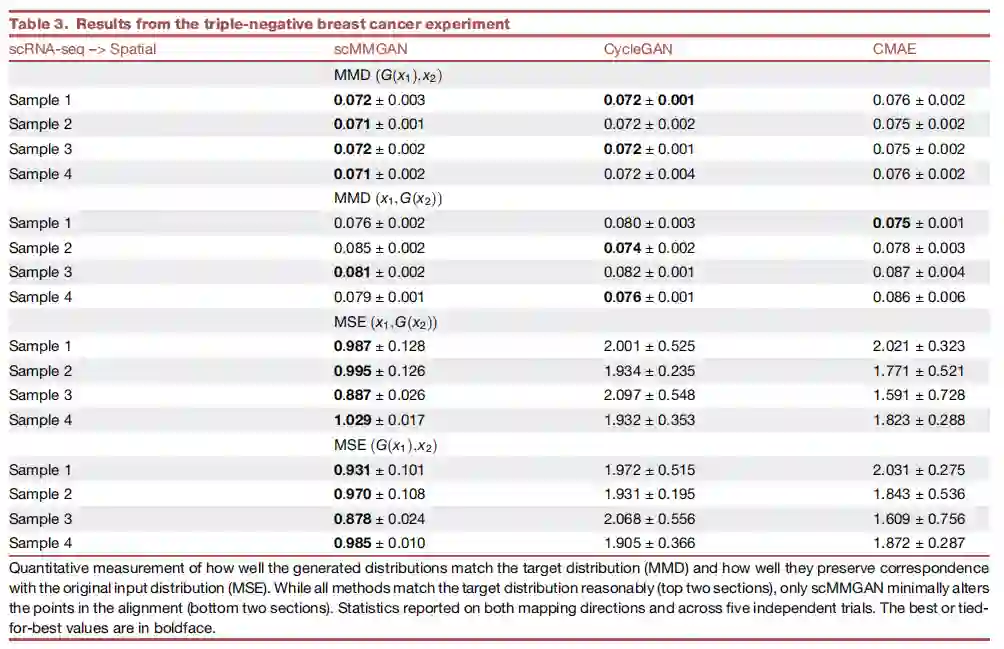
三阴性乳腺癌数据的整合
在这里,作者将scMMGAN应用于一个数据集,该数据集包括一个三阴性乳腺癌的人类异种移植模型(MDA-MB-231),其转录组的测量结果是由空间RNA-seq模式和缺乏空间信息的scRNA-seq模式共同完成的。虽然scRNA-seq和空间RNA-seq数据都是测量基因谱的转录组技术,因此它们的维度具有相同的意义,但这两个数据集不能按原样一起分析。在图5A中,可以看到在使用scMMGAN之前,这两个数据的分布是完全不重叠的。由于原始数据在联合空间中是完全可分离的,任何下游分析都只能发现两种模式之间的差异,而不能发现其中细胞之间的差异。对于使用两者信息的综合分析,只需要scMMGAN的对齐输出(图5B)。
图5 对三阴性乳腺癌数据集的scMMGAN排列和聚类分析
作者通过将空间RNA-seq,映射到scRNA-seq空间,然后对生成的scRNA-seq数据进行聚类来分析scMMGAN的排列(图5B)。正如所看到的,这些scRNA-seq簇保留了坐标中看到的空间模式,证明了该模型通过结合原始空间坐标分析生成的scRNA-seq数据得出新的空间信息结论的能力。
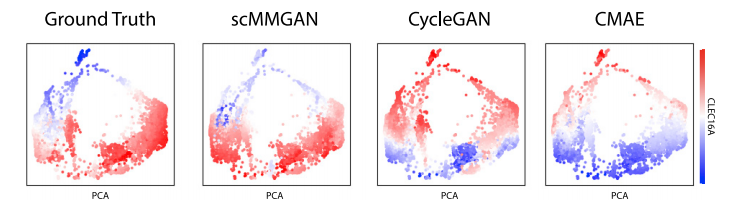
在图5C中,可以看到相反的映射方向,即采取scRNA-seq数据并用它生成空间RNA-seq。通过将scRNA-seq点映射到这些生成的坐标,可以看到特定细胞类型的空间组织。例如,在图5D中,作者为SLC2A1和CDK11A含量高的细胞绘制了生成的空间坐标,并看到这两类细胞之间的空间分化。
图6 在空间坐标上绘制生成的scMMGAN表达式值结果
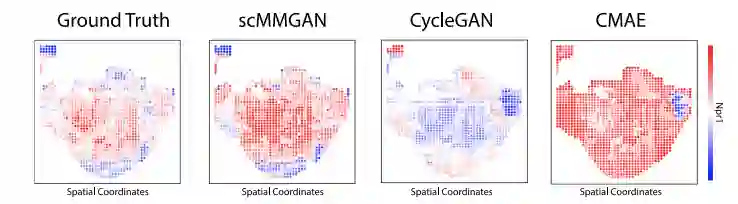
现在可以看到scMMGAN已经学会了以保留基因信号的方式在两种模式之间进行数据映射,接下来我们将其与其他排列模型进行比较。在图6中,作者显示了每个模型从空间RNA-seq到scRNA-seq的学习映射的结果。在图6的第一列中,作者绘制了空间RNA-seq数据中五个基因在原始空间坐标上的数值。
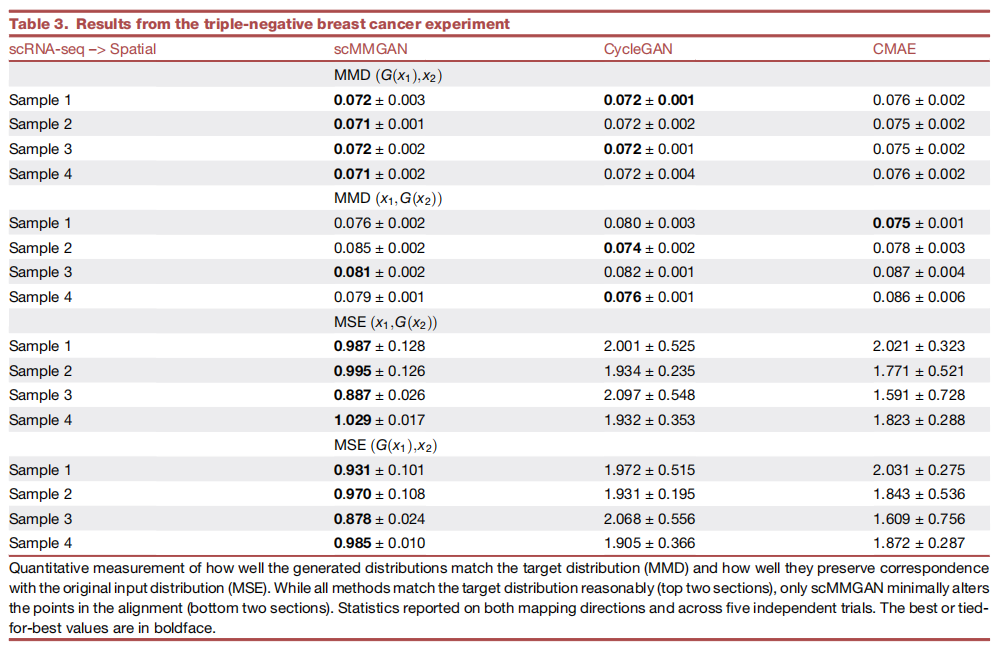
图6最下面一行显示的是RER1,展示了另一种促使scMMGAN的对应损失的典型情况。这个基因大致呈双峰分布,其中高和低的观察值数量相等。因为翻转两个种群通常就像在神经网络层的一个权重中引入一个负号一样容易,所以CMAE将该基因中高的所有空间坐标映射到该基因中低的scRNA-seq谱,反之亦然。只有在scMMGAN的对应损失下,这些同样容易学习的映射中的一个才被指定为优选,不翻转种群的那个的训练目标明显较低,而不是它相等。表3中显示的定量实验结果进一步证实了这一点。
表3 三阴性乳腺癌实验结果
3 总结与讨论 在这项工作中,作者证明了scMMGAN可以将来自相关实验但不同模态的数据以一种通过学习映射的方式最好地保留原始细胞的属性。在scMMGAN的架构中增加了对应损失,解决了在学习映射时只说明分布级损失而产生的模糊性。这适用于各种数据类型和模式、分布形状以及实际生物实验中出现的其他设置。
作者已经展示了scMMGAN如何被用来测量映射中的不确定性,并使用注入的随机性来衡量哪些信息是某个模式所特有的,哪些信息是它们之间共同的。这不仅可以用来回答关于实验中的细胞样本的问题,也可以用来回答关于技术和模式本身的优点和缺点的问题。
此外,作者还展示了scMMGAN如何被用来识别在一种模式中发现的虚假的相关关系。同样,作者展示了scMMGAN可以识别新的关联的能力,这些关联在单个模式中不可见,但当数据被映射到另一个模式中时就变得很明显。通过这些方式,scMMGAN可以被添加到传统的分析管道中,从复杂的多模态实验数据中发现进一步的见解。
**局限性:**尽管GANs在映射分布方面很有用,但它们有一些关键的缺点。首先,由于对抗性损失,它们很难训练,这可能导致不稳定。这种不稳定性意味着模型可以在训练迭代中迅速从有效恶化到无效。第二,它们经常受到模式崩溃的影响,因为它们没有受到分布级损失的惩罚以匹配整个分布。此外,在基于每个映射方向的成对生成器的框架下,必要的生成器的数量呈四级增长。这意味着,对于大量的输入模态的对齐,网络必须做得很小,或者必须单独训练。最后,基于几何学的损失不是完全的 "即插即用",因为仍然需要选择数据点之间的距离。
参考资料 Amodio, M., Youlten, S. E., Venkat, A., San Juan, B. P., Chaffer, C. L., & Krishnaswamy, S. Single-cell multi-modal GAN reveals spatial patterns in single-cell data from triple-negative breast cancer. Patterns; doi: https://doi.org/10.1016/j.patter.2022.100577
数据 https://github.com/KrishnaswamyLab/scMMGAN
代码 https://github.com/KrishnaswamyLab/scMMGAN