编译 | 杨博,王自乐 审稿 | 王海云 本文介绍美国纽约州伊萨卡康奈尔大学兽医学院贝克动物健康研究所Tinyi Chu和美国纽约州伊萨卡市康奈尔大学兽医学院生物医学系Charles G. Danko共同通讯发表在 Nature cancer 的研究成果:作者开发了贝叶斯细胞比例重构,使用统计边缘化推断(BayesPrism),一种贝叶斯方法,使用来源于患者的scRNA-seq作为先验信息,从bulk RNA-seq中预测单个细胞类型的细胞组成和基因表达。对原发性胶质母细胞瘤、头颈部鳞状细胞癌和皮肤黑色素瘤进行了整合分析,以将细胞类型组成与不同肿瘤类型的临床结果相关联,并探索恶性和非恶性细胞状态的空间异质性。作者使用排除混杂的非恶性细胞后的基因表达注释来细化当前的癌症亚型。最后确定了恶性细胞中与多种肿瘤类型的巨噬细胞浸润、T细胞、成纤维细胞和内皮细胞相关的基因表达。并且引入了一种新的视角,可以在bulk RNA-seq数据中准确地推断细胞组成和表达。
1 简介 细胞与细胞之间的相互作用非常复杂,在生物环境中会强烈影响细胞行为,通常会产生医学后果。
两层信息对于理解肿瘤组成至关重要:(1)每种细胞类型的比例;(2)每种细胞类型中的基因表达水平。单细胞RNA测序(scRNA-seq)技术的兴起最近使TME内单个细胞转录组的全基因组测量和异质性表征成为可能。然而,scRNA-seq的成本和对高质量组织的要求限制了可检测的患者样本数量。此外,scRNA-seq易受细胞捕获技术偏差的影响,这会干扰细胞类型组成的恢复。
作为一种替代方法,细胞类型丰度可以通过对一组任意定义的标记基因构建的参考表达矩阵进行回归,从大量RNA-seq数据推断。然而,现有的反卷积方法对参考样本与体样本的分布差异做了限制性的假设。这些假设经常被大量数据和参考数据之间的技术和生物学差异阻碍。因此,现有的方法未能解决下述关键问题:在TME中,恶性细胞如何影响非恶性细胞的组成,哪些基因与这些相互作用相关?
因此作者提出了名为BayesPrism的贝叶斯模型,使用scRNA-seq作为先验信息,通过bulk RNA-seq数据联合推断细胞类型的部分后验分布和基因表达。通过明确建模和边缘化单细胞参考数据和bulk数据之间的基因表达差异,BayesPrism在肿瘤和非肿瘤设置的细胞类型分数推断方面大大优于现有的方法。并且作者使用了一个包含胶质母细胞瘤(GBM)、头颈部鳞状细胞癌(HNSCC)和皮肤黑色素瘤(SKCM)的大样本数据集,其中包含1412个批量RNA-seq和85个scRNA-seq样本,证明了该方法的有效性。
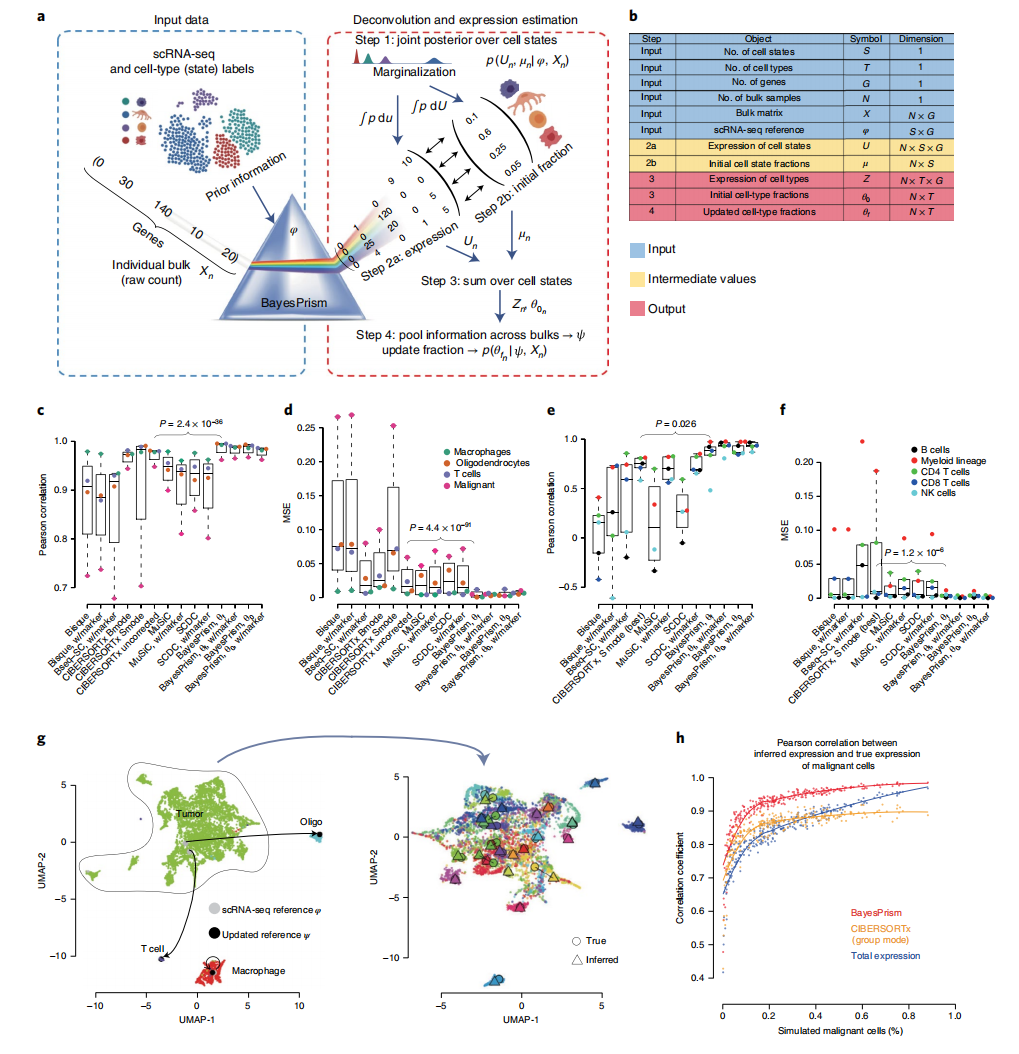
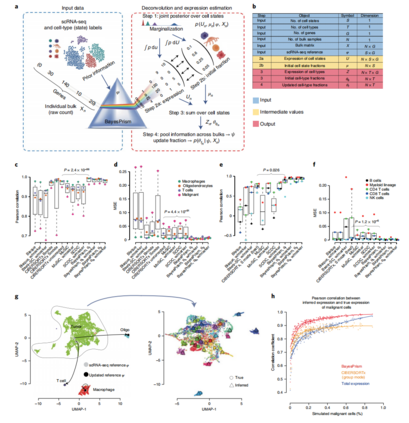
2 结果 细胞类型部分和基因表达的贝叶斯推断 BayesPrism使用一个参考的scRNA-seq来推断每个bulk RNA-seq样本的两个统计数据:(1) 从每个细胞类型获得的读数比例,假设它与该细胞类型的比例成正比;(2)每种细胞类型的基因表达水平(图1a,b)。细胞去卷积最具挑战性的方面是考虑各种不确定性的来源,包括技术和生物批次的变化,在bulk和参考scRNA-seq之间的基因表达。为了解释这些不确定性,BayesPrism采用贝叶斯策略,使用scRNA-seq对先验分布进行建模,利用每个观测数据,推断出每个细胞类型和大量样本中细胞类型比例和基因表达的联合后验分布。因此,每次估计的不确定性都可以从联合后验中剔除。
BayesPrism提高了细胞类型去卷积的准确性 为了评估BayesPrism是否在更现实的环境中提高去卷积性能,接下来作者通过在三种不同的环境中结合来自单细胞的读数来生成伪bulk数据,(1) 外周血单个核细胞(PBMC)和小鼠大脑皮质样本;(2) 在三种人类癌症类型的数据集中进行遗漏测试;(3) 使用不同的测序平台从不同的队列中产生的GBM数据集(图1c,d)。使用PBMC scRNA-seq数据作为参考,BayesPrism获得了比其他去卷积方法更准确的五个细胞类型的估计(MSE上,相关系数上P<0.03)(图1e,f)。综上所述,这些基准表明,BayesPrism 改善了现实环境中的解卷积性能。
BayesPrism估计未观察患者的基因表达 作者估计了28个GBMs的SMART-seq2伪bulk数据中的细胞类型和基因表达。并使用了来自8个GBMs的基于microwell的scRNA-seq参考数据,在bulk RNA-seq和scRNA-seq参考数据之间存在生物和技术差异的情况下,测试了BayesPrism的准确性。伪bulk样本(ψmal)中恶性细胞的基因表达估计与已知的基础真理高度相似(图1g)。对肿瘤来说,BayesPrism基因表达估计值和已知的真实值之间的相关性>0.95,纯度大于50%(图1h)。使用BayesPrism进行基因表达估计比使用CIBERSORTx或无反卷积的bulk肿瘤更准确(图1h)。
图1 BayesPrism算法流程及性能验证。
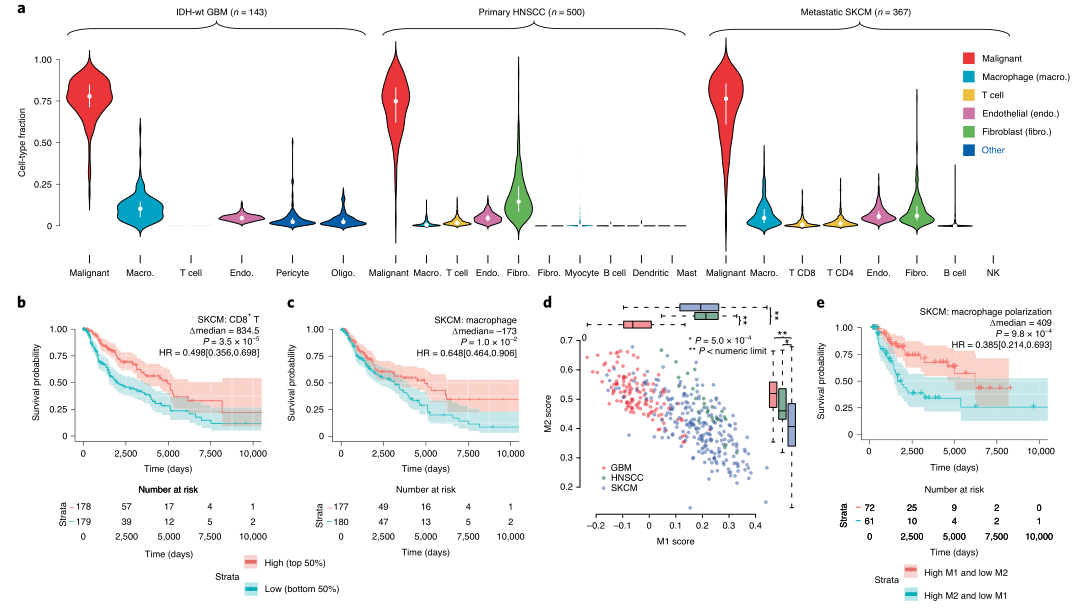
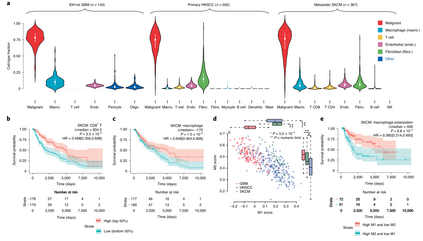
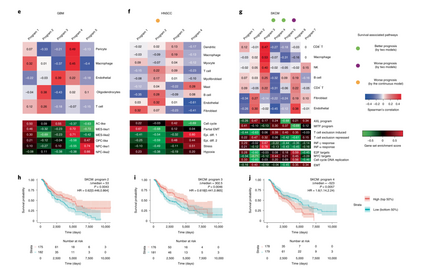
浸润性免疫细胞类型和状态对生存的影响。 作者分析了来自三种肿瘤类型GBM、HNSCC和SKCM16-18的1142份肿瘤基因组图谱(TCGA)样本的细胞类型比例。为了保持尽可能高的准确性,作者在每个去卷积任务中使用了来自相同肿瘤类型的scRNA-seq参考。利用这些参考数据集,提供了6种GBM细胞类型的估计,10种HNSCC细胞类型,8种SKCM细胞类型(图2a)。作者发现CD8+ T细胞与生存有更强的相关性,与以往报告一致。在HNSCC中,T细胞比例也与更好的临床结果相关,但仅在将细胞类型丰度作为连续变量处理的模型中,效果显著(P = 0.001, Wald检验) (图2b)。图2c表明BayesPrism估计的巨噬细胞与SKCM患者的生存呈正相关(P = 0.01, log-rank检验)。作者使用BayesPrism来评估含有5%巨噬细胞的样本中巨噬细胞特异性基因的表达。作者比较了巨噬细胞表达与两个巨噬细胞亚群特征的标记基因M1和M2,这两个亚群被认为在TME中有不同的作用。来自GBM的巨噬细胞M2评分最高,M1评分最低,而来自SKCM的巨噬细胞M2评分最低,M1评分与来自HNSCC的巨噬细胞相当(图2d)。在SKCM中,巨噬细胞极化与生存率有极强的相关性(图2e)。
图2 三种TCGA肿瘤的预后与非恶性细胞的细胞类型比例或细胞状态的关系。
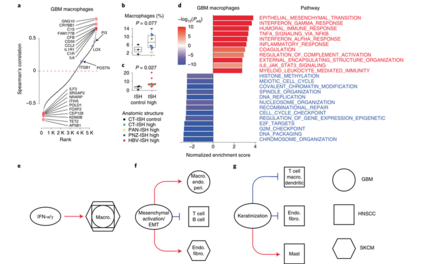
基因表达模式与TME细胞类型相关 作者利用利用BayesPrism,同时实施两个额外的过滤器,发现相互作用基因POSTN、ITGB1和LOX(图3a)均与巨噬细胞浸润具有显著正相关。尽管IVY GAP数据集中每个标记的样本量有限,但作者在PI3和POSTN的ISH阳性切片中观察到了更高的巨噬细胞含量,这是通过至少10个ISH实验分析的唯一有两个通过过滤器的基因(图3b、c)。因此,BayesPrism利用TCGA确定了可通过肿瘤内异质性复制的相关性。为总结与细胞间相互作用相关的生物过程,作者使用候选相互作用基因和非恶性细胞类型部分之间的相关系数进行了基因集富集分析(图3d),该分析揭示了几种互动模式。首先,在所有三种肿瘤类型中,许多与非恶性细胞类型比例相关的生物学过程都是独立发现的。例如,在所有三种肿瘤类型中,干扰素γ/α反应与巨噬细胞呈正相关(图3e)。间充质激活与GBM中的巨噬细胞、SKCM中的内皮细胞和成纤维细胞呈正相关,与HNSCC中的淋巴细胞呈负相关(图3d,f)。最后,一些生物过程只与一种肿瘤类型相关,但与该肿瘤中的多种细胞类型相关。例如,角质化与HNSCC中的多个非恶性细胞呈负相关,但与肥大细胞呈正相关(图3g)。这些结果突显了BayesPrism在研究恶性和非恶性细胞浸润中生物过程之间的相互作用方面有重要作用。
图3 恶性细胞基因表达与非恶性细胞分数之间的相关性。
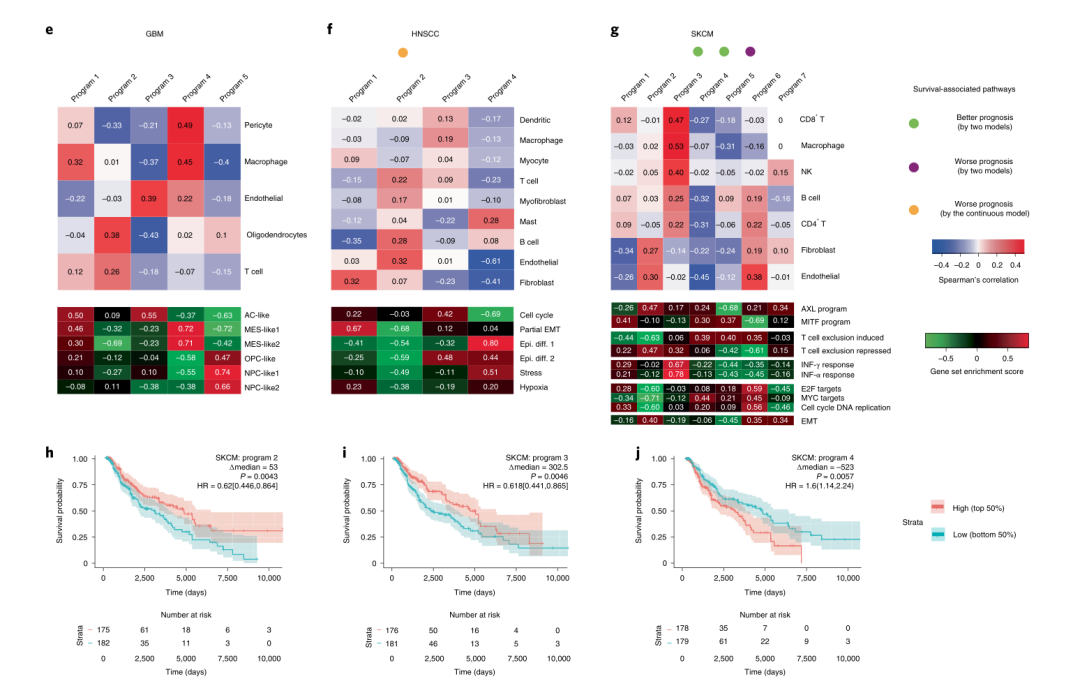
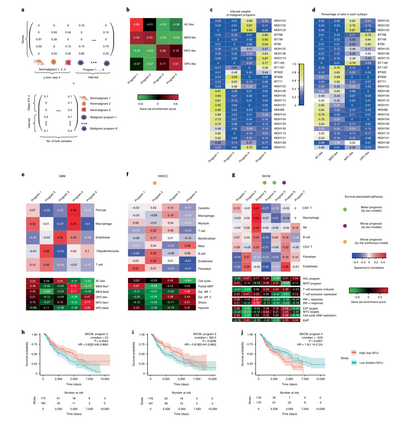
BayesPrism识别恶性细胞内在基因程序 作者在BayesPrism中开发了一个用于推断基因程序的线性组合的模块,在从非恶性细胞类型中分解出基因表达后,解释了bulk RNA-seq中的表达异质性(图4a),作者在由28GBMs读数聚集产生的伪bulk数据集上验证了该方法,BayesPrism恢复的基因程序与最近通过对同一数据集的6863个单一恶性细胞进行因子化得到的基因程序相似(图4b)。研究发现,基于BayesPrism学习的每个基因程序的权重与分配给四个主要亚型肿瘤中的细胞比例相关(图4c,d)。作者将嵌入学习应用于GBM、HNSCC和SKCM,揭示了GBM中与先前研究相似的几个程序,包括程序3(经典和AC类)、程序4(间充质)和程序5(前神经、OPC和NPC类)(图4e)。在HNSCC中,程序1因单细胞研究(图4f)确定的部分EMT程序而丰富,与存活率呈负相关(P=0.017,瓦尔德检验)。在SKCM中,作者确定了多个与AXL和MITF基因程序相关的生存相关基因程序(之前使用TCGA批量数据报告),以及一个T细胞排除程序(在最近的scRNA-seq研究中确定;图4g-j)。与基因集富集分析一致,BayesPrism没有发现任何与HNSCC中的间充质亚型或GBM中的神经亚型相似的基因程序。因此,作者认为,嵌入学习模块减少了非恶性细胞类型的影响,导致了恶性细胞固有的基因程序。
图4 BayesPrism在排除非恶性细胞表达后重新定义了GBM分子亚型。
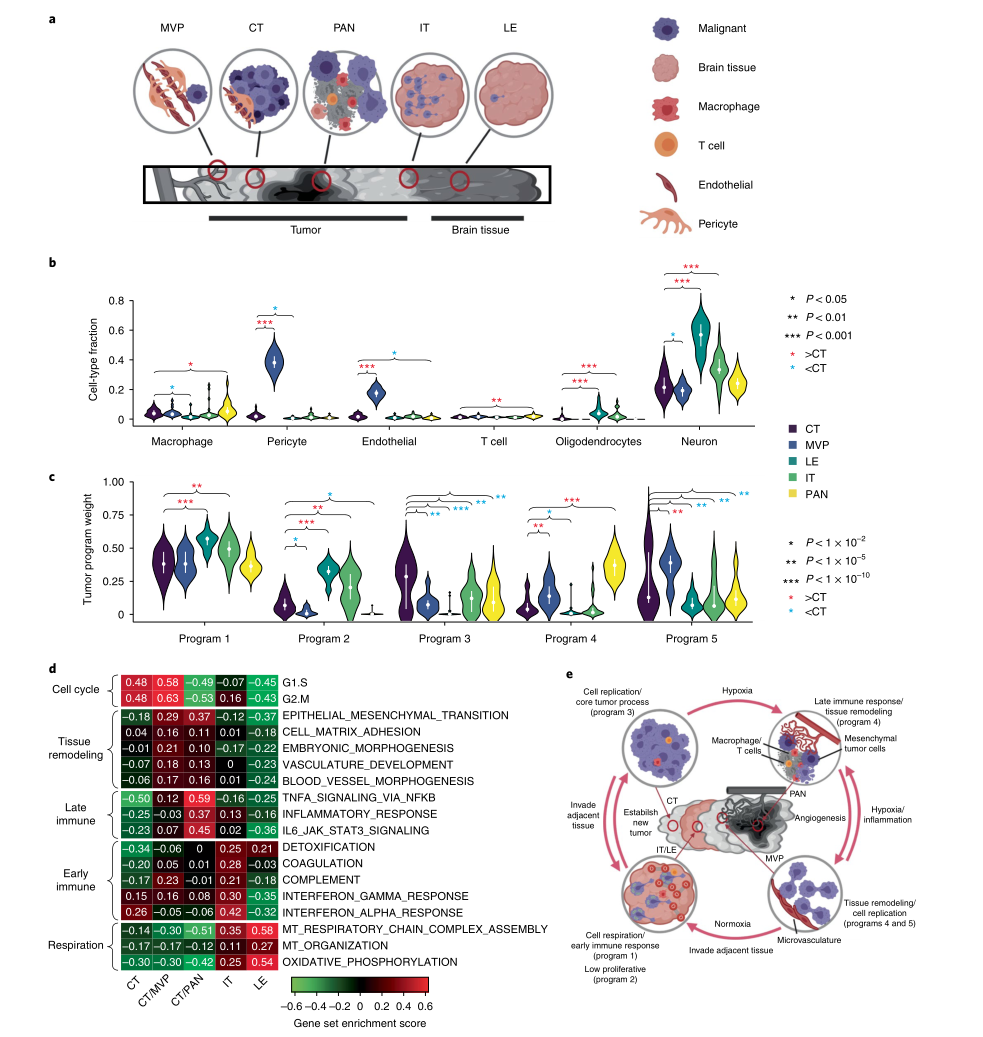
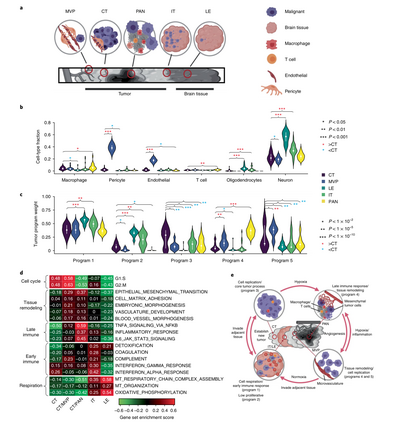
GBM基因程序和细胞类型的空间异质性 本文假设恶性细胞中基因程序的激活与微环境中非恶性细胞类型的比例之间的关系可以显示出肿瘤内的空间异质性。实验使用IVY GAP将122个RNA-seq样本分离成五个结构:前缘(LE)、浸润性肿瘤(IT)、细胞肿瘤(CT)、微血管增生(MVP)和坏死周围的假栅栏细胞(PAN)(图5a)。作者检查了IVY GAP研究的解剖结构中富集了哪些细胞类型和基因程序(上面使用TCGA识别)(图5b,c),发现MVP区域在内皮细胞和周细胞高度富集,而LE和IT区域在少突胶质细胞和神经元高度富集。同时,为有助于解释BayesPrism获得的程序中的富集情况,作者分析了每个IVY缺口结构中恶性细胞(使用BayesPrism推断)的基因集富集分数,这些生物学过程的子集显示了TCGA-GBM的显著变化(图5d)。此外,发现CT和MVP具有高度增殖性,这与它们在程序3和5中的富集程度一致, MVP和PAN在组织重塑和免疫相互作用方面都有所增强(程序4),而MVP更具血管生成性,PAN更具炎症性。综上所述,上述分析显示了BayesPrism如何利用IVY缺口数据集将路径和基因程序与空间解剖结构联系起来。
图5 BayesPrism揭示了GBM的空间异质性。
3 总结与讨论 现在有大量的文献提供了非恶性细胞如何影响恶性细胞功能的例子,证实了一个多世纪以来关于TME关键作用的猜测。scRNA-seq不仅可以系统地测量肿瘤中存在的细胞类型,还可以测量它们的基因表达状态。尽管scRNA-seq提供了正确的数据模式,但目前的研究还没有足够大的样本量来解决这些问题。除此之外,虽然可用的bulk RNA-seq数据集可达上千个,但它们只能提供关于各种恶性肿瘤中整个细胞环境中少量的信息。本文利用了两种基因组资源,开发了一个严格的统计模型来整合scRNA-seq和bulk RNA-seq数据,为肿瘤学这一重大挑战提供了一个新的视角。并且本文的综合分析也为疾病进展提供了新的见解。此外,BayesPrism满足了基因组学工具箱中的几个关键需求。与以前的方法相比,BayesPrism能够更准确地将bulk RNA-seq分解为细胞类型的比例,这部分要归功于对bulk RNA-seq和scRNA-seq数据之间差异进行建模的贝叶斯统计模型。最重要的是,BayesPrism对细胞类型及其样本特异性平均表达联合建模,这对本文报告的分析至关重要。在实际应用中,BayesPrism的精度可能会受到参考矩阵中细胞状态缺失的影响,异质性TME中细胞状态缺失的表达有时会偏离BayesPrism建模的先验分布,导致将细胞状态缺失的转录本部分分配给属于其他细胞类型的细胞。因此在探究基因表达和细胞类型分数的后验估计值之间的相关性时需要谨慎,可能需要使用与本文介绍的类似的过滤器。作者推测,随着从患者身上收集单细胞数据的增加,由于每个数据可能都涵盖了转录状态的细微差别,肿瘤样本的反卷积计算将变得更加准确。因此,BayesPrism将提供一种新的视角,将不断增长的scRNA-seq数据与现有的大量bulk RNA-seq数据进行整合,从而深入了解肿瘤与微环境的相互作用。 参考资料 Chu T , Wang Z , Pe'Er D , et al. Cell type and gene expression deconvolution with BayesPrism enables Bayesian integrative analysis across bulk and single-cell RNA sequencing in oncology. Nature Cancer(2022). https://doi.org/10.1038/s43018-022-00356-3
数据 https://doi.org/10.1038/s43018-022-00356-3
代码 https://github.com/Danko-Lab/BayesPrism.git