作者 | 应俊杰
今天给大家介绍的是剑桥大学错误折叠疾病中心(Cambridge's Centre for Misfolding Diseases)、斯坦福大学生物系等机构发表在science advances上的文章《Fragment-based computational design of antibodies targeting structured epitopes》。作者提出了一种设计抗体CDR环的新方法,该方法可针对实验确定的结构或计算模型生成结构的表位。然后将设计好的CDR移植到抗体支架上。最后通过生物实验的方式测量了抗体抗原结合的Kd值和其他亲和力指标,做了多组生物学对照实验,证明了该方法可以设计出高度耐热的有一定亲和力的单域抗体。
1 背景介绍 抗体是生物医学研究中的关键工具,越来越多地用于诊断和治疗多种人类疾病。抗体结构域相当大,并使用位于抗体表面高变环内的互补决定区 (CDR) 结合其靶标,这些环通常是延伸的且高度灵活的。现有抗体发现方法已取得广泛成功,但仍有许多局限性:如需要进行昂贵又耗时的实验室筛选、筛选出来的抗体结合良好但生物物理性质(稳定性、溶解度和产量)较差。大多数可用的结合蛋白设计方法至少部分依赖于突变空间和构象空间的采样来最小化计算出的相互作用自由能。这些基于分子建模和经典力场的计算的性质,以及实现采样的挑战,使得模拟相当不精确且资源密集。因此抗体的从头设计通常成功率较低。
2 CDR设计策略
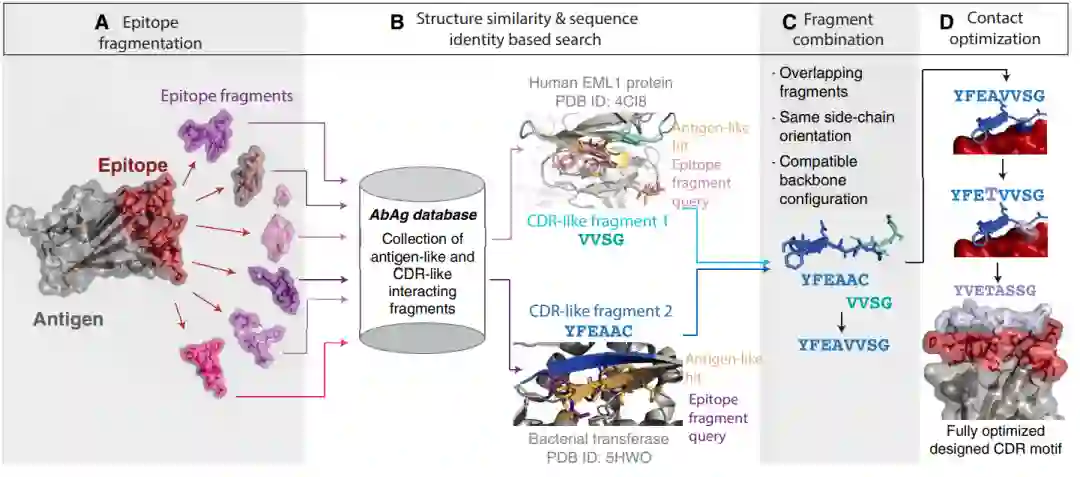
图1:基于结构组合的CDR设计的工作流程
2.1 构建AbAg数据库 为了克服上面提到的分子建模的一些局限性,特别是那些与原子间相互作用的近似值相关的局限性,作者利用大型结构数据库存储抗原抗体样片段来设计与表位互补的CDR。具体来说,作者从非冗余蛋白质数据库(PDB)中获取CDR样片段和相应的抗原样区域的数据,并将它们构成一个数据库,称之为 AbAg 数据库(图1B)。CDR样片段被定义为在结构上与抗体CDR环兼容的线性基序,它可以在 PDB 的任何蛋白质结构中找到。抗原样区域包含与CDR样片段相互作用的残基。
2.2 目标抗原表位结构碎片化 给定目标表位的结构,可以搜索数据库以识别与该表位或其片段类似的抗原样区域。通过这种方式,与搜索出的抗原样区域相互作用的CDR样片段也可能与目标表位相互作用。为了执行此搜索,使用两种不同的策略对输入表位的结构进行碎片化(图1A)。
(1)线性碎片化,其产生至少四个连续残基的片段 (2)表面补丁碎片化,该碎片化取每个残基并产生三维结构距离上最接近的n个溶剂暴露的位点(n≥4) 这两种方法允许覆盖更宽的搜索空间,因为第一种方法对连续表位进行彻底搜索,而第二种方法更适用于包含多个片段的信息表位,这些片段通常在序列空间中距离较远。
2.3 搜索与目标抗原表位碎片相近的抗原样区域 接下来,将每个目标表位片段与抗原样区域进行比较,以识别具有可匹配骨架结构和类似序列的区域。使用Master算法进行搜索,并且比较基于全主干的均方根偏差(RMSD)和序列相似性。因此,命中抗原样区域在序列和结构上都与其查询表位片段相似。由于AbAg数据库的性质,该程序产生了与鉴定的抗原样片段相互作用的CDR样片段(图1 B)
2.4 CDR样片合并 通过将每个抗原样区域及其相互作用的CDR样片段叠加到表位的匹配部分,旋转这些CDR样结构以匹配表位的方向(图1C)。在可能的情况下,主链部分重叠并与单个较长CDR环兼容的不同CDR样片段连接在一起,以产生更长的相互作用基序。
2.5 接触优化 当该片段转移到表位上时,每个CDR样片段的一些原始相互作用可能会受到影响,例如,如果抗原样区域的序列与相应的表位序列不一致,或者如果表位侧链以不同的构象存在(图1D)。类似地,当CDR样片段与与其抗原样区域不匹配的部分表位形成接触时,可能会产生新的相互作用。为了克服这些次优相互作用产生的潜在问题,作者实施了一个侧链优化程序,旨在最大化CDR样片段和抗原之间的有利相互作用的数量。
3 CDR嫁接
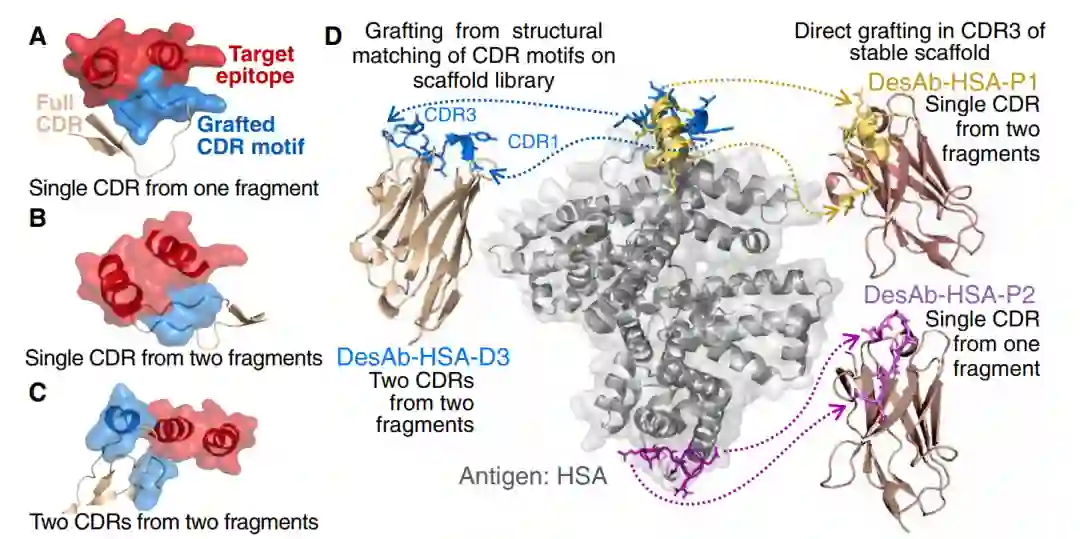
图2:CDR基序移植抗体支架、抗体抗原对接
设计好的CDR基序可以移植到抗体支架中(图2)。在结构上匹配生成的基序,使其与完整的CDR或整个抗体结构(特别是Fv区)相匹配,这可能导致含有多个基序的较长CDR环,或将多个基元嫁接到同一Fv区的不同CDR环中(图2,A至C)如果需要,使用上述侧链优化程序优化移植抗体支架和抗原之间的任何新的相互作用。此外,作为这种结构匹配的替代方案,设计的CDR基序也可以直接移植到已知对环置换高度耐受的抗体支架中。在这项工作中,我们对两种方法进行了实验测试(图2D)
4 设计结果和生物物理特性描述 作者探索两种嫁接策略,为三种不同的抗原设计了六种单域抗体:将设计的CDR直接嫁接到稳定的支架上,以及将设计的CDRs与结构上与其相容的支架匹配。第一种策略提供了通过最小化移植引起的可能并发症来测试从头CDR设计程序的机会,而第二种策略是更复杂的方法,允许在结构上与表位匹配的支架上设计多个CDR环。两种设计的单域抗体(DesAbs)针对严重急性呼吸综合征冠状病毒2(SARS-CoV-2)刺突蛋白受体结合域(RBD)、三种人血清白蛋白(HSA)和一种胰牛胰蛋白酶
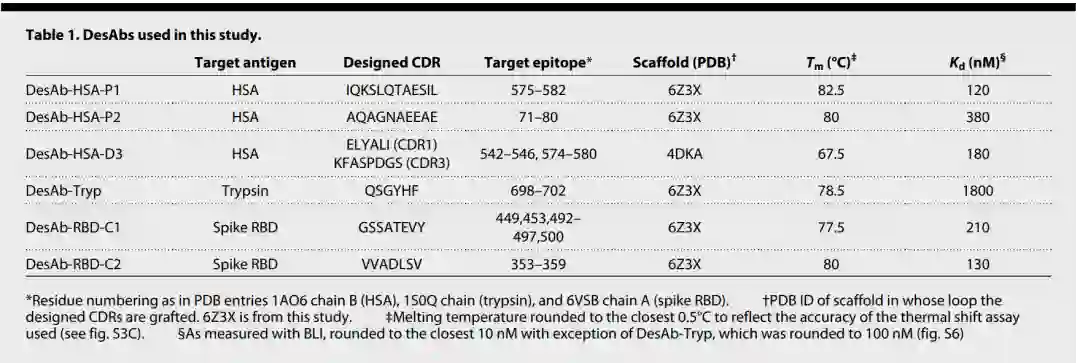
表1:研究中使用的DesAbs
三种抗HSA单结构域抗体中的两种,DesAb HAS-P1和DesAb HAS-P2(表1和图2D),由设计的CDR基序和稳定的支架组成。第三种设计DesAb HAS-D3,是通过将两个单独的CDR样候选物在结构上匹配到被鉴定为与这两个结合基序高度兼容的纳米体支架的两个CDR环上来实现的(图2D)。其中P2针对的表位与P1、C1不同。DesAb-Trip与DesAb-HAS-P1和DesAb-HAS-P2除了CDR3环序列中嫁接的设计CDR基序不通,其他都一样,因此特别合适做阴性对照,以确认观察到的结合来自嫁接的设计基序。
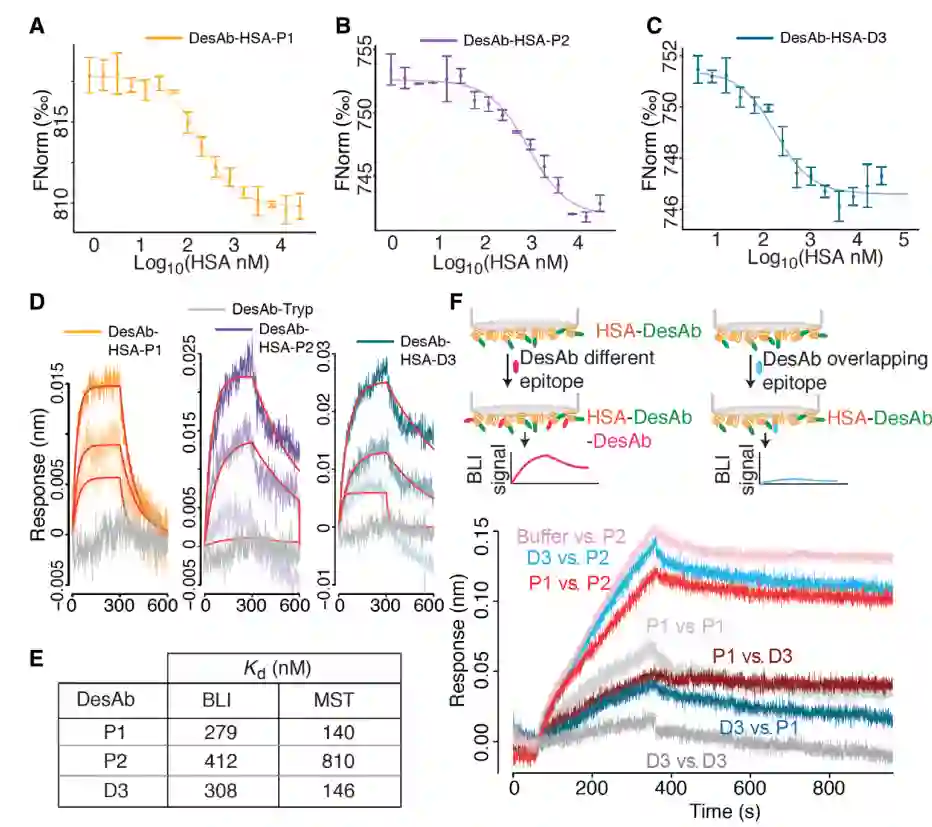
图3:HSA DesAbs结合亲和力实验
在微尺度电泳(MST)的溶液中测量与HSA的结合,其产生的Kd值范围为140至800nM(图3,A至C和E)为了确认结合,我们还使用固定化HSA进行了生物层干涉法(BLI),获得了与溶液中测量的Kd值一致的Kd(图3,D和E)。作为阴性对照使用的胰蛋白酶靶向DesAb Tryp在该测定中未给出与HSA的结合信号(图3D)。在胰蛋白酶实验中DesAb-Tryp能够结合其预期的靶胰蛋白酶,而DesAb-HAS-P1和DesAb-HAS-P2没有显示结合信号,并且在结合测定期间可能被蛋白酶消化。
作者通过BLI竞争实验分析得出,DesAb-HAS-P1和DesAb-HAS-D3相互竞争与HSA的结合,因为其中一种的结合受到另一种抗原结合的DesAb的阻碍。相反,DesAb-HAS-P2不与二者竞争,因为其结合不受其他抗原结合DesAb存在或不存在的影响(图3F)。
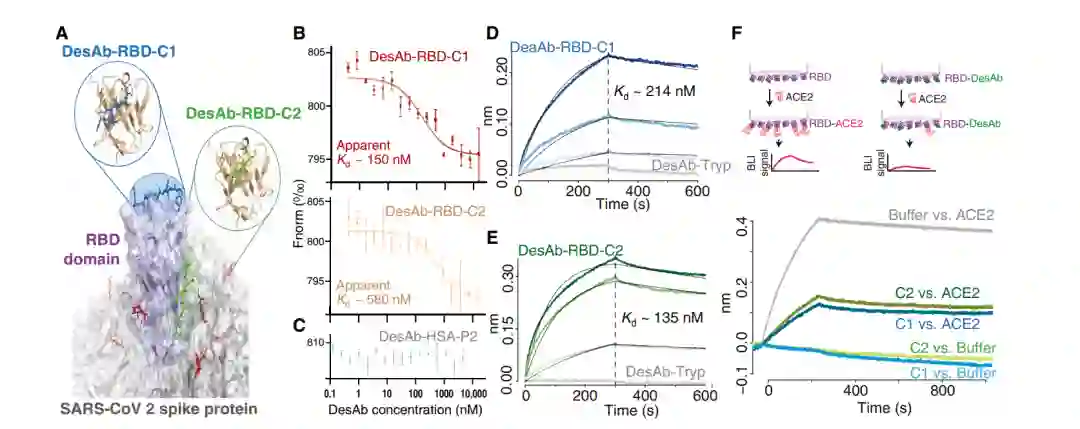
图4:RDB DesAbs结合亲和力实验
作者首先使用MST测试了溶液中与全三聚体刺突蛋白(RDB)的结合(图4B)。两种RBD靶向DesAb均显示与刺突蛋白结合,而用作阴性对照的HSA靶向DesAb-HAS-P2在测定中没有信号(图4C)。为了确认结合,作者用固定化的天然糖基化RBD进行了BLI测定,其DesAb-RBD-C1和DesAb-RBD-C2的Kd值分别为210和130 nM(图4 D和E)。
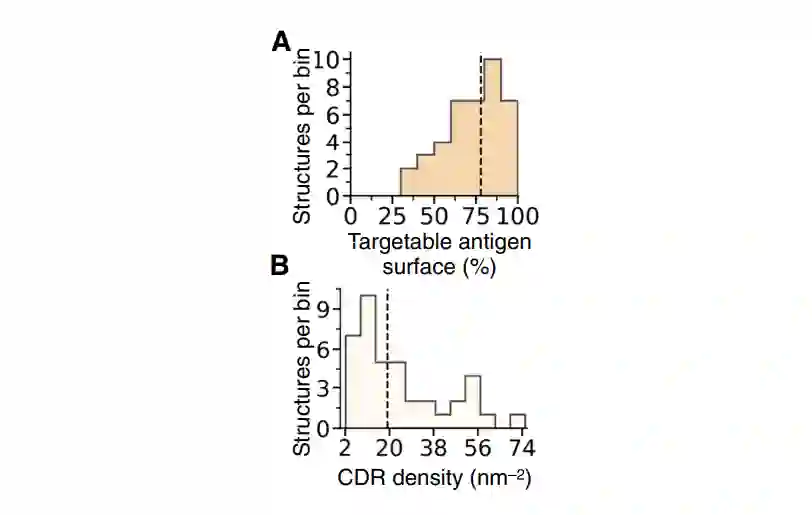
5 设计策略的适用性 确认了计算方法可以产生稳定且具有一定亲和力的单域抗体,作者研究了设计策略的容易程度和普遍适用性。测试了设计合适的CDR样片段来靶向目标表位的可能性,即表位在AbAg数据库中具有合适匹配片段的可能性。为了解决这个问题,作者在蛋白质结构预测竞赛(CASP14)的所有实验目标结构的整个表面上运行了上述的CDR设计流程。
图5:CDR设计程序的适用性
我们的结果表明,我们的策略通常可以针对每种抗原的大部分表面,平均表面覆盖率为78%(图5A)。此外,对于每个表位,通常有许多候选设计的CDR环可供选择,其中值密度为每平方nm抗原表面19个设计的CDR(图5B)。总之,这些结果表明,虽然存在一些不能用组合策略靶向的表位,但大多数表位可以通过在多个不同的设计CDR候选物之间进行选择来靶向
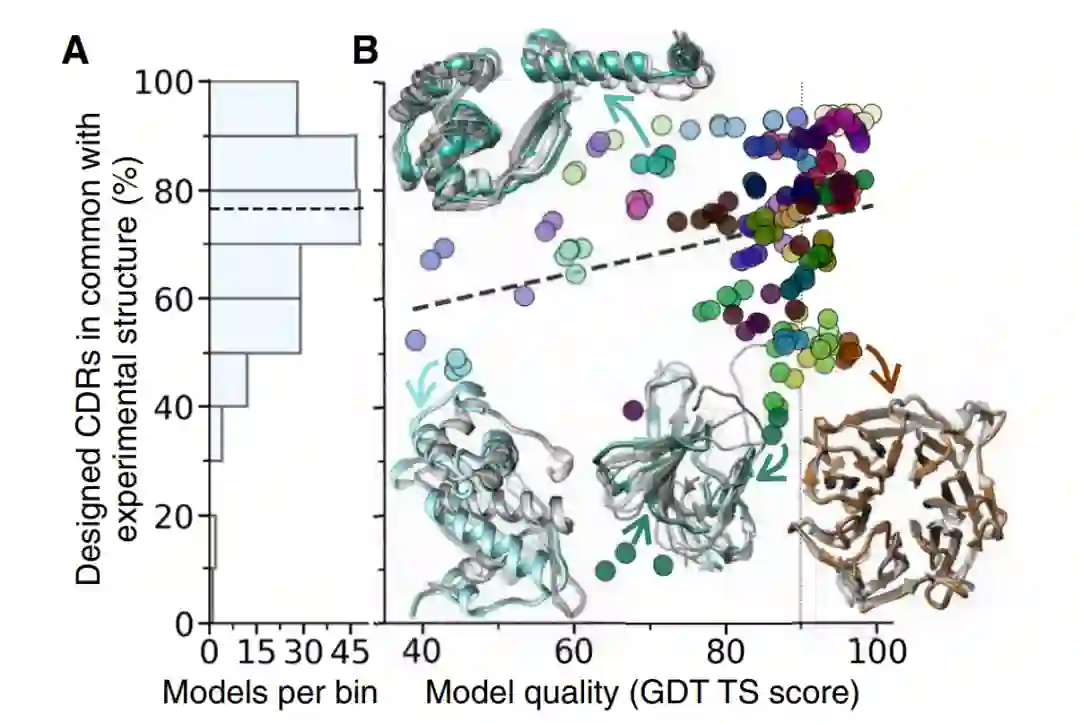
在确定了设计策略可以针对大多数表位后,设计流程最明显的瓶颈是需要一种结构作为输入。因此,作者接下来研究了该方法在不同模型产生的蛋白质结构上的适用性。作者对使用AlphaFold2生成的所有CASP14模型运行了CDR设计程序。通过对每个目标结构使用CASP14中存储的所有模型,还将CASP中排名不靠前的低质量模型纳入了分析中。
图6:CDR设计流程对模型的依赖性
结果表明,通过使用每个模型作为输入获得的大多数设计的CDR样片段与使用相应的实验确定的结构获得的片段有效相同(图6A)。每个模型及其相应实验结构之间的设计CDR的平均数量(按每个模型获得的设计CDR总数的百分比计算)为77%,分析的200个模型中只有20个(10%)的CDR与其目标结构的共有性低于50%。这些结果表明,如果使用AlphaFold2模型作为抗体设计管道的输入,通常约75%的生成CDR与从相应晶体结构获得的CDR相同,90%的情况下至少50%相同。总之,这些结果表明,当在计算机预测模型或实验结构上运行时,CDR设计程序可能会产生类似的结果,并且这些结果并不强烈依赖于用作输入的模型的质量,至少在我们探索的质量范围内(GDT>40)
6 总结 作者描述了一种基于片段的策略,用于针对结构化表位的抗体的设计方法。使用至少四个残基且通常更长的蛋白质片段以组合方式组装设计的CDR。实验结果表明,作者提出的设计方法可以产生高度耐热的单结构域抗体,并在两种独立的生物实验技术(MST和BLI)中得到证实,该方法设计出的抗体与抗原结合Kd值可在纳米摩尔范围内。
作者探索了两种嫁接策略(图2)。其中一种是将设计的基序嫁接到稳定支架的CDR3中,另一种是在结构上匹配到相容框架的两个不同环上(DesAb-HAS-D3)。作者经过实验发现通过不同策略获得的DesAbs的结合亲和力不存在显著差异。通过各种阴性对照实验,作者进一步验证了DesAbs不结合不匹配的抗原。
参考资料 Aguilar Rangel, Mauricio, et al. "Fragment-based computational design of antibodies targeting structured epitopes." Science Advances 8.45 (2022): eabp9540. DOI: 10.1126/sciadv.abp9540