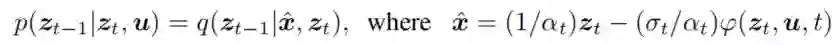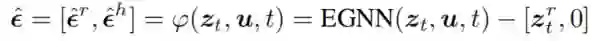编译 | 陈睿哲
审稿 | 刘名权 今天给大家介绍的是来洛桑联邦理工学院、MIT等团队2022年10月发表在Arxiv上的预印本《Equivariant 3D-Conditional Diffusion Models for Molecular Linker Design》。
1 摘要 基于片段(fragment)的药物发现已经成为早期药物开发的一个有效范式。这一领域的一个开放挑战是设计在interest的断开分子片段之间的连接(linker),以获得化学相关的候选药物分子。在这项工作中,我们提出了一个扩散模型,一个E(3)等变3D条件扩散模型的分子连接设计模型。给定一组不相连的片段,我们的模型将缺失的原子放在中间,并设计了一个包含所有初始片段的分子。与以前只能连接分子片段对的方法不同,我们的方法可以连接任意数量的片段。此外,该模型还会自动确定连接器中的原子数及其连接点指向输入片段。我们证明了扩散连接模型在生成更多样化和综合可获得的分子的标准数据集上优于其他方法。此外,我们在现实应用中实验测试了我们的方法,表明它可以成功地生成基于目标蛋白口袋条件的有效连接。
2 简介 据估计,药物的分子空间将超过10的60次方个结构,在该空间中搜索对药物设计提出了重大挑战。缩小这个空间大小的一个方法是从片段开始,片段是一种较小的分子化合物,通常有不超过20个重(非氢)原子。这种策略被称为基于片段的药物设计。给定一个蛋白质口袋(目标蛋白的一部分,利用合适的特性结合配体),计算确定与口袋相互作用的片段是一种比实验性高通量筛选方法更便宜、更有效的替代方法。一旦相关片段被识别出来并与目标蛋白对接,就需要将它们结合成一个单一的、连接的分子。在片段连接、合并和生长等各种策略中,前者是首选,因为它可以快速提高目标和化合物的结合能。这项工作解决了连接的问题。
早期的分子连接设计的计算方法是基于数据库搜索和物理模拟,这两种方法都是计算密集型的。现有的方法要么基于语法模式识别,要么基于自回归模型。前一种方法只使用SMILES,而后者则考虑了输入片段的三维位置和方向。然而,这些方法对于原子的排列并不是等变的,只能结合成对的片段。
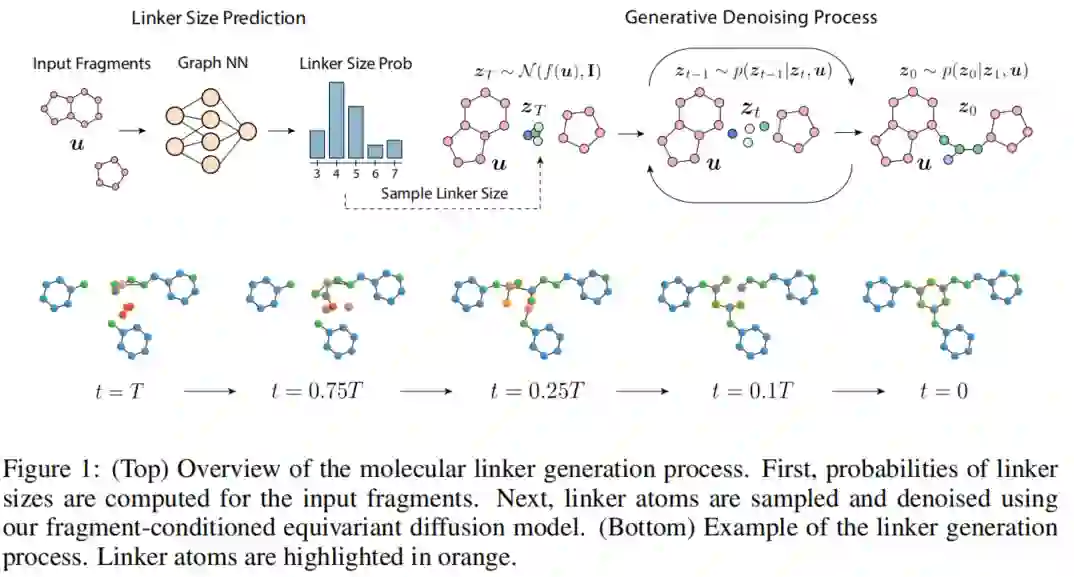
连接的设计依赖于目标蛋白口袋,正确地使用这些信息可以提高整体化合物的亲和力。然而,到目前为止,还没有计算方法的分子连接器设计,考虑到口袋。在这项工作中,我们提出了DiffLinker,一个条件扩散模型,它为一组表示为三维原子点云的输入片段生成分子连接器。首先,我们的模型生成预期连接的大小,然后从正态分布中采样初始连接原子的类型和位置。接下来,使用以输入片段为条件的神经网络迭代地更新连接原子类型和坐标。最终,去噪的连接原子和输入片段原子形成一个连接分子,如图1所示。
DiffLinker具有几个理想的特性:它关于平移、旋转、反射和排列等变,它不受输入片段数量的限制,它不需要关于附着原子的信息,并生成任意大小的连接器。此外,DiffLinker的调节机制允许传递关于周围蛋白质口袋原子的额外信息,这使该模型适用于基于结构的药物设计应用。
我们的经验表明,在生成片段对之间的化学相关连接方面比以前的方法更有效。我们的方法在合成可及性和药物相似性方面取得了最先进的结果,这使其更适合用于药物设计管道。此外,扩散连接器在生成的连接的多样性方面显著优于其他方法。我们进一步提出了一个更具挑战性的基准测试,并表明我们的方法能够成功地连接两个以上的片段,而这是其他方法无法做到的。我们还证明了扩散蛋白可以基于目标蛋白质口袋:我们的模型尊重周围蛋白质原子施加的几何约束,并生成与相应口袋有最小冲突的分子。据我们所知,DiffLinker是第一个不受输入片段数量限制并描述口袋信息的方法。这项工作的总体目标是为从业者提供一个有效的工具,分子连接子生成在现实的药物设计场景。
3 方法 在本节中,我们将介绍DiffLinker,一种新的E(3)等变扩散模型,用于生成以3D片段为条件的分子连接。我们制定了底层去噪分布的等方差要求,并再提出了一个可学习的动态函数。我们讨论了取样一个连接的大小和调节蛋白质口袋的策略。完整的连接器生成工作流程如图1所示。
3.1 EQUIVARIANT 3D-CONDITIONAL DIFFUSION MODEL 与其他分子生成的扩散模型不同,我们的方法以三维数据为条件。更具体地说,我们假设每个点云x都有一个相应的上下文u,这是另一个由所有输入片段和蛋白质口袋原子组成的点云,这些原子在整个扩散和去噪过程中保持不变,如图1所示。因此,我们考虑公式扩散过程中的生成过程在点云x上操作,同时基于固定的相应上下文。
条件去噪模型的马尔科夫链定义:
函数f的选择在很大程度上取决于要解决的问题和可用的先验。在我们的实验中,我们考虑了两个情况。首先,我们利用了应该由连接器连接的原子的信息。我们称这些原子为锚点,并将f (u)定义为锚点的质心。然而,在现实世界中,不太可能知道哪些原子应该是锚点。在这种情况下,我们将f (u)定义为整个环境u的质心。
我们注意到概率模型p与平移并不是等变的。为了克服这一问题,我们构造了附加平移不变的网络ϕ。然后,我们不再从N (f (u))采样初始噪声,而是将数据集中在f (u),从标准正态分布中采样。
3.2 EQUIVARIANT GRAPH NEURAL NETWORK 建模扩散模型动力学的可学习函数ϕ被实现为一个改进的E(3)等变图神经网络(EGNN)。它的输入是时间t和上下文u的连接z_t的噪声版本。这两部分被建模为一个单一的全连通图,其中节点用坐标r和特征向量h表示。
为了使函数ϕ对平移不变,我们从预测噪声的坐标分量中减去初始坐标:
EGNN由等变图卷积层组成,消息传递的过程如下:
节点坐标的后一种更新由可学习函数φ_vel计算。我们的图中包含了一个有噪声的连接器z_t和一个固定的上下文u,而ϕ的目的是预测应该从z_t的坐标和特征中减去的噪声。因此,在计算动态时,保持上下文坐标不变是很自然的,并且在每个EGCL步骤中只对连接部分应用非零位移。因此,我们对节点位移的建模如下:
通过构造实现了卷积层的等方差。消息φ_e和节点更新φ_h只依赖于标量节点特征和E(3)不变的节点之间的距离。坐标更新φ_vel另外线性地依赖于坐标向量之间的差值,这使得它们成为E(3)等变的。
由于我们只对与连接相关的部分感兴趣,因此我们放弃了上下文节点的坐标和特征,并将元组[z_r,z_h]视为EGNN输出。
3.3 LINKER SIZE PREDICTION 为了预测一组片段之间缺失的连接的大小,我们将片段表示为一个全连接图,使用原子类型的独热编码作为节点特征,节点之间的距离为边特征。由此,一个单独训练的GNN产生了连接大小的概率。我们的假设是,相对的片段的位置和方向以及原子类型包含了所有预测最可能的潜在连接大小的必要信息。当生成一个连接时,我们首先用在训练数据中看到的连接大小列表上的分类分布上的预测概率对其大小进行采样,如图1所示。
3.4 PROTEIN POCKET CONDITIONING 在现实世界中基于片段的药物设计应用中,经常会发生片段被选择并连接到目标蛋白口袋中。要提出一种候选药物分子,这些片段必须被连接起来。当生成连接时,应该考虑到周围的口袋,并构建一个与蛋白质口袋原子没有冲突的连接(换句话说,连接和口袋原子的配置应该是现实的),并保持较高的结合强度。为了将口袋条件反射添加到DiffLinker中,我们将一个蛋白质口袋表示为一个原子点云,并将其视为上下文u的一部分。我们还扩展了节点特征,用一个额外的二进制标记来标记属于蛋白质口袋的原子。最后,由于新的上下文点云包含了更多的原子,我们修改了数据点z_t和传递给神经网络ϕ的上下文u的联合表示。我们不考虑全连通图,而是基于4A˚的距离截止来分配节点之间的边,因为它使得到的图更不密集,并抵消了节点数量的增加带来的计算负载。
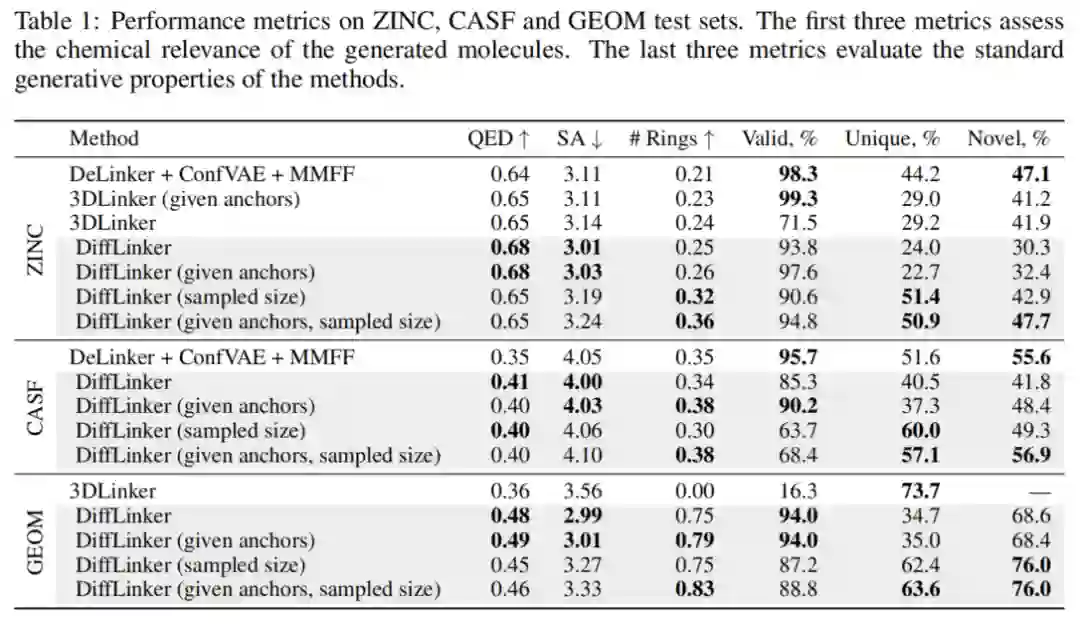
4 实验结果 在ZINC、CASF和GEOM测试集上的性能指标。前三个指标是为了评估所生成的分子的化学相关性。最后三个指标评估了这些方法的标准生成特性。
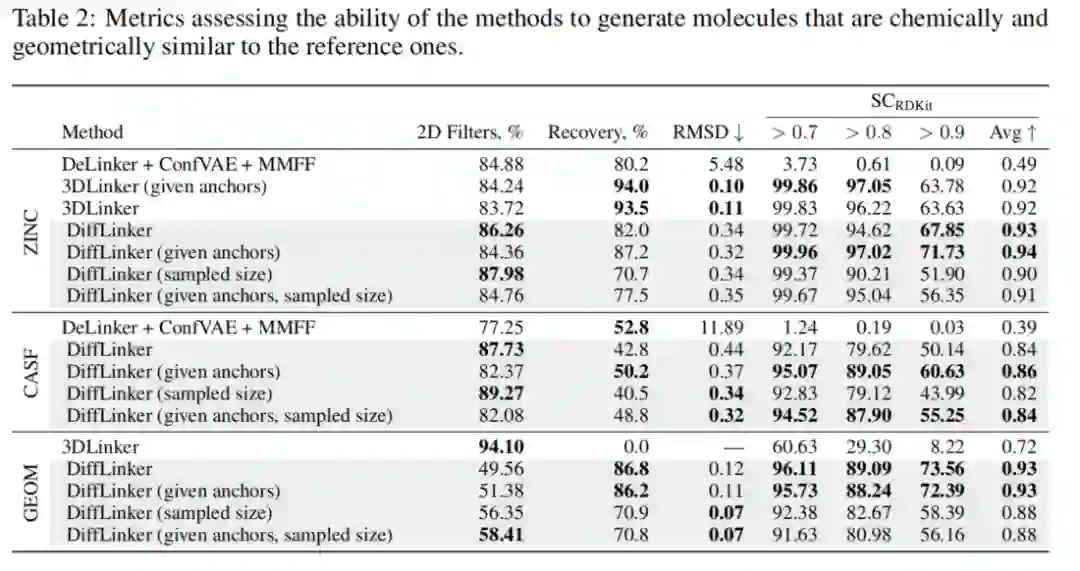
ZINC和CASF:虽然我们的模型在更多的应用中具有更大的灵活性和适用性,但在生成的分子的化学相关性方面,它们在标准基准测试的ZINC和CASF方面也优于其他方法。如表1所示,通过DiffLinker取样的分子更容易合成获得,并表现出更高的药物相似性,这对于药物设计应用尤为重要。此外,我们的模型生成了包含更多环的连接。此外,我们的分子通常具有更高的化学和几何相似性,如表2中的SCRDKit评分所示。在有效性方面,我们的模型的性能与其他方法相同。两种自回归方法在每个生成步骤中都明确地使用了价规则,而我们的模型显示出能够从数据中学习这些规则。值得注意的是,对连接的大小进行采样显著提高了生成的连接的新颖性和唯一性,而没有显著退化最重要的指标。
与最近提出的自回归模型DeLinker和3DLinker相比,它的主要优势是在任意数量的片段之间一次性生成整个连接。这克服了DeLinker和3DLinker的局限性,它们一次只能连接两个片段。虽然这些自回归模型可以进行调整,以便在生长分子时迭代地连接片段对,但在这种情况下,不能考虑到完整的背景。因此,更有可能产生次优解。为了说明这一差异,我们采用了3DLinker来迭代地连接分子中需要连接两个以上片段的片段对。如表1所示,3DLinker在几乎84%的情况下无法构建有效分子,无法恢复任何参考分子,而尽管该数据集中连接器的复杂性更高,但我们的模型达到了94%的有效性,恢复了50%以上的参考分子。此外,由3DLinker生成的分子在连接体中没有环,QED大大降低,而且更难合成。
为了说明DiffLinker考虑周围口袋的能力,我们在口袋数据集上训练了三个模型:它们分别基于全原子口袋表示,基于口袋主干原子和非条件。我们还计算了生成的分子和周围口袋之间的冲突次数。如果两个原子之间的距离小于它们的范德华半径之和,我们就说两个原子之间会发生冲突。如图2所示,以全原子口袋表示为条件的模型产生的分子具有几乎相同数量的冲突(平均每个分子7次冲突)(平均每个分子6次冲突)。根据口袋信息的数量有一个明显的趋势:口袋主干原子的模型平均产生14次冲突的分子,而非条件模型平均产生21次冲突的分子。
5 总结 在这项工作中,我们提出了DiffLinker,一个新的E(3)等变3D条件扩散模型的分子连接设计。DiffLinker通过生成一个连接,从一组断开连接的片段中设计真实的分子,即一个相互连接输入片段的原子点云。虽然以前的方法只能连接成对的片段,但扩散连接模型可以扩展到任意数量的片段。我们的方法不需要指定片段的附着点,并从片段中预测连接大小的分布。我们表明,所提出的方法在标准基准上优于其他模型,并产生更多的化学相关分子。此外,我们还证明了我们的模型可以以蛋白质口袋为条件,并产生具有最小冲突次数的连接。我们相信,我们的方法将加速潜在候选药物的开发,并有潜力在基于片段的药物设计领域得到广泛应用。 参考资料 https://doi.org/10.48550/arXiv.2210.05274