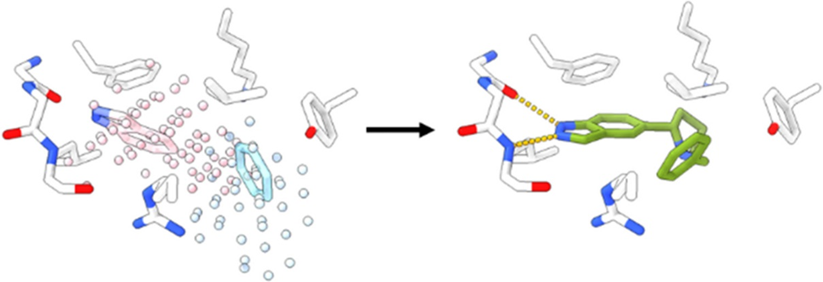
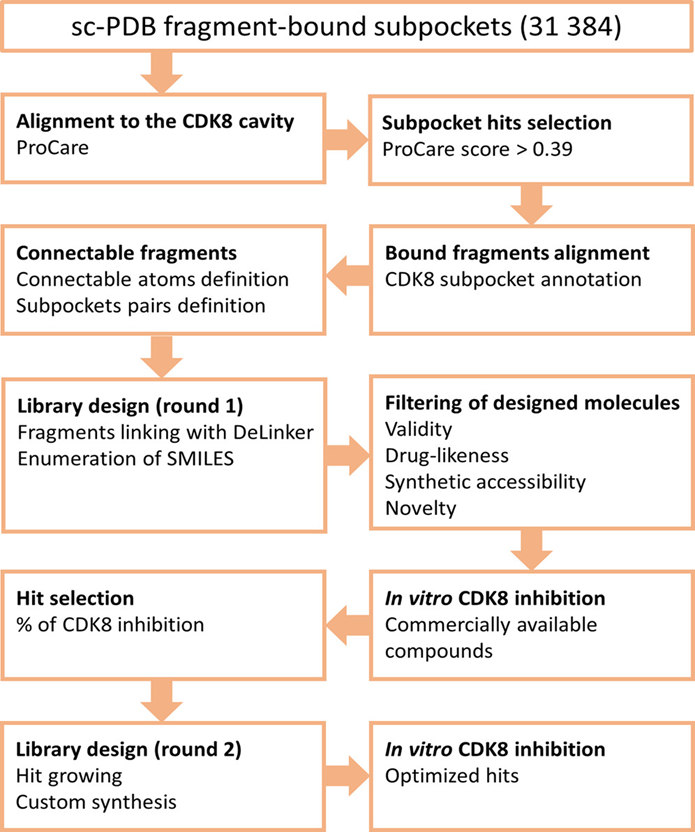
基于片段的药物设计(fragment-based drug design, FBDD)一直是药物研发的核心问题之一。通过对低分子量化合物库(分子片段库)进行高通量筛选并设计连接片段进行组装,研究人员可以在更大的化学空间内开展探索。另外,筛选出的片段往往可以通过组合获得更强的靶点亲合性,往往能在较难设计靶向药物的蛋白上取得奇效。 为了合理地连接片段,了解片段在口袋中的结合位置是必要的。通过实验手段解析晶体或突变关键残基验证结合位置难以高通量实现,因此计算方法是唯一的选择。目前,计算预测方法主要有分子对接、官能团映射和分子结构的拆分与重构。这些方法或多或少受到计算成本和人工判断的限制,不能充分利用蛋白-配体复合物的结构数据。为了解决这个问题,斯特拉斯堡大学的Didier Rognan教授小组提出了一种基于片段所处口袋环境的识别匹配的分子设计方法POEM(Pocket Oriented Elaboration of Molecules),可以实现短时间内大型化合物库设计。作者选择CDK8作为定向设计化合物库的目标,并开展了案例研究。该工作发表在美国化学会出版的药物化学核心期刊Journal of Medicinal Chemistry上(J. Med. Chem. 2022, 65, 20, 13771–13783)。【1】 研究者认为,分子片段在蛋白口袋中的空间定位是独特的,片段的周围空间可以描述蛋白口袋的局部特征,称为“子口袋”。同时,子口袋的特征又是相似的,结合类似子口袋的分子片段可能参与的类似的有利相互作用。基于上述观点,研究者使用IChem程序【2】将从sc-PDB数据库中获取的类药分子及其结合模式碎片化,并通过VolSite工具【3】以点云的形式计算分子片段周围的子口袋特征,每个点允许标注8种性质(疏水性、芳香性、氢键供体、氢键受体、氢键供受体、正电性、负电性和无性质)。按照上述方法,作者构建出了包含31384条片段-子口袋配对的候选片段库,并按如图-1的流程开展分子设计。
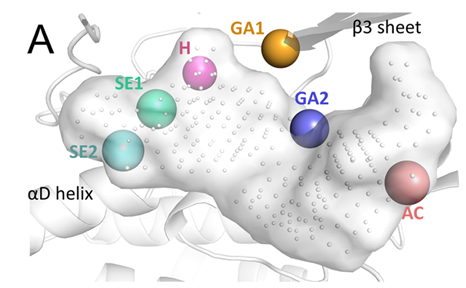
图-1 使用POEM方法设计CDK8小分子抑制剂的工作流程 研究者选择了“DFG-in”构象的CDK8 ATP结合位点作为参考口袋(PDB ID: 5HBH)并采用同样的方法以点云形式表示其特征。如图-2所示,口袋可分为铰链区、溶剂暴露区和门控区。接着,研究者使用内部开发ProCare程序【4】将来自sc-PDB的各子口袋特征分别与CDK8口袋特征进行比较,根据点云的匹配程度打分,点云的在三维空间中的比对考虑了其可旋转和平移性。除子口袋相似性外,研究者在片段库筛选过程中额外考虑了分子片段的类药性和口袋区域定位是否明确等标准,最终筛选出385个分子片段。
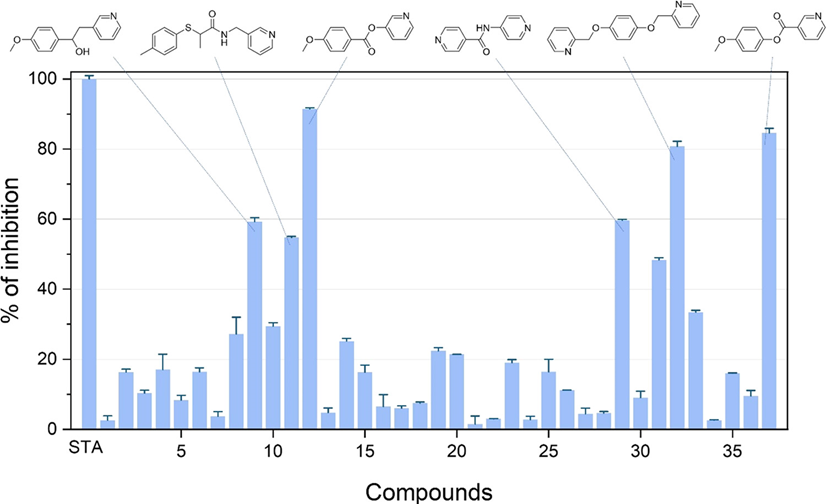
图-2 CDK8 ATP结合位点的空间特征。H表示铰链区,GA1、GA2表示门控区,SE1、SE2表示溶剂暴露区。 接下来,研究者采用深度生成模型DeLinker设计合理的连接结构。DeLinker是一种在ZINC和PDBbind数据集上训练的自动变分编码器模型,其会在连接链生成过程中考虑待连接原子间的距离和角度等限制因素。研究者使用铰链区匹H配的片段作为起点,GA1、GA2、SE2匹配的片段作为连接目标进行设计。经过连接完整性、类药性和可合成性过滤后,本轮设计共获得了114125个不同的分子,其中大部分的分子由铰链区H和溶剂暴露区SE2连接得到。为了进一步探究新分子结构在化学空间中的分布情况,研究者将分子库与ChEMBL类药库和KinFragLib激酶配体库中的分子进行比对,结果表明只有259个与ChEMBL库重复的结构,而与KinFragLib库重复的结构只有5个。计算分子指纹相似性后发现,获得分子库与两个已知数据库相似度大于0.6的比例也仅有13%和0.85%,充分说明POEM生成的结构具有独特性。 为了验证生成结构是否具有活性,研究者挑选出37个候选化合物进行CDK8抑制活性的实验测定。结果如图-3所示,6个化合物在10 μM下取得超过50%的抑制率。其中,化合物12活性最强,IC50达到376.9 nM。但是该化合物仅由铰链区H和门控区GA2连接得到,分子量较小,仍然是一类片段分子。因此,研究者在化合物12结构基础上,按同样的方法开展了第二轮基于片段的分子设计,希望获得能进一步占据SE2和GA1区域的分子结构。第二轮筛选进一步考虑了连接结构是否合适,最终研究者获得了151个新结构并选择其中6个开展合成和活性测试。从中获得的化合物49,由12与SE2定位的片段连接得到,活性达到6.4 nM。
图-3 第一轮设计所得化合物库中37个代表分子的CDK 8抑制活性测试结果【总结】Didier Rognan教授团队提出了一种新的基于片段的化合物设计方法来实现靶点特异性的分子生成。相比于经典方法,该方法不需要花费大量计算成本研究片段库在口袋中的位置,而是从已经积累的大量晶体结构数据中挖掘,构建口袋特征-片段结构的配对数据。虽然该套路不是首次提出,但本文实现了深度学习方法与基于片段的药物设计的首次结合。基于深度学习方法进行口袋环境匹配和连接结构生成保证结构的合理性和新颖性,对药物设计方法的开发者具有启发作用。【参考文献】【1】Eguida, M.; Schmitt-Valencia, C.; Hibert, M.; Villa, P.; Rognan, D., Target-Focused Library Design by Pocket-Applied Computer Vision and Fragment Deep Generative Linking. Journal of Medicinal Chemistry 2022, 65 (20), 13771-13783. (DOI: 10.1021/acs.jmedchem.2c00931)【2】Desaphy, J.; Azdimousa, K.; Kellenberger, E.; Rognan, D., Comparison and Druggability Prediction of Protein–Ligand Binding Sites from Pharmacophore-Annotated Cavity Shapes. Journal of Chemical Information and Modeling 2012, 52 (8), 2287-2299.【3】Da Silva, F.; Desaphy, J.; Rognan, D., IChem: A Versatile Toolkit for Detecting, Comparing, and Predicting Protein–Ligand Interactions. ChemMedChem 2018, 13 (6), 507-510.【4】Eguida, M.; Rognan, D., A Computer Vision Approach to Align and Compare Protein Cavities: Application to Fragment-Based Drug Design. Journal of Medicinal Chemistry 2020, 63 (13), 7127-7142.