今天给大家介绍一篇由剑桥大学、蒙特利尔学习算法研究所、蒙特利尔大学、Deepmind的研究人员发表在The 2021 ICML Workshop on Computational Biology的文章Neural message passing for joint paratope-epitope prediction。该论文研究了表位-补位联合预测任务,确定了两个任务之间固有的不对称性,并提出了表位-补位消息传递 (EPMP)模型,利用不对称性为两个任务设计分别有效的预测器。实验部分在表位预测和补位预测两个任务上都取得了更优的效果,说明模型可以更好地学习表位和补位的特征。
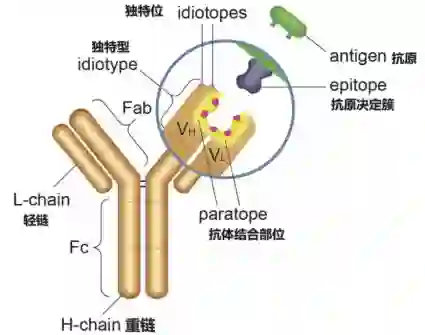
图2-1.抗体-抗原结合示意图其中,抗体抗原上分别对结合起关键作用的补位是:抗原决定部位/表位(epitope):抗原上与抗体特异性结合区域。抗体结合部位/补位(paratope):直接参与识别的抗体区域。如图2-1所示。表位和补位之间的界面对于抗体-抗原相互作用的亲和力和特异性以及抗体的功能至关重要。因此,研究抗体-抗原相互作用可以揭示免疫识别的机制,提供对免疫反应发展的见解。实际问题中,两者具有不同的特点。首先抗体结合部位紧密地定位在Y形抗体的尖端(所谓的Fv区域)上。早期工作考虑了更窄的区域,即互补决定区域(cdr)。其长度很少超过180个残基,允许丢弃大多数抗体序列,从而使预测问题更加平衡。极少数情况下补位可以位于cdr之外,将整个Fv区域作为输入。而对于抗原决定部位,抗原决定部位没有固定区域,一般需要考虑整个抗原。表位预测具有高度的类别失衡:在输入阶段无法轻易丢弃抗原的任何部分,大多数抗原残基不属于表位。表位也对靶向它们的抗体具有特异性,这意味着需要在表位预测因子内小心地利用抗体信息。抗体结合部位是高度顺序的,而且可以单独隔离进行预测。抗原决定部位本质上是结构性的,并且是以抗体结合部位作为固有条件。理想情况下,联合表位-补位预测模型应具有针对这两个任务的单独调整的架构。然而,目前用于联合表位-表位预测的最新技术,PECAN (Pittala & Bailey-Kellogg,2020),两者都使用完全对称的架构。 3.****模型方法本文研究了表位-补位联合预测任务,确定了两个任务之间固有的不对称性,并提出了表位-补位消息传递(EPMP)模型,利用不对称性为两个任务设计分别有效的预测器。 3.1数据集与特征表示
表3-1任务数据集
表位-补位联合预测任务的数据集信息如表3-1所示。特征表示中每个蛋白质结构都表示为图,其中节点表示氨基酸,边当两个氨基酸的主干碳原子之前的距离小于10埃则有边相连。节点特征包括:氨基酸类型、亲疏水性、保守性分数、局部氨基酸信息 3.2抗体结合部位预测模型
图3-1抗体结合部位预测模型抗体结合部位预测模型将整个FV区域作为输入,输入特征最初是按顺序处理的序列信息,之后合并图结构,进行图上的消息传递。氨基酸特征序列首先通过三个CNNs层,其中使用了扩张卷积,有一个扩张项,它在卷积核中产生间隙,允许整个序列被更少的层覆盖,防止过度拟合。CNN的输出嵌入在基于邻近距离的氨基酸残基图上通过两个消息传递神经网络 (MPNN) 层传递。更新函数使用求和聚合。MPNN对应于最具表现力的空间图卷积类别,使其非常适合可用于paratope任务的数据类型。模型使用了跨越连接使得可以跳过MPNN图处理层。最后通过预测层输出预测结果,如图3-1所示。 3.3抗原决定部位预测模型
图3-2 抗原决定部位预测模型抗原决定部位预测模型是结构性的,使用图卷积网络(GCN)编码器来单独处理抗体和抗原图,结合了抗原的大量上下文。然后使用抗原氨基酸和抗体氨基酸的全连接二部图的图注意网络来结合两种表征的关系,其中抗原氨基酸都注意到抗体氨基酸上,反之亦然。GCNs具有完全非参数的聚合过程,仅学习逐点共享的线性变换。这使它们能够抵抗过拟合,并可扩展到更大的图。模型如图3-2所示。 **4.**验证实验
4.1抗体结合部位预测
表4-1 抗体结合部位预测
****由于数据类别不平衡,精确召回曲线(AUC-PR)下的面积是用于对模型进行基准测试的主要指标。为了更容易比较,实验中还比较了AUC-ROC。Para-MPNN和Para-GCN为模型的消融实验,没有使用序列(CNN)组件,仅使用MPNN或GCN作为结构组件。结果如表4-1所示,本文提出的模型在两个评价指标上效果均好于先前的模型。另外,消融实验证明Para-EPMP架构效果优于仅使用MPNN和GCN的模型。 4.2抗原决定部位预测
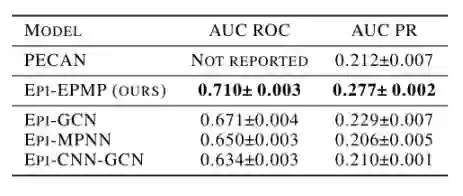
** 表4-2 抗原决定部位预测**
****在抗原决定部位预测任务中,模型比较效果如表4-2所示。其中,EPI-GCN和EPI-MPNN使用GCN或MPNN编码器仅根据抗原(无抗体)中预测表位。EPI-CNN-GCN使用了抗体结合部位预测模型的架构。最终本文的EPI-EPMP模型效果优于先前的模型以及消融实验构造的模型。 4.3定性研究实验
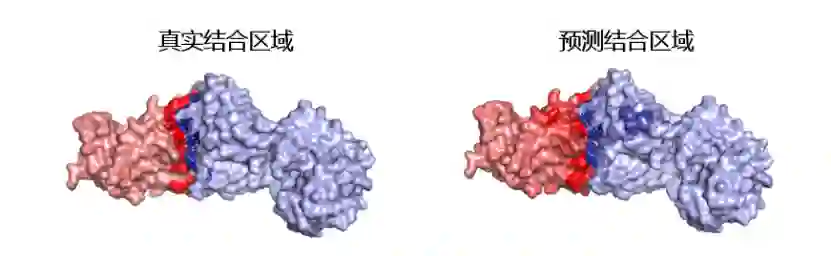
图4-1 定性研究实验图实验评估了模型预测COVID-19中和抗体(B38)与SARS-CoV-2的S1刺突蛋白RBD(受体结合域)之间结合界面的能力。图4-1中可以观察到该模型能够识别表位的正确局部区域,而且预测的假阳性位点都接近结合界面。 5**.****总结展望**本文认为抗体-抗原结合位点预测任务具有不对称性,并基于此分别针对两个任务的特点提出了独立的模型,用于分别预测抗体和抗原上的结合界面。结果表明,本文模型实现了实现表位和补位预测任务的最新性能。PARA-EPMP旨在利用paratope的序列顺序性(CNN),同时还利用了图神经网络(GNN) 的结构线索。EPI-EMPM是结构性的,并广泛地利用抗体的上下文信息,以确保抗原结合位点模型预测得到充分规范。未来的工作可以考虑基于迁移学习学习蛋白质-蛋白质结合位点信息,作为模型的一个良好的初始状态。