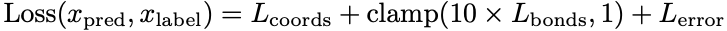Johns Hopkins | 可与AlphaFold2准确率媲美的更快的抗体结构预测工具IgFold
近日JOHNS HOPKINS大学发表了可以快速预测抗体结构的IgFold深度学习方法,其准确率可以与AlphaFold2媲美。
抗体的结合是由一组6个loops的结构决定,这个6个loops构成互补决定族(CDR)。这些CDR loops的精准建模可以深入了解这些结合机制和提高使特定抗体的合理设计成为可能。
5个CDR loops倾向于采用通过序列相似性可以有效地进行预测,然而,重链的第3个CDR loop(CDR H3),由于其在序列和长度上的增加的多样性,不能通过序列相似性有效的预测,已被证明是对模型是很大的挑战。重链和轻链之间界面上的H3 loops的位置,使其构象依赖于链间方向(inter-chain orientation),鉴于H3 loop在结合中的核心作用,预测H3 loops结构的效果对于了解抗体-抗原相互作用,实现抗体的合理设计非常重要。
IgFold,先从一个预训练的语言模型提取预序列表征(该模型在558M自然抗体序列上进行的训练),然后通过图网络直接预测backbone atoms坐标。其预测结构质量与其他工具类似或更好(包括AlphaFold),且预测所需时间不到1分钟。在这个时间尺度上进行准确的结构预测,使得以前不可行的调研与验证方法成为可能。
同时为了证明IgFold的能力,作者预测了105K配对的8个抗体序列的结构,将观察到的抗体结构空间扩大了40倍以上。
油管视频
方法
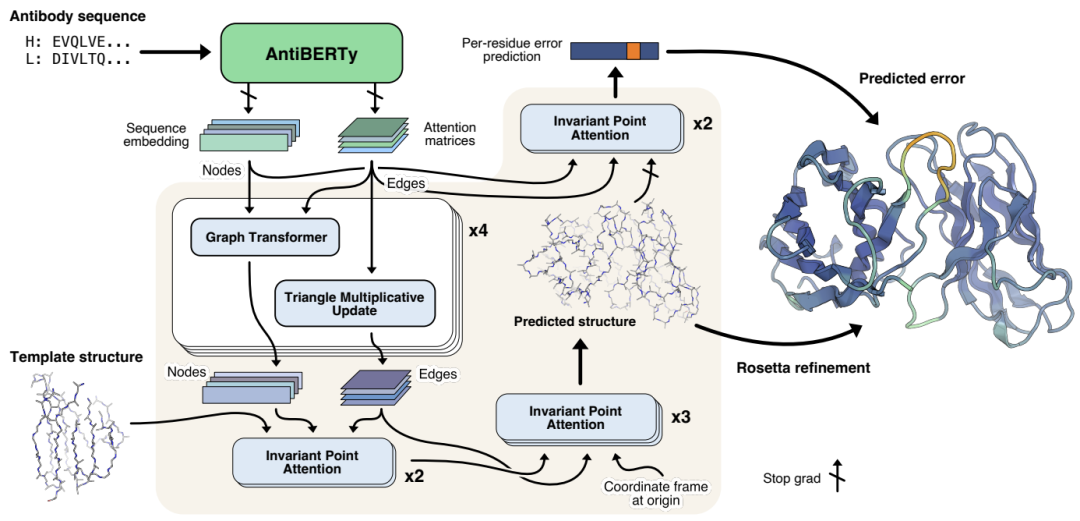
抗体结构端到端预测方法示意图。抗体序列使用AntiBERTy(一个预训练的语言模型)被转换成上下文嵌入表征。根据这些表征,IgFold使用一系列transformer层来直接预测蛋白质骨架原子的原子坐标。每个残基,IgFold也提供了预测质量的估计。Rosetta对预测的结构进行了精修,并添加了侧链。
A 从序列预测抗体结构
A.1 生成AntiBERTy编码
AntiBERTy是在558M抗体序列数据中训练的一个双向transformer掩码语言模型。给定一个序列,作者从AntiBERTy收集所有层的最终隐藏状态和注意力矩阵。隐藏层维度通过全连接层从L*512降为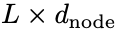
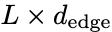
A.2 IgFold模型的应用
IgFold模型输入是per-residue embeddings(nodes)和inter-residue attention feature(edges)。
这些原始的特征通过graph transformer layers[1]进行了一系列的节点更新,通过triangular multiplicative operations[2]做了边的更新。
接下来,通过固定坐标不变的invariant point attention[2]合并模板数据。最后,利用处理后的节点和边通过不变点注意力预测抗体骨架结构。
A.3 训练过程
模型序列组合了结构预测和误差估计损失项。原始的结构预测损失使用residue frame atom coordinates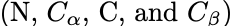
A4. 结构预测集成
给定序列,预测了四个方法(不同的随机数设置),然后根据预测的误差在四个方法预测的结果中选择一个。
B Benchmarking 抗体结构预测方法
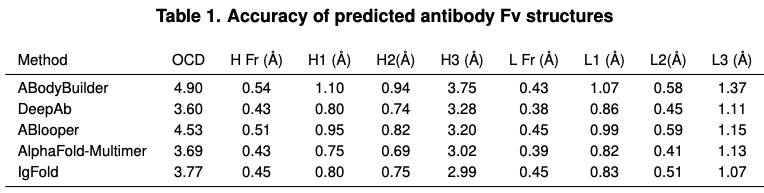
作者选择了四个可选的抗体结构预测方法:ABodyBuilder, DeepAb, ABlooper, and AlphaFold。ABodyBuilder结构预测采用web server,预测删除了2021年7月1号之后的数据以及大于70%的序列相似性。DeepAb,每个序列采用推荐的5个decoys。ABlooper结构的CDR loops built on grafted frameworks来自ABodyBuiler。AlphaFold(和AlphaFold-Multimer)结构预测采用公开代码库。
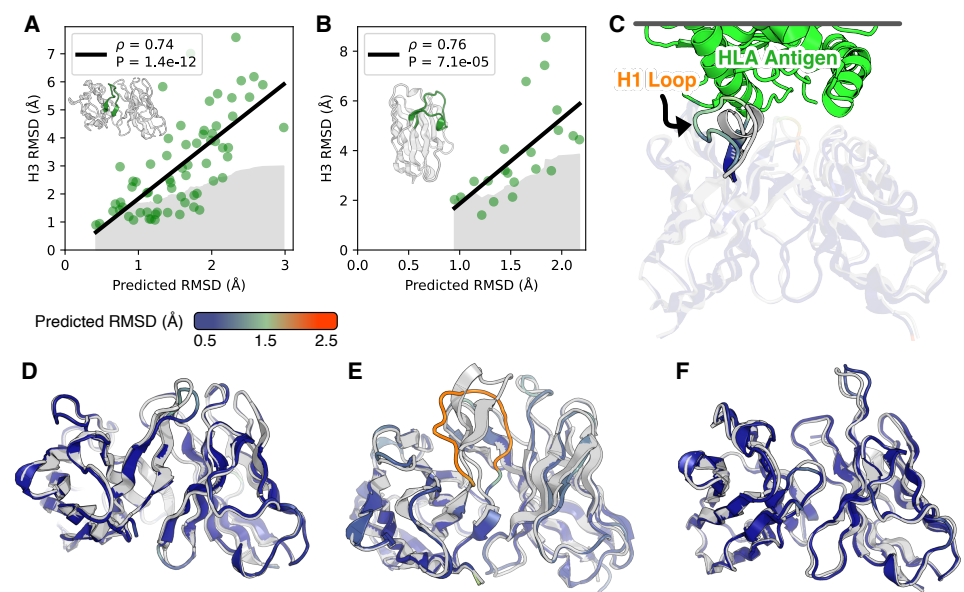
作者比较了四个方法预测的结构相似度。观察到在CDR1和CDR2 loops方面,IgFold、DeepAb,AlphaFold-Multimer平均上非常相似,但ABlooper和其他都不太一样,作者推测是模型架构准确度不够。
在速度层面,ABodyBuilder虽然最快,但是以牺牲准确率为代价。DeepAb和ABlooper虽然更准确,但是需要更多的时间预测全原子结构。IgFold预测速度比ABlooper更快,而且能够预测全原子结构。
在CDR H3预测准确率层面,AlphaFold-Multimer和IgFold稍微好一点。
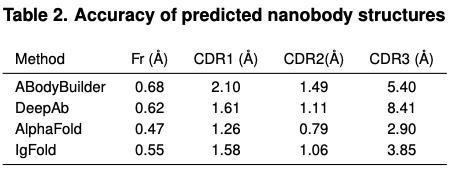
在纳米抗体预测层面,AlphaFold、IgFold预测效果更好。
C 预测误差与RMSD呈较高的相关性
讨论
(1)IgFold达到了AlphaFold-Multimer模型的精度用于成对的抗体结构预测,并接近了纳米抗体的AlphaFold的结果。
(2)IgFold显著快于AlphaFold,能在1分钟内预测结构。预测结构同时做了信息误差估计,这为结构的可靠性提供了关键信息。
(3)在很多target上面,IgFold和AlphaFold能够产生不同的构象,对结构集成时,这有很有用,因为在应用的时候,灵活性非常重要。
(4)预测的结构同时做了信息误差估计,可以降低假阳性,提供了关于结构可靠性的关键信息。
(5)传统上,免疫系统的分析仅限于序列数据,因为高通量抗体结构测定在实验上不太可能的,预测方法太慢且不准确。结合结构预测的信息对于识别常见表位的序列不相似结合物很有价值。
(6)对已有抗体抗原对接任务也有较大的帮助,对现有对接方法,IgFold在速度和准确率上会有较大的改善,在新的对接方法,IgFold的误差估计有助于指导增强采样。
IgFold与AlphaFold、RoseTTAFold有相同的应用场景,同时能额外对抗体设计提供测试和打分方法。
从IgFold提取的编码(特别是当从模板中注入结构信息时)可以作为有用的特征用于未来的抗体设计任务。
参考文献
[1] Yunsheng Shi, Zhengjie Huang, Shikun Feng, Hui Zhong, Wenjin Wang, and Yu Sun. Masked label prediction: Unified message passing model for semi-supervised classification. arXiv preprint arXiv:2009.03509, 2020.
[2] John Jumper, Richard Evans, Alexander Pritzel, Tim Green, Michael Figurnov, Olaf Ronneberger, Kathryn Tunyasuvunakool, Russ Bates, Augustin Žídek, Anna Potapenko, et al. Highly accurate protein structure prediction with alphafold. Nature, 596(7873):583–589, 2021.
代码与数据地址
https://github.com/Graylab/IgFold
抗体设计文章
ICLR 2022|迭代精调的GNN用于抗体-序列结构协同设计
补充代码地址:https://github.com/wengong-jin/RefineGNN