本文介绍由浙江大学智能创新药物研究院侯廷军教授/潘培辰研究员团队和中南大学曹东升团队联合在综合期刊Research上发表的一篇文章。该文章提出一种新型的基于图表示的深度学习方法DeepCoSI用于共价药物结合位点(半胱氨酸)的预测。DeepCoSI网络由两个模块构成,分别是PocketGNNLayer和CysInteractLayer,前者用于对半胱氨酸所在口袋的环境进行表征,后者用于表征半胱氨酸与周围原子之间的非键相互作用,二者结合实现了半胱氨酸共价可靶性的预测。作者在两个外部测试集上验证了该模型区分共价可靶半胱氨酸与其他半胱氨酸的能力,均表现出较好的预测效果。
研究背景
根据与靶标结合的不同方式,小分子抑制剂可以分为非共价抑制剂和共价抑制剂(TCI)两大类。共价抑制剂与靶标的结合亲和力强,在结合位点具有较长的保留时间,有望解决部分靶标的选择性与不可靶向性的问题,也因此共价抑制剂受到了越来越多的关注。共价抑制剂与靶标的相互作用既包含非共价部分的非键相互作用,也包括共价弹头与结合口袋亲核氨基酸之间的共价成键作用。然而,我们对共价结合位点的认识非常有限,对于共价抑制剂的研究也局限在很少部分的靶标中。因此,开发准确快速分析蛋白质结构中氨基酸的共价可靶性的方法,可以扩大共价抑制剂的研究范围,加速共价药物的发现。
数据集
作者构建了一个基准数据集用于模型的训练和评估,包含1042个晶体结构,涉及259种蛋白,这些结构中有7490个半胱氨酸,其中,1076个半胱氨酸连接有共价抑制剂,作为正样本,其余的半胱氨酸为负样本。为了尽可能降低训练集和测试集之间的相似性,在数据集划分之前,使用cd-hit对蛋白质进行聚类,根据聚类结果进行训练集、验证集和测试集的划分。
DeepCoSI模型
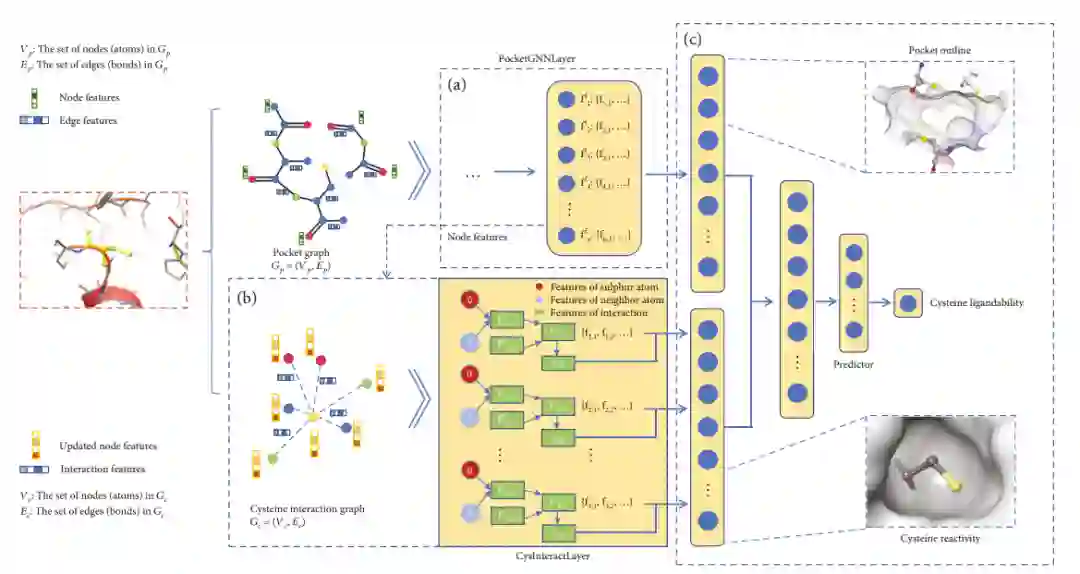
半胱氨酸的共价可靶性主要由以下两个方面决定:(1)其周围是否有合理的口袋便于药物分子的结合?(2)其是否有足够的内在反应性(亲核性)与亲电弹头发生共价反应?作者基于这两个问题设计了模型的结构框架,其中PocketGNNLayer模块用于表征半胱氨酸周围口袋的结构,CysInteractLayer模块用于表征半胱氨酸的内在反应性。模型结构如图所示:
图 1. DeepCoSI模型结构框架
作者截取目标半胱氨酸一定范围内(15埃)的氨基酸作为该半胱氨酸所在口袋(pocket),并将此口袋结构作为模型的输入。在PocketGNNLayer模块中,原子作为图结构中的节点,共价键作为图结构中的边,通过图卷积模块,将化学信息和三维结构特征转化为表征“口袋轮廓”的向量;在CysInteractLayer模块中,半胱氨酸的巯基与周围原子之间的隐式非共价相互作用作为图结构中的边,通过图卷积模块产生巯基的特征向量,用于表征半胱氨酸的“内在反应性”。最后用“口袋轮廓”与“内在反应性”特征共同预测半胱氨酸的共价可靶性。
实验
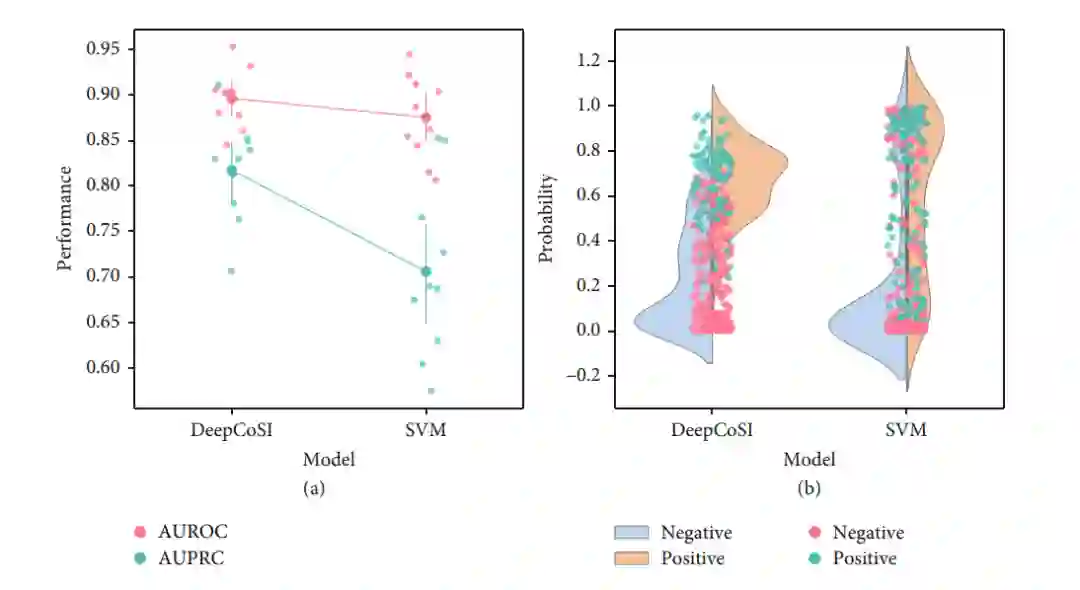
作者首先对比了DeepCoSI与基于描述符的传统支持向量机模型的预测能力,采用相同的数据集进行训练和测试,二者的AUPRC值分别为0.82和0.71。进一步查看二者对正负样本预测值的分布差异,SVM模型对负样本的识别能力比较强,预测值主要分布在0-0.2之间,而正样本则均匀分布在0-1之间,由DeepCoSI得到的正负样本的预测值则具有明显不同的分布。这说明,与传统模型相比,DeepCoSI具有更强的区分共价可靶半胱氨酸与其他半胱氨酸的能力。
图2. DeepCoSI与支持向量机模型的性能对比
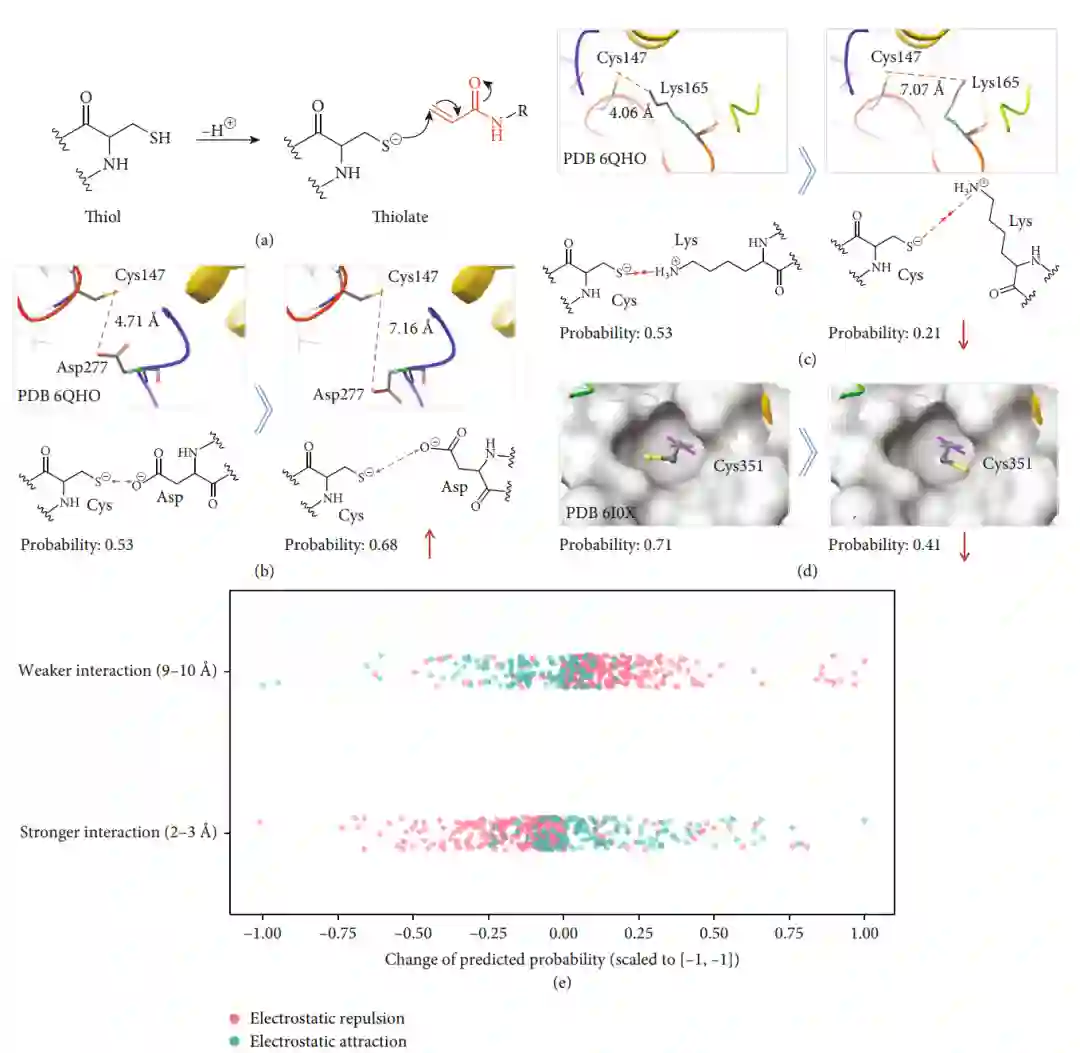
随后,作者通过微调输入的口袋结构,来查看DeepCoSI是否学习到了共价可靶半胱氨酸的隐范式。半胱氨酸的内在反应性与静电相互作用有密切关系,因此,通过调节其与周围带电氨基酸之间的距离,可以改变其质子化状态,进而影响其共价可靶性。作者选取了几个正电氨基酸和负电氨基酸分别进行了调整,模型预测值的改变方向符合预期。作者又进一步对整个测试集进行了分析,得到相同的实验结果。这说明,模型可以感知任务相关的因素对结果的影响,从而识别出共价可靶半胱氨酸的隐范式。
图 3. DeepCoSI对输入口袋结构变化的响应
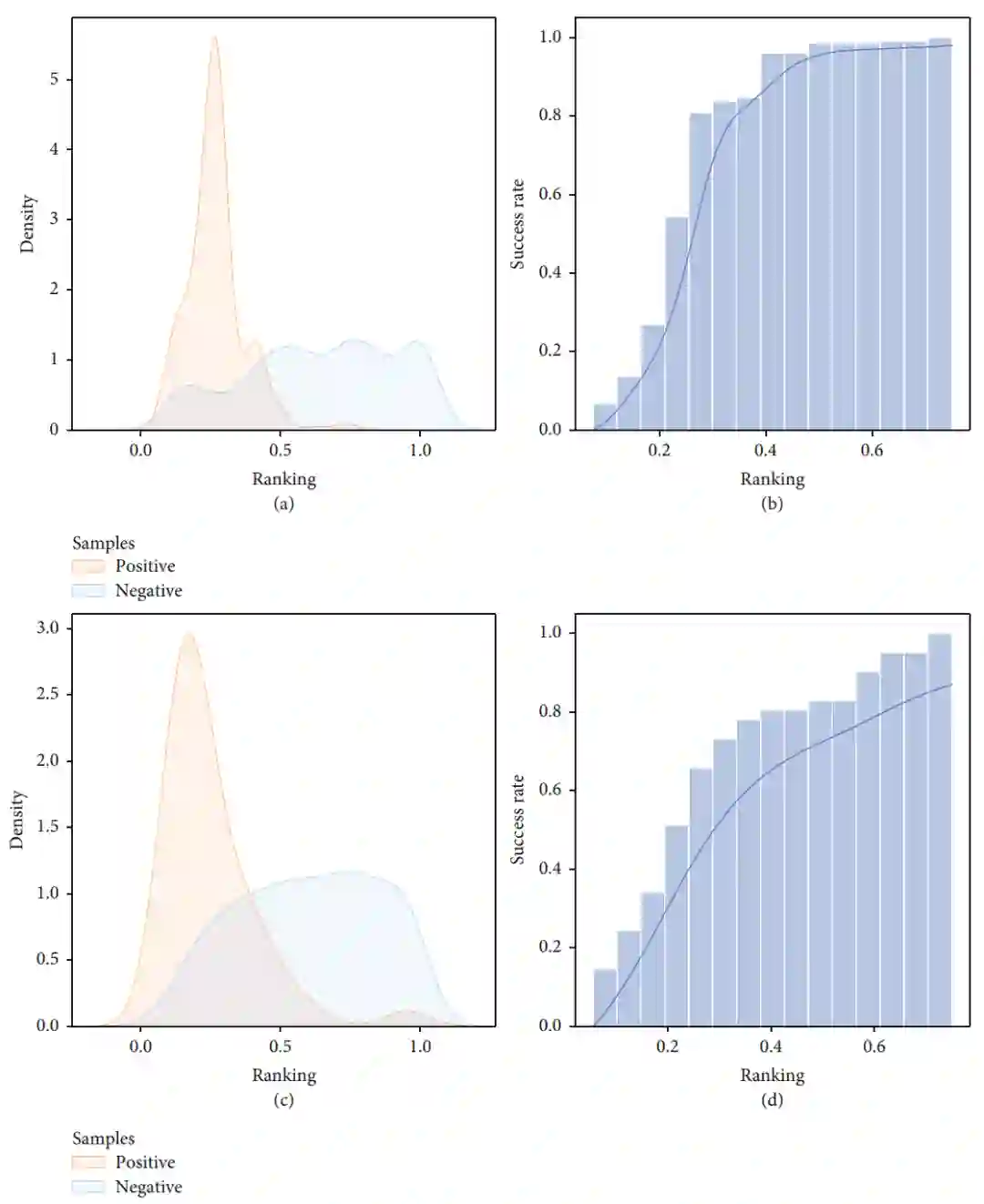
为了进一步验证模型在真实应用场景中的表现,作者构建了两个外部测试集。第一个外部测试集与原测试集的蛋白质种类相同,但所用晶体结构为apo形式(没有共价配体的结合)。第二个外部测试集来源于化学蛋白质组学数据(isoTOP-ABPP)。作者根据DeepCoSI的预测值给每个晶体结构中的半胱氨酸排序,结果显示,正负样本的排序值表现出明显的分布差异,这进一步说明在真实的应用场景中,DeepCoSI具有识别共价可靶半胱氨酸的能力。以归一化后的排序值(阳性样本排序/半胱氨酸数目)作为标准,当阈值设为0.5时,模型在两个测试集中的预测成功率分别可以达到98%和82.9%。
图 4. DeepCoSI在外部测试集上的表现
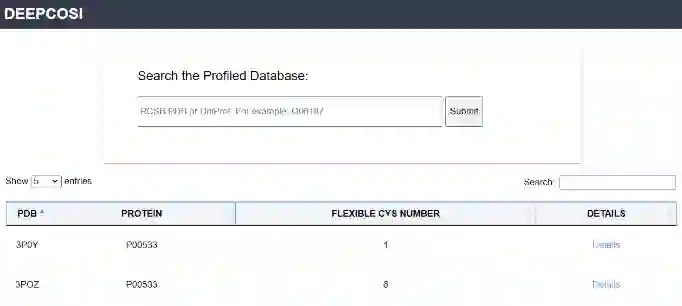
最后,作者将RCSB PDB中所有人源蛋白结构中的半胱氨酸进行了共价可靶性分析,研究人员可在共价抑制剂数据库CovalentInDB(http://cadd.zju.edu.cn/cidb/deepcosi/cys)中检索这部分预测数据。
图 5. 对RCSB PDB中半胱氨酸共价可靶性预测数据
总结
本文作者首次将深度学习应用在共价药物结合位点预测的问题上,构建了新型图神经网络模型DeepCoSI,结合物理化学特征和几何特征,分别对“口袋”和半胱氨酸“内在反应性”进行表征,模型能够学习到共价可靶半胱氨酸的隐范式,从而具备从结构中识别出共价可靶半胱氨酸的能力,将共价抑制剂研究范围扩大到蛋白质结构组学范围,促进新型共价药物的发现。 参考资料 Jiang, Y.Z., Wu, Z., Shen, C. and Lei Xu, D., Proteome-wide Profiling of the Covalent-Druggable Cysteines with a Structure-based Deep Graph Learning Network 2.