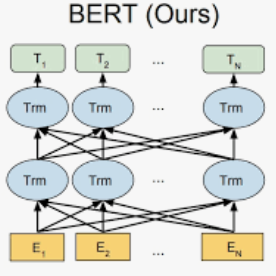
Pretrained language models such as Bidirectional Encoder Representations from Transformers (BERT) have achieved state-of-the-art performance in natural language processing (NLP) tasks. Recently, BERT has been adapted to the biomedical domain. Despite the effectiveness, these models have hundreds of millions of parameters and are computationally expensive when applied to large-scale NLP applications. We hypothesized that the number of parameters of the original BERT can be dramatically reduced with minor impact on performance. In this study, we present Bioformer, a compact BERT model for biomedical text mining. We pretrained two Bioformer models (named Bioformer8L and Bioformer16L) which reduced the model size by 60% compared to BERTBase. Bioformer uses a biomedical vocabulary and was pre-trained from scratch on PubMed abstracts and PubMed Central full-text articles. We thoroughly evaluated the performance of Bioformer as well as existing biomedical BERT models including BioBERT and PubMedBERT on 15 benchmark datasets of four different biomedical NLP tasks: named entity recognition, relation extraction, question answering and document classification. The results show that with 60% fewer parameters, Bioformer16L is only 0.1% less accurate than PubMedBERT while Bioformer8L is 0.9% less accurate than PubMedBERT. Both Bioformer16L and Bioformer8L outperformed BioBERTBase-v1.1. In addition, Bioformer16L and Bioformer8L are 2-3 fold as fast as PubMedBERT/BioBERTBase-v1.1. Bioformer has been successfully deployed to PubTator Central providing gene annotations over 35 million PubMed abstracts and 5 million PubMed Central full-text articles. We make Bioformer publicly available via https://github.com/WGLab/bioformer, including pre-trained models, datasets, and instructions for downstream use.
翻译:诸如来自变换器(BERT)的Bidirectional Ecoder Spresents (BERT) 等预选语言模型在自然语言处理(NLP)任务中达到了最先进的性能。 最近, BERT 已经适应到生物医学领域。 尽管这些模型具有效力, 这些模型有数亿参数, 在应用到大规模 NLP 应用程序时计算成本昂贵。 我们假设原始BERT 参数的数量可以大幅下降, 并对绩效影响较小。 在本研究中, 我们为生物医学文本开采提供了Biolexexex, 一个BERT 的常规BERT 模型。 我们预先培训了两个Bioforest 模型(名为BioExform8L 和 Bioexerform 16L), 与BioxerformalexiveBM 相比, 将模型减少了60%。 我们彻底评估了BERM 以及现有的BERT 模型的性能表现, 包括Beiob-BERT 和Pub-Serferviorent Ex Ex-Blent mess mess mess 数据输出, 的模型中, 和Publexerviewlorlent mold mold mold mold mold mess real8, 提供比L 而不是 而不是 的精确的精确的精确的版本数据, 10B 。