导语
核酸(DNA和RNA)是非常重要的生物大分子,研究其空间结构对揭示其功能机理和指导相关生物医学应用(如药物设计、DNA纳米)至关重要。由于通过实验方法(如X-射线衍射、核磁共振、冷冻电镜)测定核酸的结构具有一定的局限性,目前已知的核酸结构数量非常有限,这也限制了诸如AlphaFold2的深度学习预测方法在核酸结构上的运用。而现有的核酸结构预测方法往往需要依赖有限的数据库结构或预先给定的二级结构,也忽略了核酸结构的高负电性。近期,发表于PLoS Computational Biology上的一篇论文提出了一个粗粒化模型,通过计入静电及序列相关的碱基堆积等相互作用,能够仅基于序列准确预测单/双链DNA在离子溶液中的空间结构。此外,该模型还能同时定量给出简单DNA结构的热力学稳定性及其中的离子效应。尽管该模型目前在处理复杂DNA结构上存在困难,但从头预测的提出能有效弥补现有方法的不足。****
关键词:核酸,空间结构预测,粗粒化模型
时亚洲** | 作者******
邓一雪** **| 编辑
论文标题:Ab initio predictions for 3D structure and stability of single- and double-stranded DNAs in ion solutions论文链接:https://journals.plos.org/ploscompbiol/article?id=10.1371/journal.pcbi.1010501
1. DNA结构还需要预测,
难道不是标准双螺旋吗?
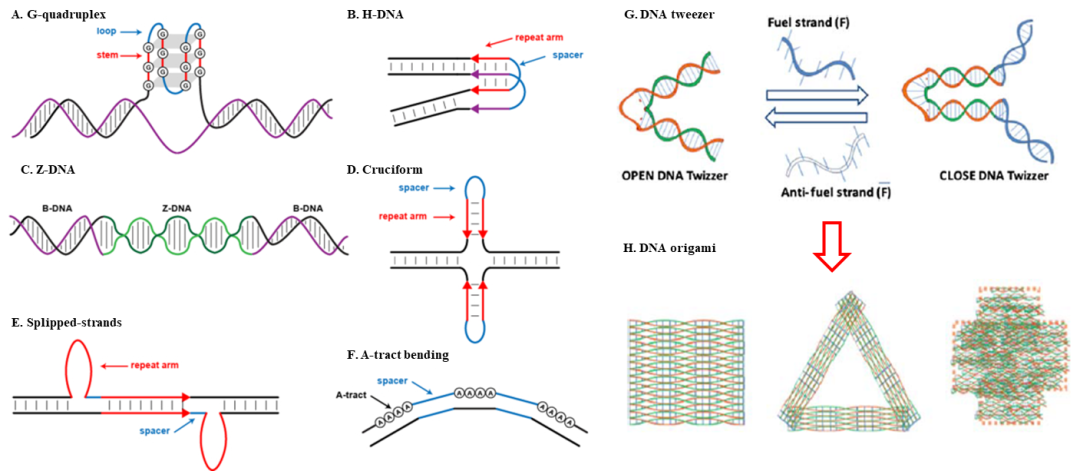
DNA是非常重要的生物大分子,负责遗传基因的存储和传递。然而,早在1953年J.D. Watson和F.H. Crick就指出位于染色体上的DNA通常呈现规则的右手螺旋结构(即B-型结构)[1],那么,如此标准的结构为何还需要预测呢?近期的一些研究发现,人类基因组中约13%的序列能折叠成非标准(即non-B型)的DNA结构(如发夹、假结、三链体、Z-DNA、G-四联体、i-motif等),以参与重要的细胞过程(如调控基因表达、协助端粒保护、促进突变、调控染色质的表观遗传、承担蛋白结合的靶点等),并与多种人类疾病有关 [2-4];见图1。例如,G-四联体结构的形成可以调节癌症相关基因的表达,从而拟制c-myc启动子域的转录灵活性,DNA三链体结构可以用于阻断各种致病基因的转录[4]。 此外,由于DNA结构的多样性,自组装DNA结构已被证明是设计与实现纳米结构及设备的优秀材料。目前已经实现的功能DNA纳米结构包括镊子、马达、穿梭器、折纸、逻辑电路、纳米容器等,它们在光子器件、药物输送、组织再生及生物成像等领域有着广阔的应用前景(图1)[5,6]。无论non-B DNA的折叠,还是DNA纳米的组装,都基于简单的DNA模体结构(如单链DNA发夹/假结及短链DNA螺旋)。因此,基于序列获取单/双DNA的空间结构对理解其新奇的生物功能及指导DNA新型纳米结构的设计具有重要意义。
图1. Non-B 型结构示例及典型DNA纳米结构展示[3-6]。
2. DNA结构研究进展
尽管现有的一些实验手段(如冷冻电镜、X射线衍射、NMR、光镊/磁镊等)能够用来测定DNA的空间结构或力学性能,但是由于其耗时耗力,在研究DNA折叠方面存在局限性。因此,人们开发了一些基于计算机的模型或方法以预测DNA的结构或探索DNA的结构折叠和热力学性质,如WLC模型[7]、3dRNA/DNA[8]、oxDNA[9]、TIS[10]、3SPN[11]、NARES-2P[12]、HiRE-RNA[13]等,但这些方法往往依赖DNA二级结构信息或预设的约束条件或有限的数据库结构。此外,由于DNA的聚电性质,溶液中的金属离子(如Na+和Mg2+)在DNA的折叠过程中发挥着非常重要作用,然而现有这些模型大都忽略了离子对DNA折叠的影响。因此,在该论文中,作者提出了一个新的DNA粗粒化模型,以仅基于序列预测简单DNA分子在离子溶液中的空间结构及其热力学稳定性。
3. 模型简介
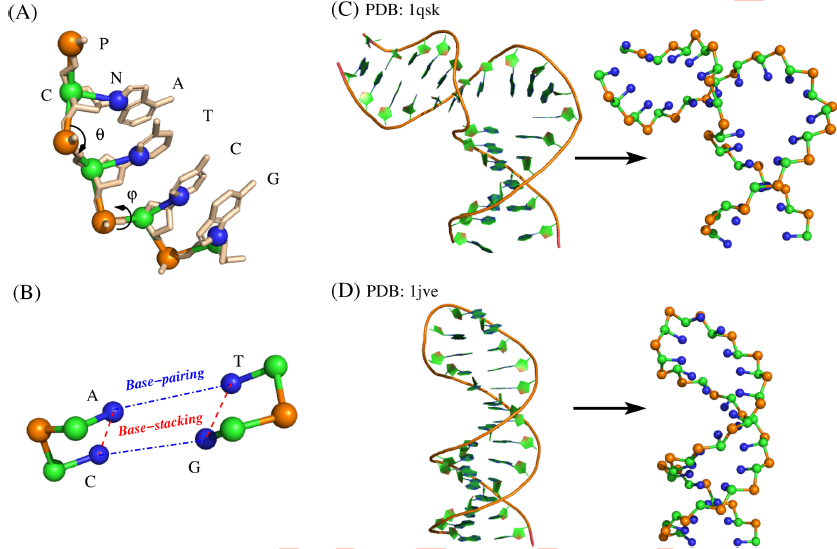
本文提出的DNA粗粒化模型是基于作者团队之前与武汉大学谭志杰教授合作开发的RNA模型的进一步推广[14-16]。该模型将DNA或RNA中的一个核苷酸简化为三个粗粒化的球原子,分别代表其中三个重要的功能基团:磷酸基团(P)、糖环(C4’)和碱基(N1或N9),并计入一个单位的负电荷,放置在P的中心(图2)。为了有效描述DNA的折叠,针对粗粒化模型,提出了如下能量函数: U=Ub+Ua+Ud+Uexc+Ubp+Ubs+Ucs+Uel
该能量函数共包含八个能量项,其中包括常规的近邻球原子之间的键长、键角、二面角等几何约束及非相邻球原子之间的体积排斥作用。除此之外,还计入了对核酸结构折叠至关重要的碱基配对(即氢键)和碱基堆积(即疏水、π-π)相互作用,以及相邻螺旋之间的共轴堆积作用。由于借助了比较成熟的实验热力学参数对碱基堆积作用项进行参数化,该模型能够比较准确地处理DNA结构及稳定性中的序列效应。更重要的是,该模型利用经典的Debye-Hückel近似,并结合离子凝聚理论和紧束缚离子理论,构建了能够处理单价、高价及单/双价混合离子与DNA结构相互作用的隐含离子模型,使得该模型能够用于预测离子条件对DNA结构及其稳定性的影响。
图2. DNA模型简化及碱基配对和碱基堆积作用示意图。
4. 模型验证
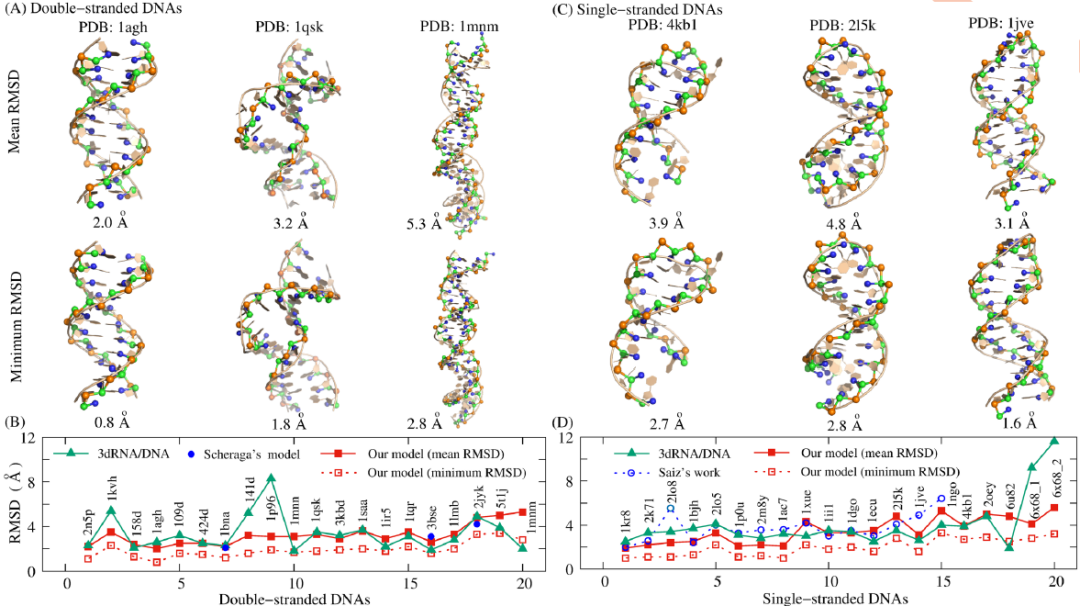
该模型与高效的蒙特卡洛模拟退火算法相结合,能够实现DNA由序列到结构的有效折叠。为了验证该模型的效果,作者分别选取了20个双链(18-52nt)和20 个单链(7-74nt)DNA序列,基于序列预测了其在1M NaCl条件下的空间结构,并与实验结构及现有模型(如3dDNA)的预测结果进行对比(图3)。结果表明,该模型能够比较准确地预测较短单/双链DNA的空间结构(如平均的RMSD接近3Å)。
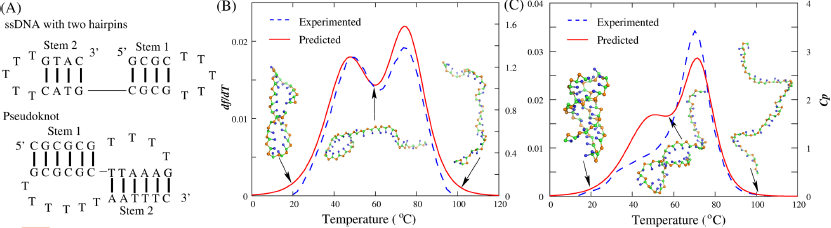
图3. 单/双链DNA预测结构展示及与现有模型结果的对比。 此外,作者还利用该模型定量预测27个双链DNA(包含凸环和内环等结构元素)和24个单链DNA(包括双发夹及假结结构)在相应实验离子条件下的热力学稳定性(图4)。预测的溶解温度与实验数据的平均偏差仅~2.0℃,同时系统分析了单/双链序列、长度、结构元素及双链的链浓度等因素对结构稳定性的影响。
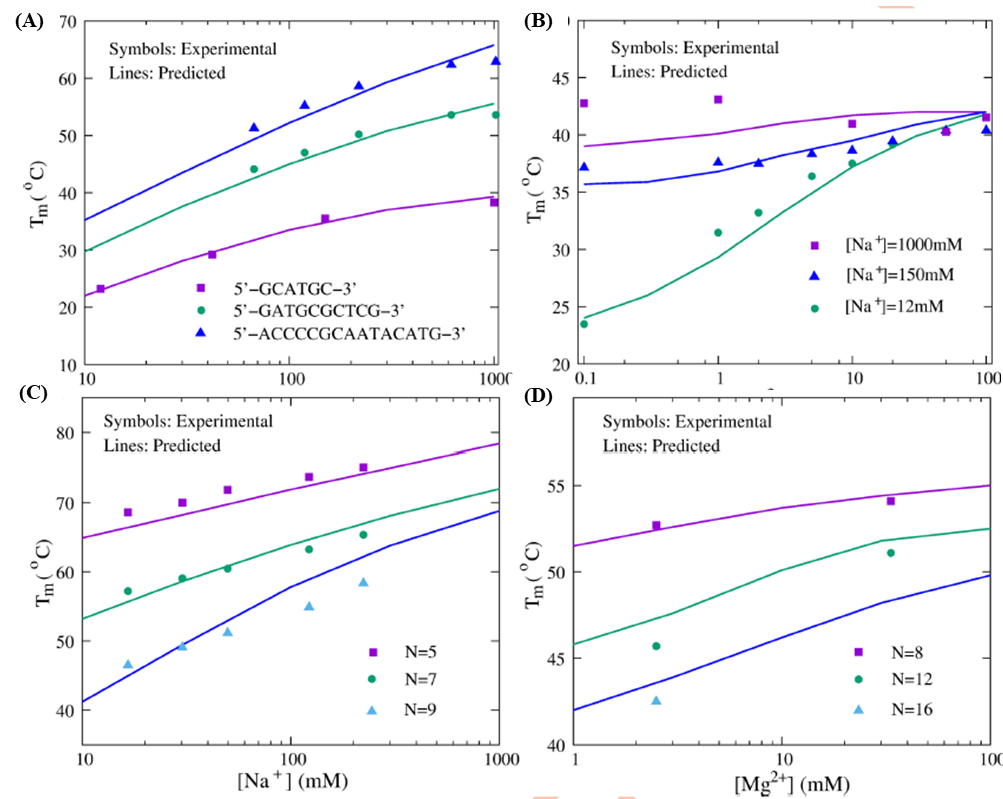
图4. 模型预测单链双发夹结构及假结结构热力学稳定性的结果展示。 最后,针对不同环长度和不同序列的单/双链DNA,分别预测了其在单价Na+、双价Mg2+及其二者混合离子条件下的结构稳定性,与相应实验数据的定量对比表明,该模型能够很好地给出简单DNA结构稳定性中的离子效应以及其中的单双价离子的竞争效应(图5)。
图4. 模型预测单/双链DNA结构稳定性随离子条件的变化结果展示。
5. 总结与展望
本文提出了一个DNA粗粒化模型,并利用该模型实现了离子溶液中单/双链DNA空间结构的从头预测,并通过与大量的实验数据及现有模型的预测结果进行直接对比,对模型在空间结构及其稳定性预测方面的性能进行了验证。尽管如此,目前模型在预测较大的复杂DNA结构方面仍然存在局限性,并且由于缺乏一些非标准的碱基-碱基等相互作用项,该模型也难以预测诸如G四联体、i-motif等非常规DNA的结构。因此,针对能量函数及抽样方法的进一步改进仍需进行。
参考文献
- Watson JD, Crick FH. Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid. Nature. 1953, 171:737-8.
- Bansal A, Kaushik S, Kukreti S. Non-canonical DNA structures: Diversity and disease association. Front Genet. 2022,13:959258.
- Crespi M, Ariel F. Non-B DNA structures emerging from plant genomes. Trends Plant Sci. 2022, 27:624-626.
- Guiblet WM, Cremona MA, Harris RS, Chen D, Eckert KA, Chiaromonte F, Huang YF, Makova KD. Non-B DNA: a major contributor to small- and large-scale variation in nucleotide substitution frequencies across the genome. Nucleic Acids Res. 2021, 49:1497-1516.
- Ma W, Zhan Y, Zhang Y, Mao C, Xie X, Lin Y. The biological applications of DNA nanomaterials: current challenges and future directions. Signal Transduct Target Ther. 2021, 6:351.
- Sharma A, Vaghasiya K, Verma RK, Yadav AB. DNA nanostructures: chemistry, self-assembly, and applications. Curr Prospects Future Trends Micro Nano Tech. 2018, 71-78.
- Marko JF, Siggia ED. Fluctuations and supercoiling of DNA. Science. 1994, 265:506-8.
- Zhang Y, Xiong Y, Xiao Y. 3dDNA: A computational method of building DNA 3D structures. Molecules. 2022, 27: 5936.
- Ouldridge TE, Louis AA, Doye JP. Structural, mechanical, and thermodynamic properties of a coarse-grained DNA model. J Chem Phys. 2011, 134: 085101.
- Chakraborty D, Hori N, Thirumalai D. Sequence-dependent three interaction site model for single- and double-stranded DNA. J Chem Theory Comput. 2018, 14: 3763–3779.
- Knotts TAt, Rathore N, Schwartz DC, de Pablo JJ. A coarse grain model for DNA. J Chem Phys. 2007, 126: 084901.
- He Y, Maciejczyk M, Oldziej S, Scheraga HA, Liwo A. Mean-field interactions between nucleic-acid base dipoles can drive the formation of a double helix. Phys Rev Lett. 2013, 110: 098101.
- Cragnolini T, Derreumaux P, Pasquali S. Coarse-grained simulations of RNA and DNA duplexes. J Phys Chem B. 2015, 117:8047–8060.
- Shi YZ, Wang FH, Wu YY, Tan ZJ. A coarse-grained model with implicit salt for RNAs: predicting 3D structure, stability and salt effect. J Chem Phys. 2014, 141:105102.
- Shi YZ, Jin L, Feng CJ, Tan YL, Tan ZJ. Predicting 3D structure and stability of RNA pseudoknots in monovalent and divalent ion solutions. PLoS Comput Biol. 2018, 14:e1006222.
- Jin L, Tan YL, Wu Y, Wang X, Shi YZ, Tan ZJ. Structure folding of RNA kissing complexes in salt solutions: predicting 3D structure, stability, and folding pathway. RNA. 2019, 25:1532–1548.
(参考文献可上下滑动查看)
本文作者所在单位:武汉纺织大学、非线性科学研究中心、应用数学与交叉科学研究中心(团队主页:https://nonlinear.wtu.edu.cn/)