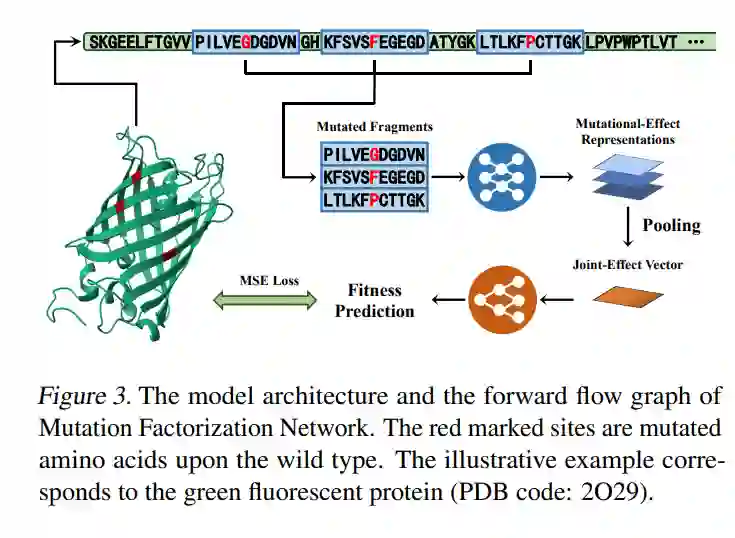
本文研究了预训练模型的生物序列反向设计。设计具有特定生物功能的蛋白质序列是蛋白质工程的一个长期挑战。本文研究了模型指导下的序列设计的探索机制。本文利用了蛋白质fitness landscape的一个自然属性,即在野生型序列上的一组简单突变通常足以增强所需的功能。利用这一特性,本文提出了近端探索PEX算法,该算法在蛋白预训练语言模型TAPE的基础上采用近端优化框架,优先寻找突变次数少的功能突变体。此外,本文构建了一个专门的模型架构MuFacNet来预测低阶突变效应,这进一步提高了模型引导的进化搜索的样本效率。本文在一套蛋白质序列设计任务中对本文的方法进行了广泛的评估,并证明了比基线算法的实质性改进。
论文下载:https://doi.org/10.1101/2022.04.12.487986
成为VIP会员查看完整内容
相关内容
Arxiv
26+阅读 · 2020年12月29日