近日,北京大学化学与分子工程学院、北大-清华生命联合中心、北京大学合成与功能生物分子中心王初课题组与北京大学生命科学学院、蛋白质与植物基因研究国家重点实验室、北京大学生物医学前沿创新中心苏晓东课题组合作在Nature Chemical Biology杂志上发表了题为“Co-evolution-based prediction of metal-binding sites in proteomes by machine learning”的研究文章。在这项工作中,作者开发了一种名为MetalNet的计算方法,采用机器学习方法通过蛋白质共进化信号进行金属结合位点预测,为研究金属蛋白质组和金属生物学提供了新的工具。金属离子具有独特的物理和化学性质,因而经常被用来稳定蛋白质结构,参与物质运输,作为蛋白质中的辅助因子来帮助催化生化反应和信号传导等等,在许多生物过程中起着不可或缺的作用。据估计,整个蛋白质组中超过三分之一的蛋白是金属结合蛋白,然而通常只有少数氨基酸残基参与金属结合,因此准确预测金属结合蛋白及其结合位点并非易事。对蛋白质组中金属结合蛋白以及具体金属结合位点的鉴定有助于加深人们对蛋白质功能的理解和认识,对后基因组时代的基础生物学和工业应用大有裨益。目前,研究人员开发了多种实验方法来直接探索蛋白质组中的金属结合蛋白,其中包括电感耦合等离子体质谱(ICP-MS)、基于活性的蛋白质分析(ABPP)和金属同位素天然放射自显影 (MIRAGE)等。此外,还有许多生物信息学方法可以根据序列或/和结构信息预测金属结合位点。例如,Claudia Andreini等人通过与已知金属蛋白的序列和结构同源性检查来系统预测人蛋白质组中的锌结合蛋白【Andreini, C., Banci, L., Bertini, I. & Rosato, A. Counting the zinc-proteins encoded in the human genome. J. Proteome Res. 5, 196–201 (2006).】;MetalDetector算法能够从蛋白质一级序列中识别过渡金属结合位点中的半胱氨酸和组氨酸【Passerini, A., Lippi, M. & Frasconi, P. MetalDetector v2.0:predicting the geometry of metal binding sites from protein sequence. Nucleic Acids Res. 39, W288–W292 (2011).】;MIB算法通过将所查询的未知蛋白质的片段与已知的金属结合蛋白进行结构比对来对蛋白质上金属结合位点进行预测【Lin, Y. F. et al. MIB: metal ion-binding site prediction and docking server. J. Chem. Inf. Model. 56, 2287–2291 (2016).】。然而,从蛋白质组中通过计算方法发现没有序列或结构同源性的新型金属蛋白仍然具有挑战性。最近,通过从多序列比对中计算得到残基之间的共进化信号并结合机器学习,科学家们可以实现对蛋白质结构和蛋白-蛋白相互作用的精准预测【Ovchinnikov, S. et al. Protein structure determination using metagenome sequence data. Science 355, 294-298 (2017).;Cong, Q., Anishchenko, I., Ovchinnikov, S. & Baker, D. Protein interaction networks revealed by proteome coevolution. Science365, 185-189 (2019); Jumper, J., Evans, R., Pritzel, A. et al. Highly accurate protein structure prediction with AlphaFold. Nature 596, 583–589 (2021).】。受此启发,作者探究了共进化信号在蛋白质金属结合位点上的分布情况,并发展了基于共进化信号和机器学习预测蛋白质组中金属结合蛋白和金属结合位点的计算方法。
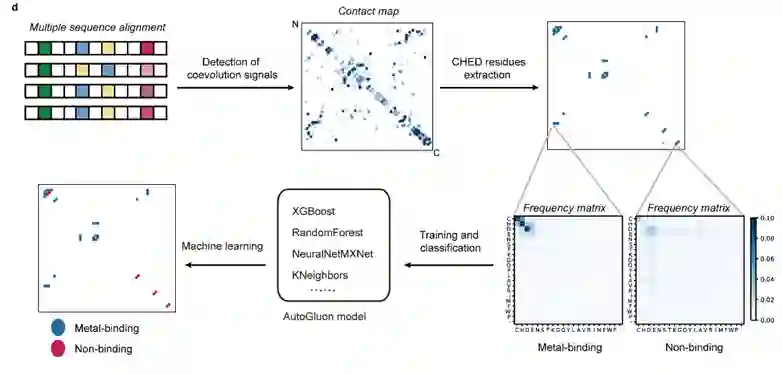
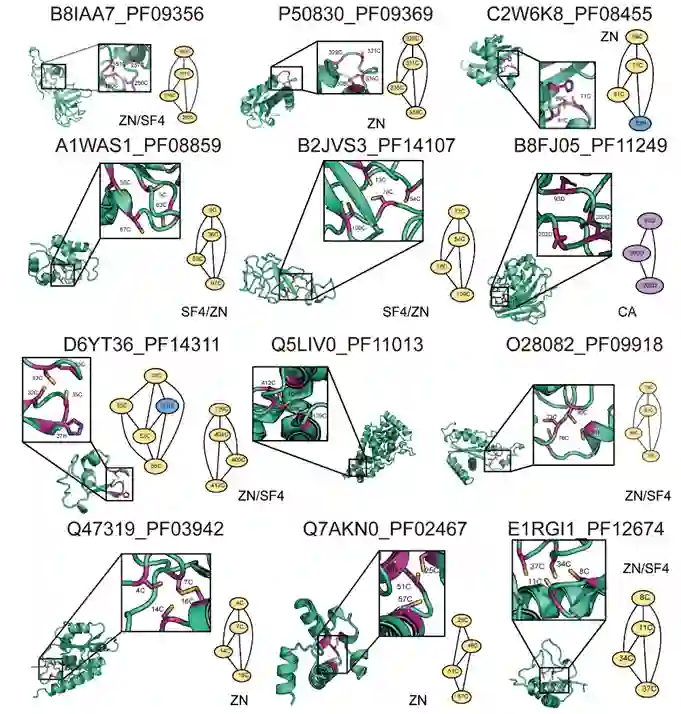
作者首先分析了蛋白质结构数据库PDB中金属蛋白结合位点的残基组成,发现91%的金属结合残基是由半胱氨酸、组氨酸、谷氨酸、天冬氨酸这四种氨基酸组成(简称“CHED”)。作者统计了由这四种残基两两组合而成的十种共进化的残基对作为金属结合位点的比例,发现共进化信号在这些金属结合位点中高度富集。因此,作者以残基对的氨基酸类型频率矩阵作为输入,将金属结合的CHED共进化残基对作为正样本,将非金属结合的CHED共进化残基对作为负样本,用于训练机器学习模型。最终得到的预测模型效果良好,平均AUC(受试者工作特征曲线下面积)为0.88,F1值为0.66。
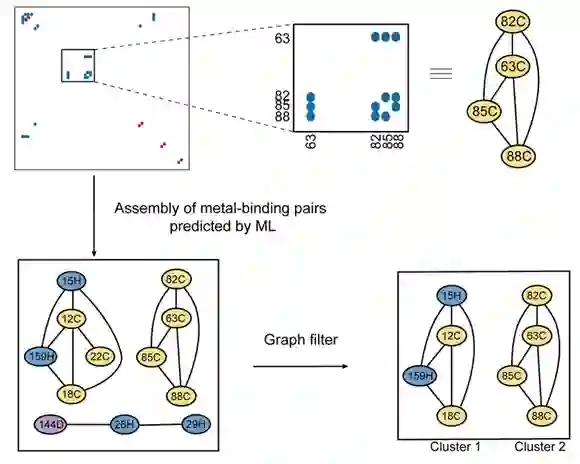
鉴于金属结合位点通常有多个残基与金属离子配位,作者将机器学习模型预测的残基对组装成残基网络,通过基于图模型的过滤器得到一个相对完整的网络簇,大大提高了对金属结合位点预测结果的可信度,将预测方法的平均F1值提升至0.72。当预测得到的网络簇的拓扑结构足够完整时,还可以通过与已知金属结合蛋白所包含的共进化残基网络进行比对检索,从而推断出该位点可能结合的金属离子类型。
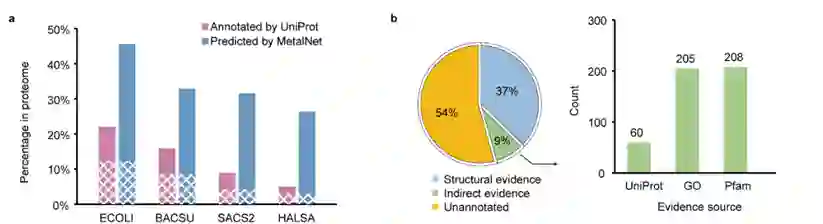
**作者将该预测方法命名为MetalNet,并将该方法首先对包括两种细菌与两种古细菌的原核物种蛋白质组进行预测。在这四种代表性原核物种中,MetalNet总共预测得到4849个可能的金属结合蛋白,这些预测的潜在金属结合蛋白中接近一半可以被同源蛋白中的结构直接支持或从其他蛋白质数据库中获得间接支持。**接下来,作者利用宏基因组测序数据得到的共进化信号,对Pfam蛋白质家族数据库中此前缺少结构的蛋白质家族进行了预测。MetalNet在1116个家族代表性蛋白中预测到175个具有共进化残基网络簇的金属结合蛋白。
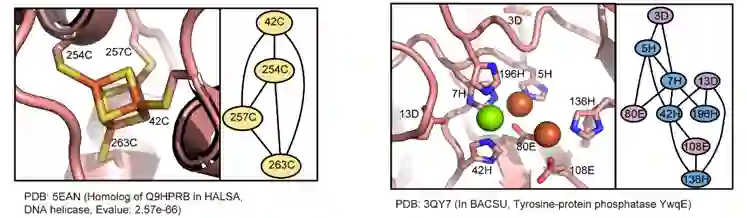
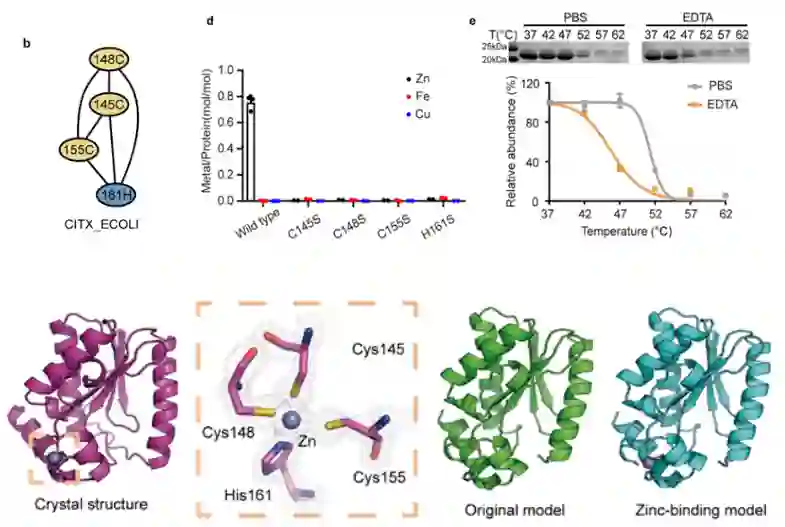
**作者在生化和结构水平上验证了几个之前未被注释的 MetalNet预测的金属结合蛋白,其中包括一个来自大肠杆菌的蛋白citX。MetalNet在citX 中预测到一个由三个半胱氨酸(C145、C148 和 C155)和一个组氨酸H161组成的共进化网络,并且网络拓扑结构与锌结合位点相匹配。**作者在大肠杆菌中表达纯化了野生型 citX 以及四个单突变体(C145S、C148S、C155S 和 H161S)。ICP-MS分析证实野生型蛋白质特异性结合锌离子,并且单突变体中未检测到锌结合信号。当用金属螯合剂 EDTA 处理野生型citX后,蛋白热稳定性曲线向左移动,表明蛋白质在丢失锌离子后变得不稳定。最后,作者通过 X 射线晶体衍射解析了 citX 的结构,明确地证明了citX 是一个锌离子结合蛋白,而且蛋白中的四面体锌结合位点与MetalNet的预测相符。此外,作者还基于MetalNet预测结果利用蛋白质建模软件Rosetta对citX进行重新建模,得到新的结合锌离子的结构模型与后来通过X射线晶体衍射得到的晶体结构能够精确匹配。
最后,作者将MetalNet应用于人剪接体蛋白质组的预测,发现目前已经解析的剪接体蛋白结构中报道的所有已知锌离子结合位点都可以被捕获,展示了MetalNet预测真核蛋白金属结合位点的潜力。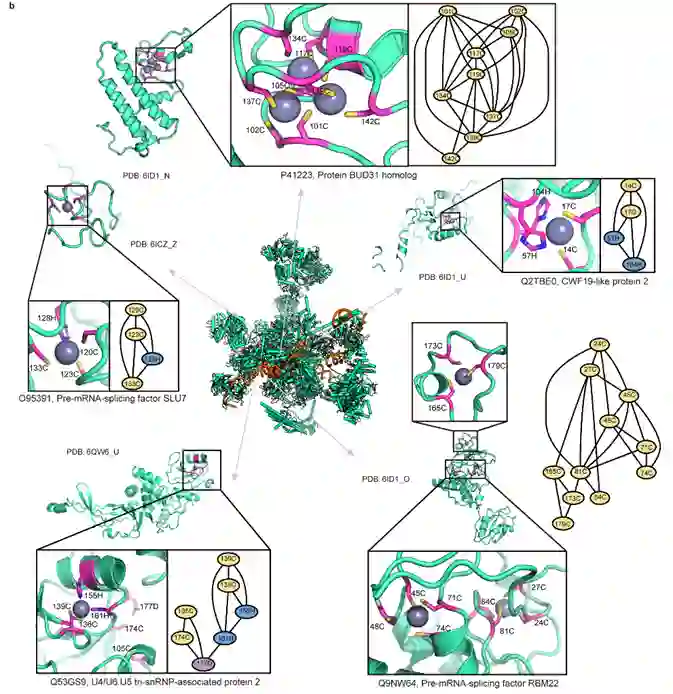
本文作者:程瑶 责任编辑:WQW 文章链接: https://www.nature.com/articles/s41589-022-01223-z 原文引用:DOI: 10.1038/s41589-022-01223-z