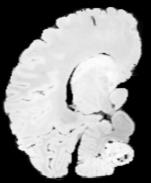
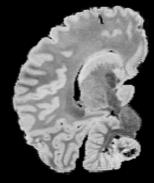
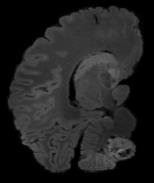
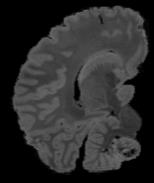
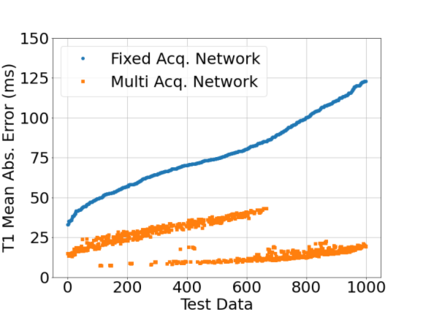
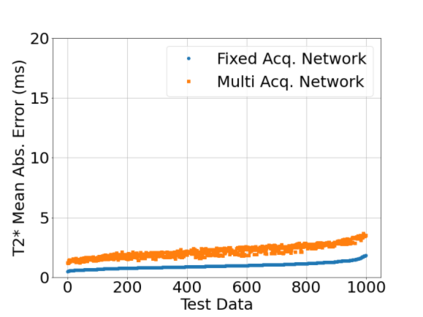
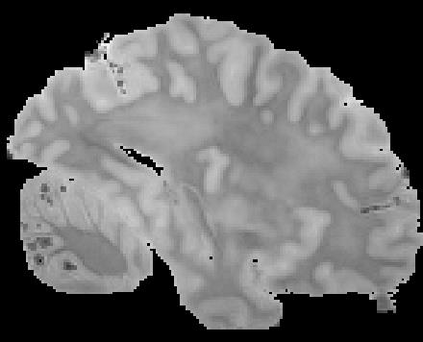
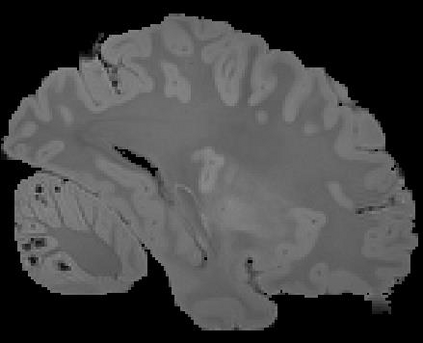
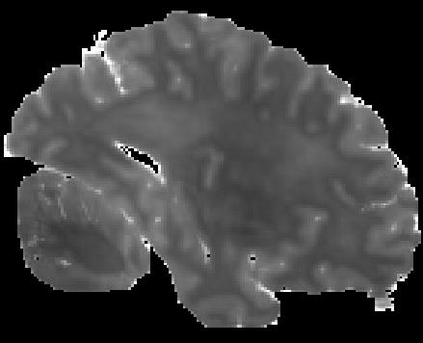
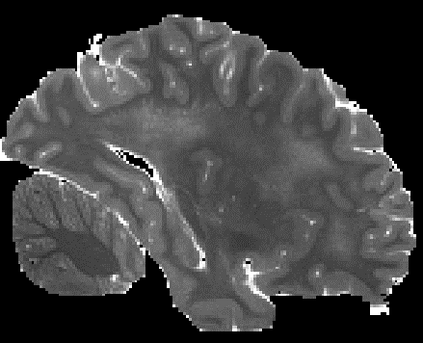
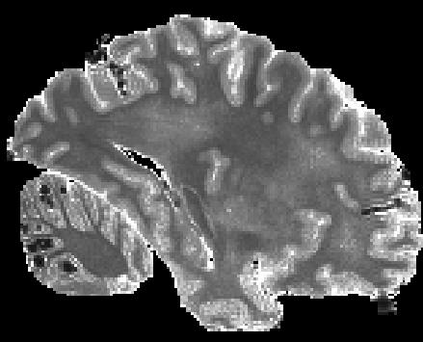
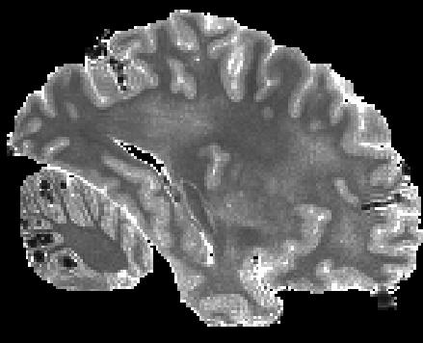
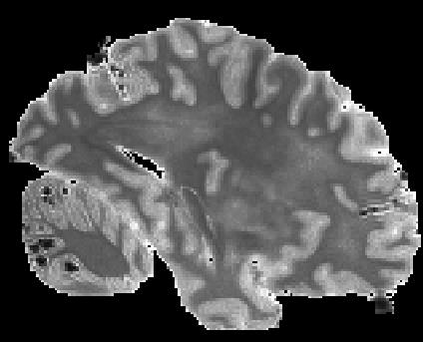
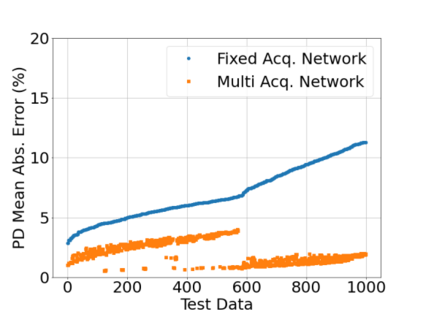
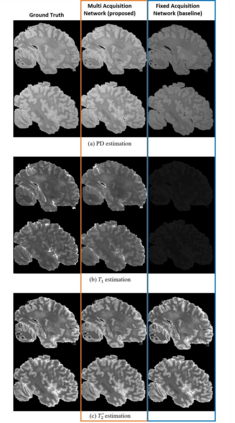
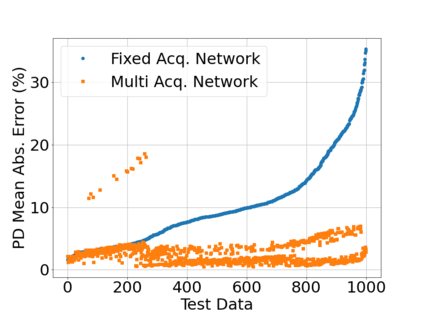
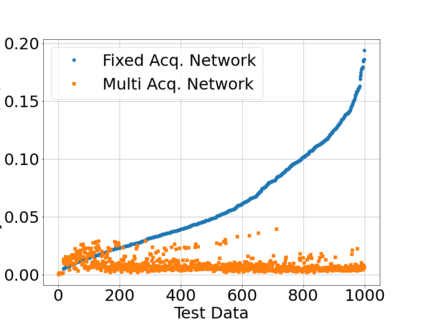
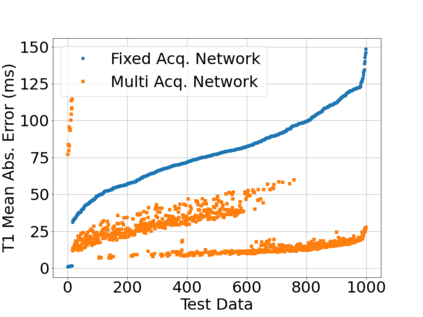
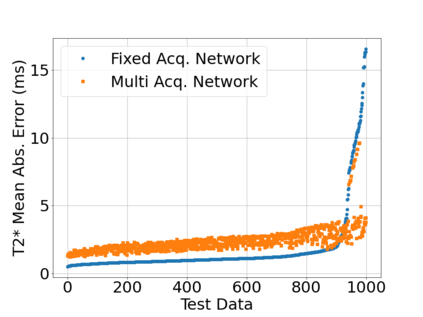
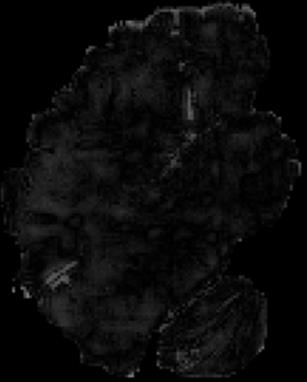
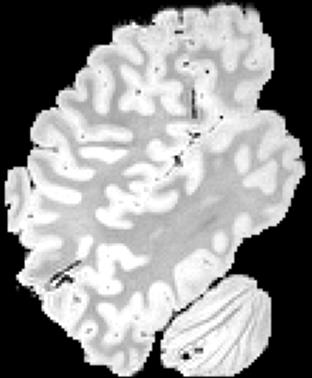
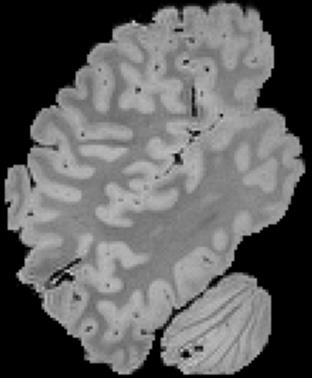
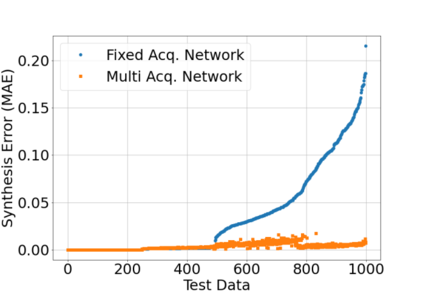
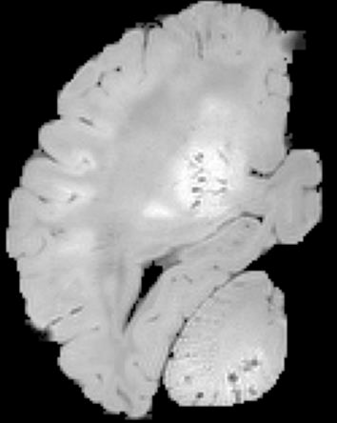
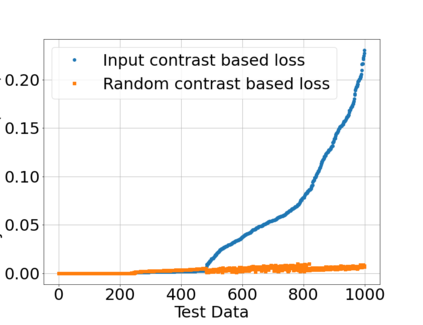
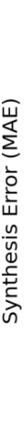In neuroimaging, MRI tissue properties characterize underlying neurobiology, provide quantitative biomarkers for neurological disease detection and analysis, and can be used to synthesize arbitrary MRI contrasts. Estimating tissue properties from a single scan session using a protocol available on all clinical scanners promises to reduce scan time and cost, enable quantitative analysis in routine clinical scans and provide scan-independent biomarkers of disease. However, existing tissue properties estimation methods - most often $\mathbf{T_1}$ relaxation, $\mathbf{T_2^*}$ relaxation, and proton density ($\mathbf{PD}$) - require data from multiple scan sessions and cannot estimate all properties from a single clinically available MRI protocol such as the multiecho MRI scan. In addition, the widespread use of non-standard acquisition parameters across clinical imaging sites require estimation methods that can generalize across varying scanner parameters. However, existing learning methods are acquisition protocol specific and cannot estimate from heterogenous clinical data from different imaging sites. In this work we propose an unsupervised deep-learning strategy that employs MRI physics to estimate all three tissue properties from a single multiecho MRI scan session, and generalizes across varying acquisition parameters. The proposed strategy optimizes accurate synthesis of new MRI contrasts from estimated latent tissue properties, enabling unsupervised training, we also employ random acquisition parameters during training to achieve acquisition generalization. We provide the first demonstration of estimating all tissue properties from a single multiecho scan session. We demonstrate improved accuracy and generalizability for tissue property estimation and MRI synthesis.
翻译:在神经成像学中,MRI组织特性是基本神经生物学的特征,为神经疾病检测和分析提供定量生物标志,并可用于合成任意MRI对比。使用所有临床扫描仪上可用的规程从单一扫描会话中估算组织属性,这有可能减少扫描时间和成本,在常规临床扫描中进行定量分析,并提供疾病依赖扫描的生物标志。然而,现有的组织属性估计方法――最常见的是$mathbf{T_1}放松,为神经疾病检测和分析提供定量生物标志,并可用于合成质子体密度($mathbf{T_2 ⁇ _2美元)――需要多次扫描会话的数据,无法从单一临床可用的MRI协议中估算所有属性。此外,广泛使用非标准获取参数对各种扫描仪参数进行概括。然而,现有的学习方法是专门获取协议的,无法从不同成像站点的随机性临床数据中估算。在这项工作中,我们建议从一个不精确的深度学习战略,即利用MRI的常规性能评估,从一个通用的购买性能评估,从一个通用的精度测试到一个通用的精度分析,从一个通用的精度分析系统,从一个总的精度评估整个的精度,从MRI的精度分析,从一个全的精度,从一个全的性能性能性能性能性能性能性能性能性能性能性能性能性能性能学到一个从一个从一个全部到一个从一个部到三个组织的计算,从一个全的精度学,从一个全的精度学学系,从一个从一个全的性能性能性能性能性能性能性能性能性能性能性能性能学学,从一个全性能学,从一个全的精度学到一个从一个全的精度学,从一个性能学学,从一个全学,从一个性能学,从一个性能学,从一个性能学测测测测测测测测测测测测测测测测测测测测测测测测算到一个全。