【Nature通信】基于知识图谱推荐框架识别EGFR突变型非小细胞肺癌耐药的驱动因子
随着生物医学数据呈指数级增长,经典的推荐系统方法移植至生物医学领域呈现出巨大潜力,可以帮助研究者快速浏览数据并从中进行知识推理。
耐药性阻碍肿瘤治疗的发展。本研究关注激活表皮生长因子受体(EGFR)突变的非小细胞肺癌(NSCLC)。NSCLC占肺癌患者的15-20%,使用第一代或第二代EGFR- TKI(如吉非替尼、安罗替尼或阿法替尼)进行治疗的患者往往初期疗效显著,但后期出现耐药。大多数耐药病例由于EGFR获得性耐药引起的,如出现T790M突变阳性。奥希替尼作为第三代EGFR-TKI,被用于EGFR-T790M突变阳性的局部晚期或转移性NSCLC的一线治疗。尽管奥希替尼疗效显著,但在6–26%的病例中,EGFR的获得性突变(如C797S)导致了奥希替尼耐药。
CRISPR-Cas9的基因敲除、敲降、敲入技术是系统性研究耐药机制有效的高通量技术。CRISPR筛选的典型输出是识别出的数百个耐药机制有关基因,随后进行人工分类和验证。在这个过程中,专家结合疾病的先验知识和临床、临床前证据,决定优先对哪些基因进行实验验证。选择过程繁琐耗时,且需要深厚的专业知识,因此容易出现个体偏差。
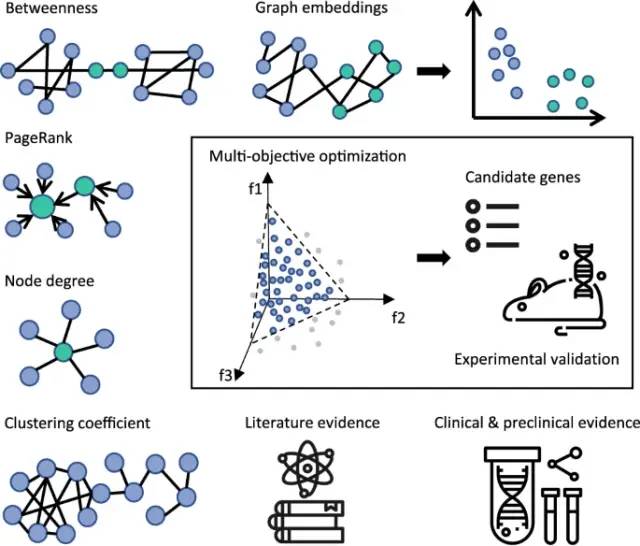
将问题移植到推荐系统领域面临两个主要挑战。第一缺乏训练数据;第二,与典型的推荐系统应用场景不同,该问题中缺乏显式和隐式反馈,逐步训练和改进模型的能力受限。考虑到以上问题采用无监督的,基于内容的推荐系统,将CRISPR筛选结果重新排序的任务变为一个多目标优化问题,支持基因相关性的各种相互矛盾的证据类型映射到目标。在优化过程中,可行解(基因)被识别和比较,直到找不到更好的为止,采用一组混合特征,每个特征代表一种不同类型的证据,并从BIKG知识图谱中获得辅助数据信息源。
https://www.nature.com/articles/s41467-022-29292-7
为了更快地找到耐药的关键驱动因素,我们在整合临床前、临床和文献证据的异构生物医学知识图谱之上构建了推荐系统。该推荐系统基于不同类型的证据之间的权衡对基因进行排序,这些证据将它们与EGFRi抗性的潜在机制联系起来。这种无偏的方法从> 3000个基因中识别出57个抗性标记,将识别时间从几个月减少到几分钟。除了复制已知的抗性标记,我们的方法确定了以前未探索的抗性机制,我们预期验证。
专知便捷查看
便捷下载,请关注专知公众号(点击上方蓝色专知关注)
后台回复“EGFR” 就可以获取《【Nature通信】基于知识图谱推荐框架识别EGFR突变型非小细胞肺癌耐药的驱动因子》专知下载链接