Nature 子刊|多癌症集成的图表示网络规约算法预测药物应答
近日,Nature子刊发表了一篇关于生物医学图谱的规约方法,作者应用GO-GO相似性将同一个聚类且属于同一个信号通路中的非激酶节点删减,使得最初信息稀疏的生物网络转换为信息更加丰富的图。
癌症细胞的基因谱对基因变异提供了宝贵的信息。由于癌症复杂的作用机制,仅基于遗传信息预测药物治疗对特定的细胞系的影响是有问题的。
作者设计了将多种异质数据(包括生物网络,基因组学,抑制剂分析和基因疾病关联)整合到统一图结构的流程中。
开发了一种新颖的graph reduction algorithm的癌症特异性网络。该系统融合了拓扑信息、生物学知识,graph reduction 增加了特征熵,同时保留了有价值的图特征信息。癌症特异性数据的非欧几里得表示提高了机器学习的性能,以预测癌症对药物治疗的反应。
将机器学习系统应用于生物网络的一个主要挑战是确保这些数据包含足够高的信噪比,以便学习框架有效的执行信息提取和高级归纳。此外,许多生物网络(例如包括数千个节点的PPI网络)的大小对于许多算法来说非常大,因此,最好缩小以促进快速学习过程。
原始网络有两类节点构成,激酶(用圆表示)和非激酶(用方框表示)。给定一个细胞系,基因表达上调用绿色表示,下调用红色表示,一些节点会赋予适应症基因的关联分数,如果该细胞系被激酶抑制剂处理,则针对其靶点的pIC50值将被添加到图表中(数字以斜体显示)。所以在图中,可以看到激酶节点会有基因表达值,一些激酶节点也会有pIC50值和疾病和基因关联分数。非激酶节点有基因表达值,一些非激酶节点也有疾病基因关联分数。请注意,所有细胞系药物组合具有相同的潜在PPI网络,然而,不同的细胞系通常具有不同的基因表达值和疾病关联分数,这取决于肿瘤类型。类似地,各种药物通常抑制不同的激酶组,因此,对于细胞系药物组合而言,节点特征通常是唯一的。
生物医学知识图谱的全网络分析
在机器学习的背景下,对应于原始PPI网络的全尺寸图不一定是特定癌症数据的最佳表示。
首先,所有的实例分享一样的图拓扑,仅仅在节点特征数据不同,比如基因表达,适应症关联,以及pIC50值,使机器学习模型难以收集有效学习所需的信息。第二,全图是非常稀疏的,浪费计算资源。第三,图中的大多数(98%)节点是无抑制数据的非激酶蛋白质,大多数蛋白质通常根据差异基因表达进行调节,导致重要特征显著稀疏。
因此,很多项的特征矩阵携带了非有效信息,导致很差的预测效果。
基于特定癌症网络的缩减
作者通过边的缩减设计了基于特定癌症网络缩减的流程。该想法是通过删除边,然后合并该边的关联节点以形成新节点。边收缩在计算任意连通图的生成树个数的递归公式中被广泛使用.在我们的例子中,仅仅基于连通性的简单边收缩不会产生期望的结果,因为还需要考虑节点的特征。
作者开发了一种基于知识的边缘收缩算法,利用连通性和生物特征信息来满足以下条件:两个事件节点都需要是非激酶蛋白,共享相同的差异基因表达,并且在相同的生物过程聚类中。
最后一个条件非常重要,可以确保缩减只合并属于同一信号通路的节点,从而支持生物学知识。肿瘤特异性网络中的生物过程由聚类节点根据其基因本体(GO)术语的相似性确定。
GOGO使用Directed Acyclic Graphs[1]对两个GO terms计算语义相似度。为了验证在使用从BP(biology process)本体中导出的GOGO相似性时,网络局部性得到了保留,我们首先计算了全PPI网络中一阶、二阶、三阶和四阶邻居之间的相似性值。
上图显示了,1阶邻居节点GOGO相似性最高,其他随着阶数升高,GOGO相似度降低。这些结果证实了之前的研究表明,两种蛋白质在网络中的距离越近,它们的生物学功能就越相似[2]。
接下来,使用GOGO相似性和层次聚类分析(HCA),将图中的所有蛋白质分为30个(HCA-30)、100个(HCA-100)和300个(HCA-300)聚类簇。在图缩减期间,只允许合并属于同一聚类中的节点。
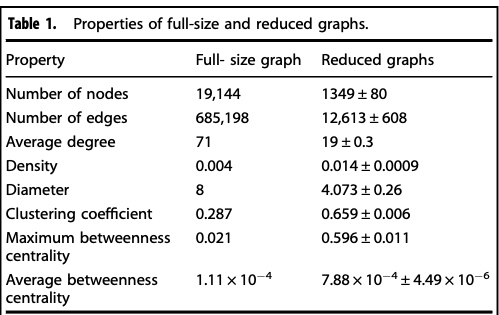
对生物医学图谱的全网络和缩减网络分析
图缩减方法极大的增加了所有359个癌细胞系的图拓扑和特征的多样性,同时保留了每个图中的中重要信息和生物学知识。
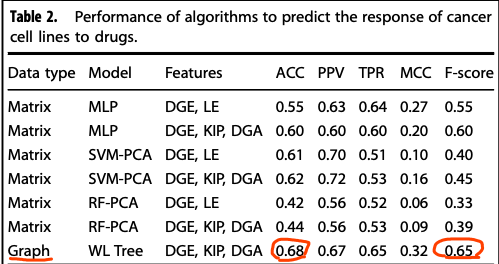
知识图谱癌症药物应答预测方法效果比较
基于图的算法(缩减的数据建模)与其他两种基于矩阵的方法(全网络数据建模)比较,图算法得到了最好的效果。
数据地址
https://osf.io/dzx7b/
参考文献
[1] Zhao, C. & Wang, Z. GOGO: An improved algorithm to measure the semantic similarity between gene ontology terms. Sci. Rep. 8, 15107 (2018)
[2] Sharan, R., Ulitsky, I. & Shamir, R. Network-based prediction of protein function.Mol. Syst. Biol. 3, 88 (2007)