【Nature. Mach. Intell. 】基于Transformer的多肽-HLA I类结合预测和疫苗的新生抗原序列设计
近日,国际知名期刊《Nature Machine Intelligence》在线发表了上海交通大学生命科学技术学院魏冬青团队的研究论文《A transformer-based model to predict peptide–HLA class I binding and optimize mutated peptides for vaccine design》。生命科学技术学院2017级博士研究生褚晏伊和香港大学深圳医院的张艳博士为该论文的共同第一作者。生命科学技术学院魏冬青教授和熊毅副研究员为该论文的共同通讯作者。
人类白细胞抗原(HLA)和肽(pHLA)之间相互作用的计算预测可以加快表位筛选和疫苗。该研究开发了TransMut框架,包含了用于pHLA结合预测的TransPHLA和用于突变肽优化的AOMP程序,它可以推广到生物分子的任何结合和突变任务(图1)。
TransPHLA设计了Transformer衍生模型来预测pHLA的结合。在pHLA结合预测、新抗原鉴定和HPV疫苗鉴定方面,TransPHLA均优于现有的14种方法。基于TransPHLA开发的AOMP程序可用于疫苗设计,它可以自动优化突变肽,以搜索对目标HLA具有更高亲和力并与源肽具有高度同源性的突变肽。在3660个非结合pHLA中,有3630个源肽被成功突变。其中,94%通过IEDB的推荐方法得到验证,88%与源肽的同源性高于80%。
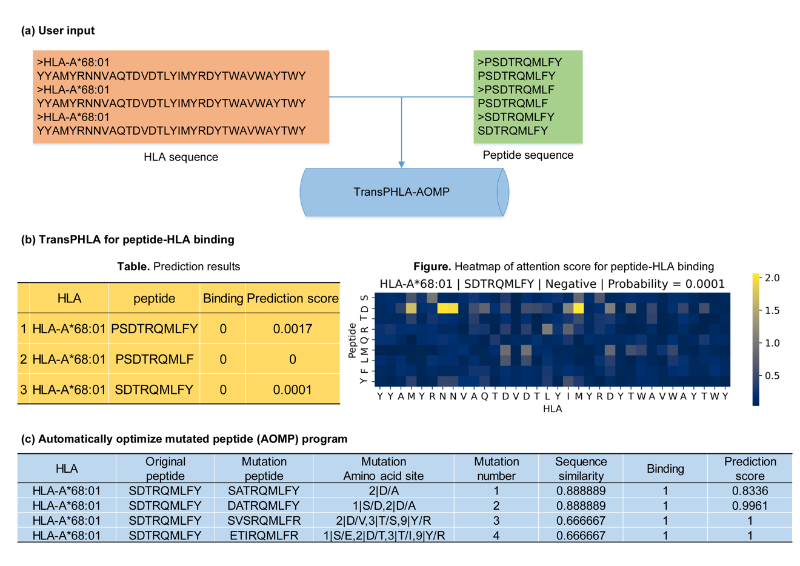
图1.TransMut框架在webserver上的输入和输出
1.数据集
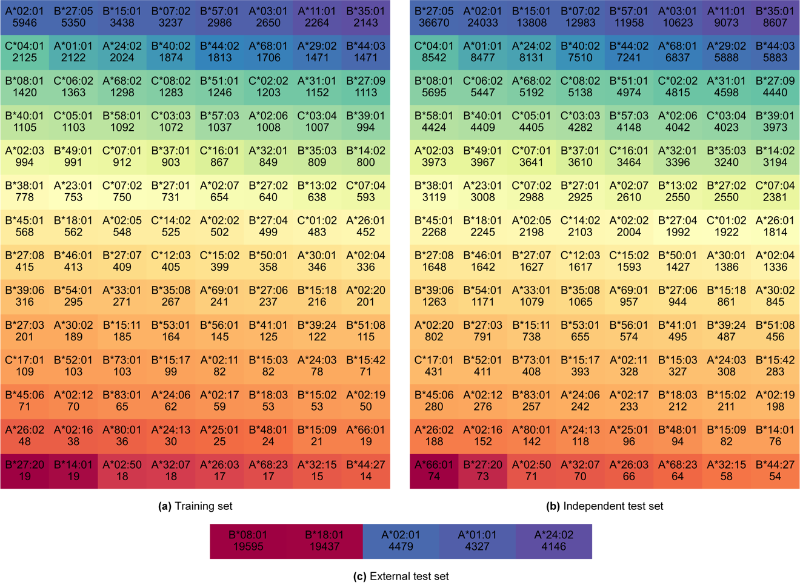
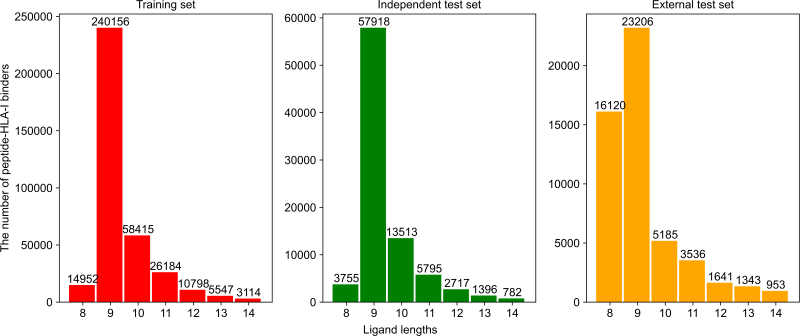
该研究包含112种HLA,肽长度从8到14,共有366种HLA-肽长度组合。详情见图2和图3。
图2.不同数据集中每个HLA相关的可结合的pHLA样本数
图3.不同数据集中肽长度相关的可结合的pHLA样本数
2.TransPHLA模型
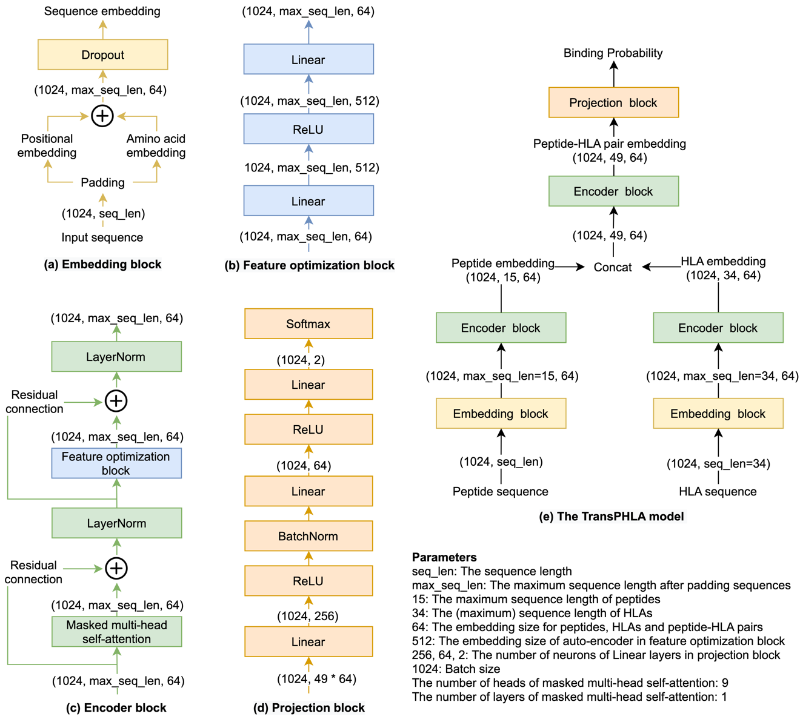
TransPHLA的核心思想是自注意力机制的应用。TransPHLA由以下四个模块组成(图4):首先使用embedding block将positional embedding添加到amino acid embedding中,以生成sequence embedding,然后应用dropout技术来增强鲁棒性。通过embedding block,TransPHLA分别生成肽和HLA的embedding。接下来,这些embedding将分别作为Encoder block的输入。Encoder block包含masked multi-head self-attention mechanism和feature optimization block。Feature optimization block是先上升后下降的全连接层的组合,这个模块使得注意力机制得到的特征表示更好。然后,将输出的肽和HLA的特征表示连接,作为pHLA的embedding。在pHLA的embedding通过encoder block后,使用projection block预测pHLA的结合分数。
图4.TransPHLA模型图
论文链接:https://www.nature.com/articles/s42256-022-00459-7
参考链接:
https://news.sjtu.edu.cn/jdzh/20220325/169006.html
专知便捷查看
便捷下载,请关注专知公众号(点击上方蓝色专知关注)
后台回复“THLA” 就可以获取《【Nature. Mach. Intell. 】基于Transformer的多肽-HLA I类结合预测和疫苗的新生抗原序列设计》专知下载链接