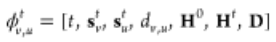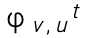JCIM | 深度图生成模型用于设计PROTAC、FBDD、骨架跃迁的linker
本期分享的是Imrie团队在2020年6发表在JCIM上的文章“Deep Generative Models for 3D Linker Design”。该模型既可以用于基于片段连接FBDD、骨架跃迁的linker设计,也可以用于PROTAC分子的linker设计。
合理的化合物设计对于计算方法和药物化学家来说,仍然是一个具有挑战性的问题。深度生成模型已经开始在设计问题上显示出有希望的结果。但是还没有将三维(3D)结构信息的整合进生成模型中。
作者开发了一种新颖的基于图的深度生成模型,它将最先进的机器学习技术与结构知识相结合。DeLinker采用两个片段或部分结构,并设计了一个连接两者的分子。生成过程依赖于蛋白质上下文,利用部分结构之间的相对距离和方向。这种3D信息对于成功的化合物设计至关重要,我们展示了它对生成过程的影响以及丢掉此类信息的局限性。这是第一个将3D结构信息直接纳入设计过程的分子生成模型。
通过大规模计算评估证明了该深度生成方法相对于基于数据库更有效。在与独立测试集上与基于数据库的方法相比,Delinker设计与原始分子具有更高的3D相似性的化合物多出60%,当评估仅局限于具有至少5个原子的linker时,Delinker的性能优于基于数据库方法200%。
一、方法
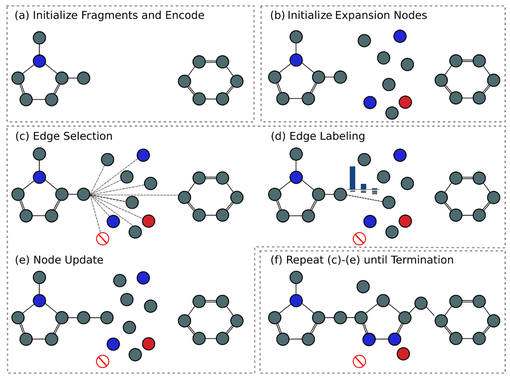
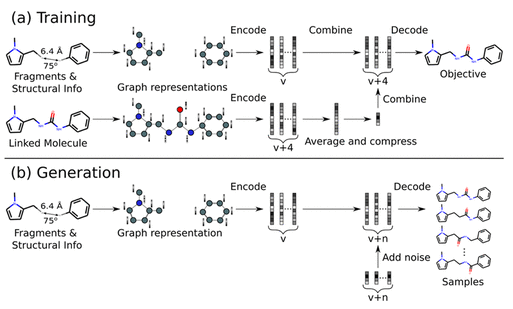
该方法采用两个片段及其相对位置和方向,并生成或替换它们之间的linker。这是通过从可以用部分结构初始化的原子池中以“atom-by-atom"的迭代方式构建新分子来实现的。
生成过程概览:原始片段(a)通过逐个键的迭代扩展(c-e)来产生分子,包括两个片段(f)原子由图中的节点表示,节点的颜色代表不同的原子类型,而键由边表示,单键、双键和三键的边类型不同
在这个框架中,用户可以通过指定应该连接的子结构和它们之间的linker的最大长度来控制生成过程。起始子结构始终保留在生成的分子中,仅改变指定的出口向量。
此外,起始子结构之间的距离和角度的3D结构信息会提供给模型以参与生成过程。分子被编码为14种原子类型,我们的模型通过掩码程序强制执行简单的原子价规则以确保化学有效性。这是唯一直接纳入我们模型的化学知识;生成分子所需的所有其他决策都是通过有监督的训练程序来学习的。
二、生成过程
生成过程以广度优先的方式建立分子键。生成由两个片段或子结构初始化。这些片段或子结构将与提供子结构之间的距离和角度的结构信息连接在一起。片段被转换为图表示,其中原子和键分别由节点和边表示。每个节点都与隐藏状态z和标签L相关联,表示节点的原子类型。该图通过一个编码器网络,一个标准的门控图神经网络(GGNN),并且被更新的节点的隐藏状态合并它们的局部环境中。
接下来,随机初始化一组扩展节点,隐藏状态Z从h维标准正态分布中提取,其中h时隐藏状态的长度。然后根据从学习映射f的softmax输出采样节点的隐藏状态Z和结构信息标记节点的原子类型。这里,f被实现为线性分类器,但可以是将节点的隐藏状态映射到原子类型的任何函数。扩展节点的数量决定了linker的最大长度,是由用户选择的参数。
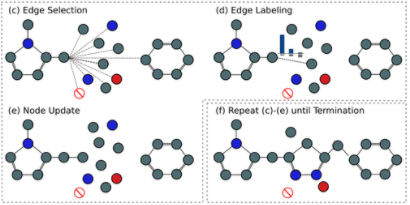
新分子是从上述随机生成的节点中,通过边选择、边标记和节点更新组成的迭代过程构建的。
在每一步,我们都会考虑是否在图中的一个节点v和另一个节点之间添加一条边。v是根据确定性的先进先出队列选择的,该队列使用每个片段的退出向量进行初始化。当一个节点第一次连接到图时,它被添加到队列中。将新边添加到节点v,直到选择到停止节点的边。然后该节点变为“关闭”,没有允许该节点的其他边。
考虑到节点v和图中其他节点之间的所有可能边,受基础化学价约束。使用单层神经网络计算的特征向量评估候选边,节点v和候选节点u之间的边的特征向量由下式给出。
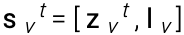
因此,在选择要添加到图中的边时,模型利用(1)关于节点的局部信息,(2)关于未连接片段和当前图状态的全局信息,以及(3)3D结构信息。
一旦选择了节点u,v和u之间的被另一个单层神经网络标记为单键、双键或三键(受化合价约束),输入相同的特征向量
最后更加GGNN更新所有节点的隐藏状态。在每一步,我们丢弃当前隐藏状态并计算新的表示,考虑它们邻域。
对队列中的每个节点上图中的步骤c-e,直到队列为空,此时生成过程终止。在终止时,所有未连接的节点都被删除,最大的连接组件作为生成的分子返回。
三、多模态encoder-decoder设置
我们的目标时学习从未连接片段到连接分子的多个映射的分子(多模态,即生成多个符合的连接的分子)。在训练中,使用了一组配对片段和分子的数据集,并以监督的方式训练我们的模型以构建已知的linkers。虽然在这个数据集中可能有一个独特的分子linker‘和两个片段相连,但实际上,有很多方法可以连接两个片段。因此,给定一对新的片段起点,模型应该能够生成一组不同的输出化合物。
使用显式编码合适的多模式的linker分布的低维隐向量,增强基础encoder-decoder模型,生成映射从F : X → Y转换为F : ( X , z ) → Y,其中X表示起始子结构,Y表示连接的分子,隐编码z从先验分布中提取,选择为标准正态分布。
四、训练过程
在VAE框架下对片段-分子对的集合训练我们的生成模型。对给定的一对片段X和连接分子Y,该模型被训练为从(X,z)重建Y,同时对z和X的编码执行标准正则化约束,z是在训练期间从linker molecule的嵌入导出z。
训练和生成过程说明:(a)提供成对的片段和连接的分子作为输入。该模型经过训练,可以从片段和连接分子的编码组合中复制连接分子。(b)在生成时,该模型仅给出未连接的片段和结构信息,并且能够将片段的编码与随机噪声结合来采样生成各种连接的分子。
为了编码有意义的隐变量z,隐变量z取连接分子Y(linked molecule)的节点的平均,至关重要的是,将z限制为低维向量,以防止模型忽略输入X(两个片段和结构信息)并退化为Y的自动编码器。解码器输入低维的z和没有连接的两个片段输入X的节点编码Zx的拼接。
训练目标函数类似于标准的VAE损失,包括重构损失和KL正则化项。
四、结论
作者开发了一种基于图的深度生成模型,用于集成3D结构信息的片段连接或scaffold hopping,利用设计过程中起始子结构之间的相对距离和方向。与之前的计算片段连接或scaffold hopping的尝试不同,我们的方法不依赖于从中选择连接器的片段数据库,而是根据提供的片段和3D信息设计一个连接器。
在两次大规模评估,作者的生成方法能够学习生成与训练集中存在的约束相匹配的连接器分布,同时能够推广到同时满足2D和3D约束的新型连接器。此外,生成的分子始终与初始片段和原始分子具有高度的3D相似性,在CASF评估中超过数据库基线60%,在将评估限制为具有至少5个原子的接头时提高到200%。
该方法可以应用于片段连接,scaffold hopping和PROTAC设计。