今天给大家介绍中南大学曹东升教授/国防科技大学赵文涛教授、吴城堃教授/浙江大学侯廷军教授团队共同在国际期刊Bioinformatics上发表的分子图片识别的文章《MICER: A Pre-trained Encoder-Decoder Architecture for Molecular Image Captioning》。该文章受编码器-解码器架构的启发,提出了MICER分子图像识别架构,结合迁移学习、注意力机制和几种数据构造策略增强不同数据集的有效性和可塑性;并评估了不同因素对该架构的影响以及数据集错误分析,为后续研究提供方向。该方法在构造的数据集以及基准测试集上较传统的方法取得了显著的提升。
1 摘要 动机 从分子图像中自动识别化学结构为重新发现化合物提供了一个重要途径。传统的基于规则的方法依赖于专家知识,未考虑分子图像的所有风格变化,通常存在识别过程繁琐和泛化能力低的问题。基于深度学习的方法可以整合不同的图像风格并自动学习有价值的特征,这种方法很灵活。但目前研究不足,存在局限性,因此没有得到充分的利用。
结果 MICER是一个基于编码器-解码器的、用于分子图像识别的重构架构,它结合了迁移学习、注意机制和几种策略,以加强不同数据集的有效性和可塑性。评估了立体化学信息、分子复杂性、数据量和预训练的编码器对MICER性能的影响。实验结果表明,分子图像的内在特征和子模型的匹配对该任务的性能有很大影响。这些发现启发了我们设计训练数据集和最终验证模型的编码器。实验结果表明,MICER模型在四个数据集上的表现一直优于最先进的方法。MICER由于其可解释性和迁移能力而更加可靠和可扩展,并为开发全面和准确的自动分子结构识别工具提供了一个实用的框架,以探索未知的化学空间。
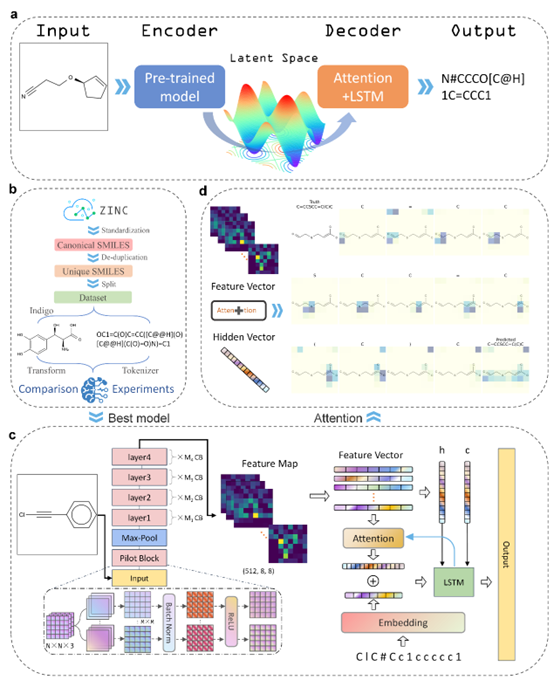
2 方法 2.1 模型架构 MICER采用经典编码器-解码器架构,编码器和解码器分别使用卷积神经网络和循环神经网络(图1a)。在该方法中,编码器使用ResNet网络,解码器使用LSTM网络。在解码器中加入注意力机制,每个时间步模型学习特征图64块(将图像均分割成8*8块)的注意力分数,并指导SMILES字符的生成。
2.2 解码器字典 为了在解码器中表示和操作SMILES字符,作者将所有的字符类型添加到一个字典中。字典包括以下标记。[pad], [sos], [eos], [0], [1], [2], [3], [4], [5], [6], [7], [8], [9], [C], [l], [c], [O], [N], [f], [H [o], [S], [s], [B], [r], [I], [i], [P], [p], [(], [)], [=], [[], [@], []], [#], [/], [-], [+], [\] 和 [%]。根据文章报告,即使按字符进行标记,如[B]和[r],模型也能学到相应的原子组成范式。
2.3 评估指标 * 序列准确度(SA,强约束) * 平均Levenstein距离(ALD,序列相似度) * 平均Tanimoto相似度和Tanimoto@1.0(AMFTS和MFTS @1.0,分子指纹相似度)生成的分子指纹和原始分子指纹之间的相似性用Tanimoto系数来衡量。平均Tanimoto相似度和Tanimoto@1.0(Tanimoto相似度为1.0的百分比)被分别计算。该指标在分子相似度层面上进行分析,可以为下游任务如分子特性预测打下基础。文中,作者选择ECFP4指纹来代表分子,然后根据Tanimoto系数计算分子相似度。
图1 工作流程图。(a) MICER模型的概述。(b) 实验数据预处理。(c) 模型的一般结构。(d) 注意机制的例子。当进行解码时,在每个时间步骤,模型将计算出特征图和隐藏向量的注意力得分,并根据得分生成下一个字符。
3 模型评估 因素对比实验探索了MICER架构在不同因素影响下的性能,并可以在数据和模型层面对最终验证模型的性能进行更有针对性的指导和改进。
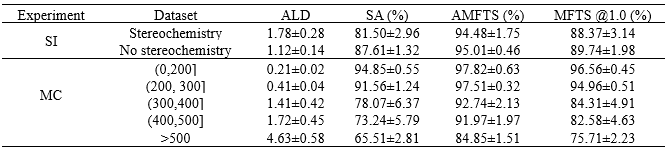
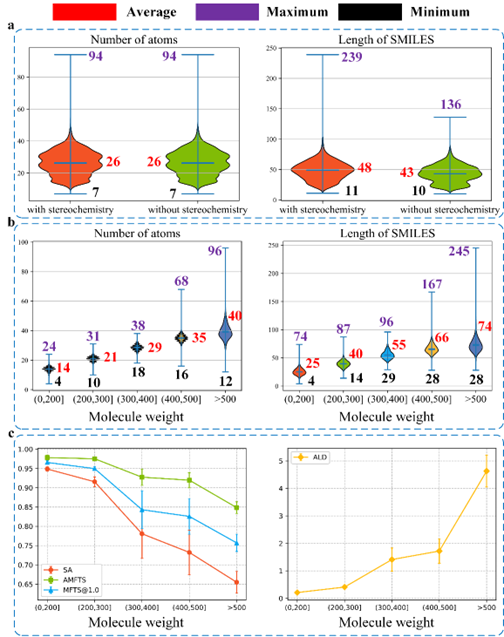
3.1 立体化学信息和分子复杂度 这一部分对比了分子图中有无立体化学信息和不同分子复杂度对模型的影响。分子的复杂性越高,分子图像在准确的分辨率下包含的信息就越多。过于详细的分子图像可能会导致错误的模型;例如,"F "可能会被误认为是一个虚线键。在SI实验中,产生了两个数据集,有立体结构的和没有立体结构的;两个数据集都来自同一个原始SMILES。在MC实验中,五个生成的数据集是根据分子量划分的。如图2b所示,SMILES序列的原子数和长度都显示出与分子复杂性的正相关关系(其中最大的序列长度为245)。
如表1所列,没有立体化学的MICER模型(以下简称'WO')比有立体化学的模型(以下简称'W')具有更好的识别性能,其中SA值增加了约6.1%。第一和第二数据集的ALD分别为1.78和1.12,表明每个预测的SMILES平均有1.78和1.12个错误识别的字符。值得注意的是,虽然两个数据集的SA值相差6.1%,但AMFTS和MFTS @1.0都达到了预期的效果。差异不超过2%,说明与 "W "相比,"W "预测的SMILES字符串中的大多数错误字符都不是原子型错误。换句话说,这些错误识别的字符大部分属于原子型字符以外的字符,比如'@@'、'@'、'['和']'。作者推测,这种错误的识别可能是由于楔形和虚线键造成的干扰,在SMILES中,楔形和虚线键是由'[@@]'和'[@]'表示的。
表1 立体信息和分子复杂度对性能的影响
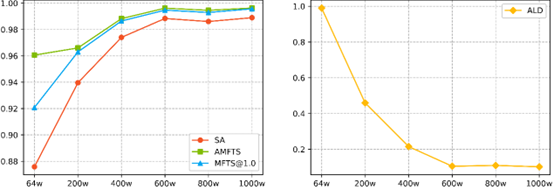
3.2 数据量 根据图3所示的比较结果,训练数据与模型识别性能呈正相关关系。训练数据从64万增加到1000万,SA值提高了约10%(从87.58%到98.91%)。然而,对于超过六百万的训练数据,模型识别性能趋于稳定。在六百万数据量的水平上,模型的准确度达到了98.84%,这已经是一个令人满意的结果。
图2 原子数和SMILES序列长度统计图以及识别性能与分子复杂性的趋势
图3 数据量对识别性能的影响
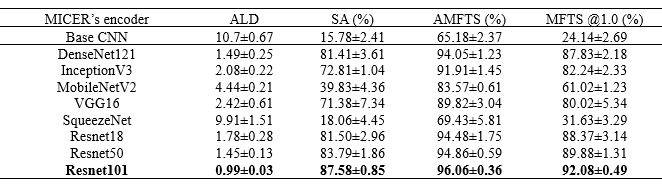
表2预训练编码器对性能的影响
3.3 预训练编码器 Base CNN是一个简单的浅层CNN,包含三个CB。前两个CB包含一个卷积层和一个最大池化层,最后一个包含三个卷积层和一个最大池化层。如表2所示,Base CNN的SA值仅约为15.78%, Base CNN模型未能提取分子图像字幕的固有特征信息。InceptionV3模型有超过40层,其SA值达到了72.81%。这一结果表明,在编码器-解码器训练过程中加入深度CNN,即让一些模型层参与梯度更新,可以更有效地提取图像表征。对于DenseNet121和VGG16的结果也可以得出类似的结论,它们包含121层和16层,SA值分别为81.41%和71.38%。然而,作者希望找出更多有效的模型用于分子图像字幕。如表2所示,ResNet18、ResNet50和ResNet101取得了比InceptionV3更好的结果,表明DECIMER在模型方面仍有改进空间。作者认为ResNet的性能提高可能归功于它的剩余机制,它能在一定程度上解决梯度分散、梯度爆炸和网络退化的问题。此外,作者还探讨了ResNet层数的影响。实验结果表明,ResNet101在三个网络中取得了最好的识别结果,SA值为87.58%。这一结果也为最终的公开审查版本提供了重要的参考价值。此外,从表2也可以看出,SqueezeNet和MobileNetV2的识别精度分别只有18.06%和39.83%。SqueezeNet和MobileNetV2是高度压缩的深度模型,其卷积核很小;因此,它们只能提供有限的感受野和特征提取能力。上述计算结果表明,不同网络的预训练模型对分子识别有明显影响。在未来,将探索更先进的预训练框架来适应编码器或解码器。
3.4 与其他工具的对比 表2与其他工具的对比结果
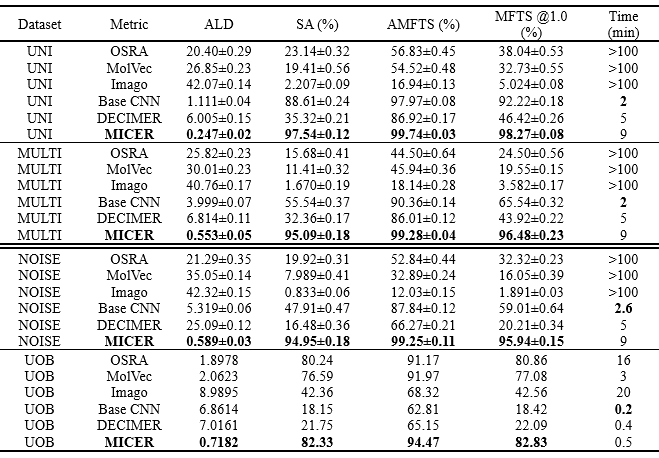
总的来说,MICER在所有测试集上都取得了极具竞争力的结果,在前三个数据集上的SA值超过94%,在UOB数据集上的SA值为82.33%。前三个是基于规则的工具。在这三个基于规则的工具中,OSRA在四个实验中取得了最好的性能,而Imago取得了最差的性能。更具体地说,OSRA在前三个数据集上的SA值低于25%,而Imago的SA值低于3%,这是一个非常不理想的结果。三种基于规则的方法在噪声数据集上的表现很差,这可能是因为它们没有纳入对噪声进行判别的规则。表3还显示,基于规则的工具在多风格数据集上表现出比在噪声数据集上更低的性能,这表明它们对分子图像内容的风格变化比对噪声的变化更敏感。这也表明OSRA对图像噪声的敏感度低于MolVec和Imago,这可能是由于OSAR中包含了一个更稳健的OCR算法。OSRA和MolVec在真实世界的数据集上的表现是可以接受的,这表明这些专家规则比通过化学工具箱生成的规则更符合作者的习惯。此外,三个基于规则的工具在前三个数据集上的识别时间超过了100分钟,因为基于规则的工具的识别步骤繁琐,无法进行批量操作。
值得注意的是,DECIMER的结果并不令人满意,在两个数据集上的SA值都低于40%。此外,据观察,DECIMER生成的SMILES字符串中有很大一部分具有相同的特征,这可以归因于DECIMER使用预训练的模型作为分子图像的特征提取器,而没有微调步骤,这一点是不可或缺的。此外,作为一个基于DL的模型,Base CNN可以通过在与MICER训练数据相等的大量数据上进行训练,在类似的分子图像上取得理想的结果,即使该模型很简单。如表3所示,Base CNN在前三个数据集上的表现优于DECIMER,尤其是uni-style数据集。它的SA值达到了88.61%,验证了关键的编码器学习过程。然而,它在现实世界的UOB数据集上表现不佳,表明这个模型的鲁棒性是不可接受的。
从表3也可以看出,MICER在前三个数据集上取得了超过94%的SA值和超过99%的AMFTS指标,这是一个令人震惊的结果,并且超过了所有其他的工具。此外,对真实世界数据集的比较显示,MICER表现良好,其SA值为82.33%。此外,基于ALD结果,我们比较了MICER预测的SMILES和原始SMILES之间的差异,发现它们之间的差异只有一到两个原子。
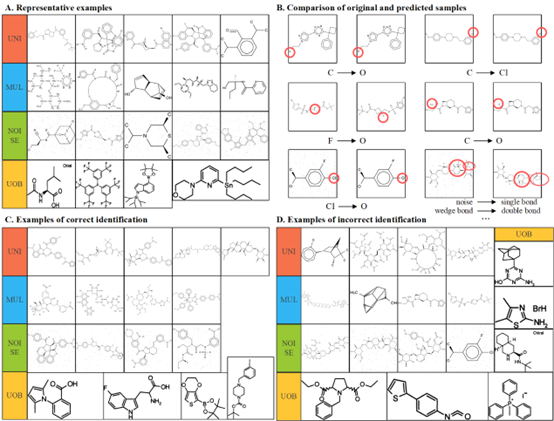
3.5 样例分析 四个数据集的代表性例子如图4a所示。此外,图4c和图4d分别显示了使用MICER架构建立的模型正确和错误的识别样本。从图中可以看出,在前三个数据集中,MICER可以识别SMILES长度大于150的分子图像。预测错误的样本表明,MICER的错误一般是由于存在太多的立体化学键造成的。对于现实世界的数据集,MICER可以识别大多数标准的分子图像,但对于训练期间没有学习过的样本却不能正确识别。
图4 样本分析和错误比较
最后,作者生成了预测分子的图像,并与原始图像进行比较,如图4b所示。从图中可以看出,大部分错误属于单个原子的误分类,其中'C'、'O'和'Cl'原子的误分类率较高,因为这些原子在低分辨率的图像中比较相似。此外,在噪声数据中,作者发现在Cl原子周围有噪声点的情况下,模型会把它们错误地分类为O原子;这也许可以通过减少关注块的面积来改善。带有噪声的复杂分子图像更容易被模型误判;例如,模型会把多个噪声点判断为单键,把楔形键判断为双键,等等。总的来说,未来的研究将关注识别超级原子、R-基团、不规则立体化学键和超复杂原子。
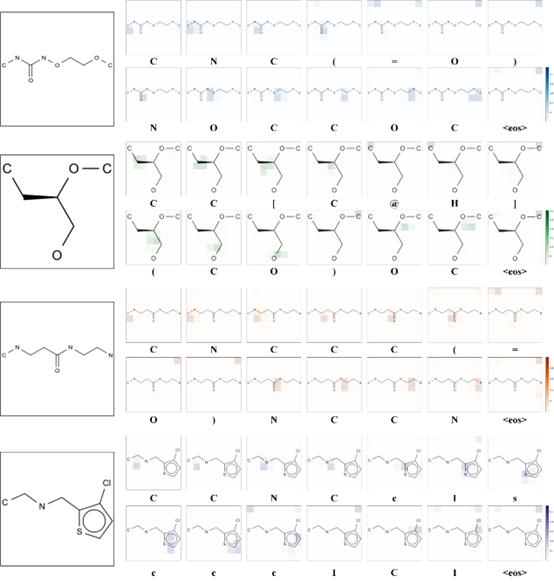
3.6 模型注意力分析 图1d显示了对注意力机制的直观解释。从图中可以看出,在每个时间步骤中,对原始分子图像模型分配不同的权重分数,其中颜色较深的块表示模型对该区域的关注度较高。一些正确预测的例子显示在图5中。左边的图像代表原始分子图,右边的色块作为注意力分数的参考。每个例子的注意权重图由14个子图组成。每个子图上面的特征表示第n个预测步骤的结果。每个子图是一个256×256的加权图像,其中每个加权的图像块是8×8像素。图像块的颜色越深,其注意力得分越高,其中模型几乎能准确识别所有的原子字符。总的来说,注意力机制的引入提高了模型的特征解码能力,获得了更好的可解释性。
图5 注意力权重图示
4 总结 本文中,作者介绍了一种基于编码器-解码器的架构,称为MICER,用于分子图像字幕,具有良好的可塑性。MICER结合了迁移学习和注意力机制。此外,作者还介绍了几种策略,在四个数据集上全面验证了其有效性。作者通过四组因素对比实验,探讨了不同的内在数据特征和模型变化对MICER架构的影响。在这个架构中,一个预先训练好的CNN作为编码器,一个具有注意力机制的RNN作为解码器,它们可以共同学习高维表征,这对于涉及分子图像的许多下游任务来说是很重要的。此外,作者还对注意力权重和代表性样本进行了可视化分析,这为未来的研究提供了直观的见解。未来将继续探索基于MICER架构的解释性和稳健性模型。 参考资料 Jiacai Yi, Chengkun Wu, Xiaochen Zhang, Xinyi Xiao, Yanlong Qiu, Wentao Zhao, Tingjun Hou, Dongsheng Cao, MICER: A Pre-trained Encoder-Decoder Architecture for Molecular Image Captioning, Bioinformatics, 2022;, btac545, https://doi.org/10.1093/bioinformatics/btac545