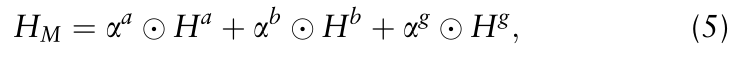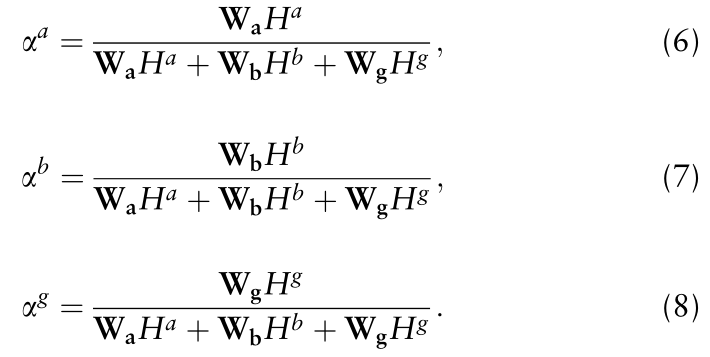作者 | 夏宇航 审核 | 刘 旋今天给大家介绍的是来兰州大学张瑞生教授团队发表在Bioinformatics 2022上的文章"MultiGran-SMILES: multi-granularity SMILES learning for molecular property prediction".原子水平表示是分子的一种常见表示,在一定程度上忽略了分子的子结构或支链信息;而子串水平表示则相反。原子级别和子串级别的表示都可能丢失分子的邻域或空间信息。而聚集分子邻域信息的分子图表示法在表示手性分子或对称结构方面能力较弱。在这篇文章中,作者利用不同粒度表示法的优势同时用于分子性质预测,提出了一种融合模型MultiGran-SMILES,与分子的单一粒度表示相比,作者的方法同时利用了各种粒度表示的优点,并自适应地调整每种表示对分子性质预测的贡献。
1.摘要
分子性质预测是物理、化学和材料科学领域的热点问题。提取有用的分子特征对于分子性质预测至关重要。原子级表示通过原子级标记化,根据SMILES串给出了分子的细节。与原子级别的表示相比,子串级别的表示详细地提供了分子的一些子结构信息或片段。原子级别和子串级别的表示都可能丢失分子的邻域或空间信息。而聚集分子邻域信息的分子图表示法在表示手性分子或对称结构方面能力较弱。图1为分子的三种粒度表示。为了充分利用不同分子表示的优势,本文提出了一种多粒度融合学习模型,该模型同时利用了不同粒度表示的优势,并自适应地调整每种粒度对分子性质预测的贡献。作者的方法在广泛使用的数据集上得到了验证。此外,作者还与单粒度表示(即原子级别、子串级别或分子图)以及分子图和SMILES串的融合方法进行了比较。
2.模型介绍
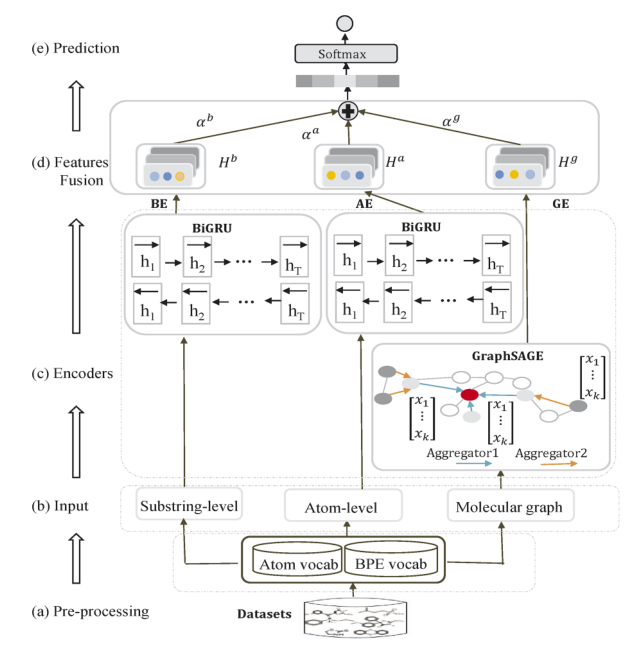
模型由五个组件组成,包括输入数据的预处理(a)、输入层(b)、编码器(c)、特征融合(d)和最后一层预测(e)。将分子序列送入前处理部分。输入层由三种粒度表示(即原子级、子串级和分子图)组成。该模型的编码器由两种类型的子编码器组成。BiGRU编码器利用共享参数对原子级和子级信息进行编码。GraphSAGE用于对分子图进行编码。融合层结合了原子级表示(记为AE)、子串表示(记为BE)和分子图表示(记为GE)的输出,便于同时融合不同粒度的优势。
2.2 Bi-GRU双向编码器
作者使用GRU作为RNN单元,每个循环单元可以通过GRU自适应捕获不同时间尺度的依赖性。重置门和更新门是两种类型的门,和的计算方法如下:
新的隐藏单位ht的计算方法如下:
2.3 分子图编码器
本文使用GraphSAGE作为分子图的编码器,它可以通过采样和聚合节点的邻居嵌入来有效地捕捉图的结构信息。SMILES字符串可以表示为, 被送入GraphSAGE编码器,输出,具有相同的长度。 2.4 多粒度SMILES特征表示融合
作者融合了三种类型的编码器的输出,并自适应地调整每种粒度对分子性质预测的贡献,称为。是来自原子级Ha、子串级Hb和分子图Hg的表示的融合。融合层如图2d所示。融合表示的计算如下:
表示三个隐藏状态的权重,门的大小与H的隐藏态相同。是提出的多粒度分子表示。作者借鉴前人的工作也对样本进行了填充,以统一它们的长度。因此,可以将不同长度的三种不同粒度的表示直接相加,得到用于预测的融合表示。在这项工作中,作者将定义为向量,计算方法如下:
3.实验
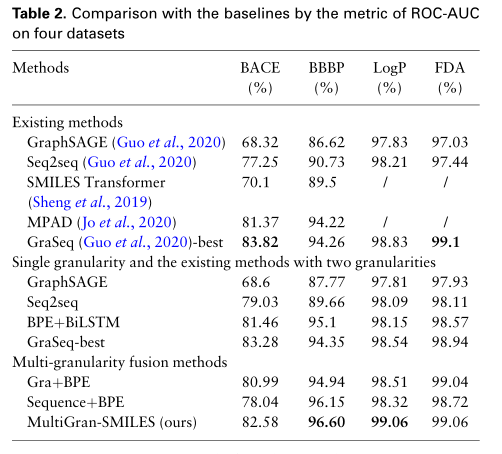
作者广泛使用的MoleculeNet中选择数据集,即BACE、BBBP、HIV、Tox21和ClinTox。另一类数据集如LogP和FDA来自ZINC数据集。 BACE、BBBP、LogP和FDA数据集的ROC-AUC评估指标结果表2所示。可以看出作者的三种粒度融合的表示学习方法优于基准方法。
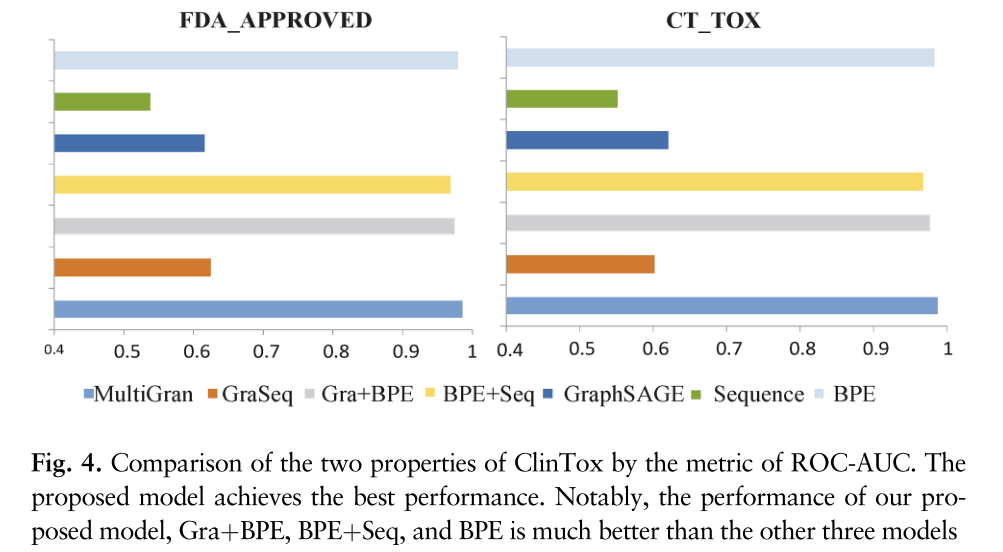
为了进一步验证模型性能,作者将Tox21和ClinTox上的每个任务视为单个属性,然后在每个任务中相互比较ROC-AUC。图3显示了使用不同粒度的不同模型对Tox21上12种属性的ROC-AUC进行比较,作者的模型在12个任务中的6个任务上达到了最先进的性能。图4显示了两种属性在ClinTox上的ROC-AUC比较****
作者还比较了一些有关LogP、FDA和HIV的最先进模型的准确性。实验结果如表3所示,说明了FDA和HIV的准确性。表4显示了在LogP上的比较结果。
上述实验结果表明,通过融合不同分子粒度的表示可以捕获分子特征,对于具有明显官能团或分支的分子,该方法的增益更大。因此,作者的方法能够自适应地调整和选择分子属性预测的有利特征,并对不同的数据集具有较好的泛化能力。 4.总结
本文通过充分利用各种粒度表示法的优势,并自适应地调整每种粒度表示法的贡献度,从而为分子性质预测提供了一种综合的方法,大大缓解了单一粒度表示法的不足。与现有的融合方法相比,该方法通过引入子串级别的表示或分子片段,具有更好的泛化能力。 参考文献****