WIREs CMS | 基于深度学习的药物重定位:方法、数据库和应用
作者|潘小琴
近期,来自湖南大学曾湘祥教授的科研团队,与弗雷德里克国家癌症研究所Ruth Nussinov教授、伊利诺伊大学芝加哥分校Philip Yu教授、里海大学Lifang He教授、中南大学湘雅医学院曹东升教授、湘潭大学林轩博士以及凯斯西储大学和克利夫兰诊所的学者合作,以“Deep learning for drug repurposing: Methods, databases, and application”为题,在Wires computational molecular science(IF: 25.113)上发表了利用深度学习方法和工具进行药物重定位主题的综述文章。

新药研发既费时又费钱。据东方研究小组称开发一种候选药物通常需花费约10-15年和0.8-15亿美元,而开发新分子实体的成功率仅为2.0%。从已批准或成熟的临床药物中有效识别新适应症在药物发现中起着至关重要的作用,这样的过程也称为药物重定位。随着药物发现中可用的化学和生物医学数据量稳步增加,如何有效地探索大规模领域数据中的有价值信息成为药物重定位的关键。另一方面,深度学习在广泛的复杂任务中取得了显着的成功,包括自然语言处理、语音识别和计算机视觉等,深度学习方法也开始应用于药物重定位。与传统的机器学习技术不同,深度学习的优势在于它能够学习输入特征和输出决策之间的复杂关系,特别是深度学习方法可从其输入数据中自动学习多层次的表示,而无需额外的信息输入。尽管它们在药物再利用方面的应用仍处于起步阶段,但它们已显示出巨大的潜力。
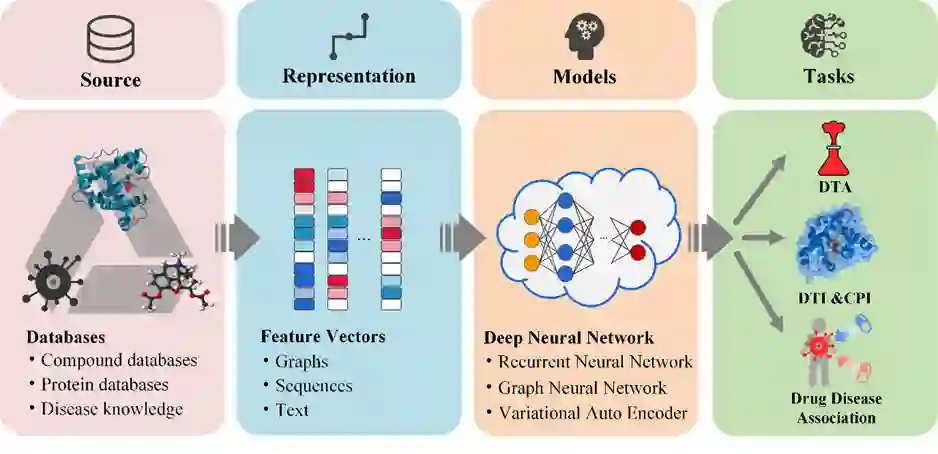
这篇综述首先概述了常用于药物重定位的生物信息学和药物基因组学数据库;其次,分别讨论了基于序列和基于图的表示学习方法及最先进的基于深度学习的方法;最后,介绍了药物重定位在抗击新型冠状病毒肺炎中的应用,并总结了其未来的挑战。图1为基于深度学习的药物重定位主流研究过程:通过表示学习提取数据源中的有用信息以获得其特征向量,然后将它们输入到各种深度学习模型中,这些模型可用于预测下游任务,例如药物-靶点结合亲和力预测和化合物-蛋白质相互作用预测。
图1:基于深度学习的药物重定位的当前主流研究过程
表示学习方法
学习数据表示,可在构建分类器或其他预测器时更容易提取有用信息。用于药物重定位的表示学习主要可分为基于序列和基于图的表示学习方法。药物表示方法包括三类,如图2所示。一维表示即SMILES字符串的One-hot表示;二维表示为分子图,其中每个子结构与预定义的位向量相关联;第三种方法采用图神经网络(GNN)将分子图转移到向量中,其中原子和键分别由节点和边表示。如图3的靶点蛋白质表示方法所示,包括氨基酸序列的One-hot表示、接触图的二维(2D)表示以及图卷积神经网络用于学习三维(3D)蛋白质图的表示,其中节点代表各种组成的非氢原子。
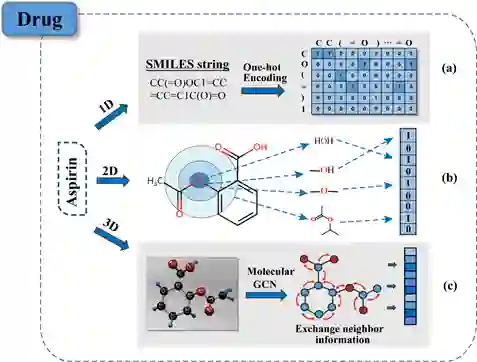
图2:药物表示方法
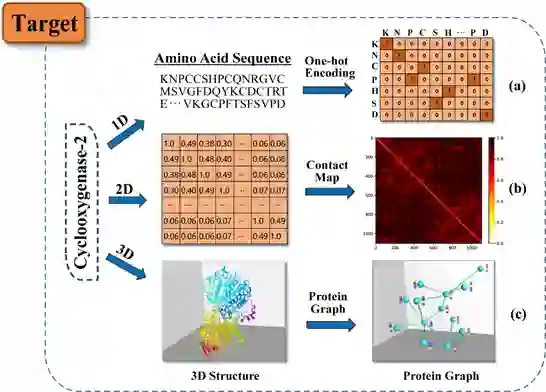
图3:靶点蛋白质表示方法
2
基于深度学习的药物重定位模型
药物重定位工具通常表示预测未知的药物-靶点或药物-疾病之间的相互作用,可将其分类为“以靶点为中心”和“以疾病为中心”的计算方法。靶向捕捞策略对药物的化学结构进行编码以筛选靶向蛋白质,从而提供详细的多药理学解释。然而,单一的预测目标并不能完全描述疾病的特征。因此,有效识别药物和疾病之间的关联对于理解潜在的生物学机制至关重要。每种方法都提出了信息学的独特挑战,该综述分别总结了近年来基于药物和基于靶点的药物重定位计算模型。图4介绍了多种深度学习模型的架构,包括:(a)图卷积神经网络(GCN);(b)网络嵌入(NE)或知识图谱嵌入模型(KGE);(c)自动编码器(AE);(d)全连接深度神经网络(DNN);(e)循环神经网络(RNN)以及(f)卷积神经网络(CNN)。
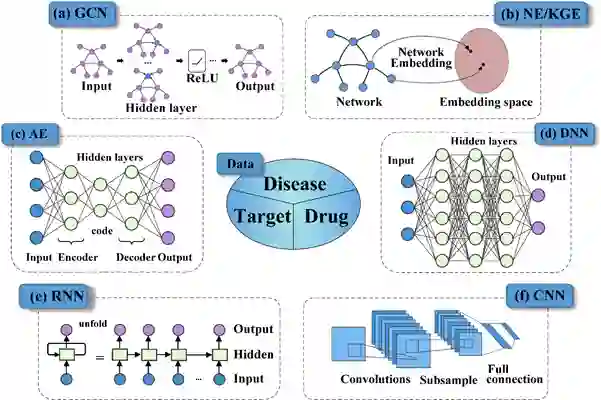
图4:深度学习模型的架构
3
药物重定位应用
药物重定位已被证明是一种有前途的策略,可用于多种人类疾病的药物发现和开发,例如罕见疾病,神经退行性疾病,癌症,传染病等。在这篇综述中,作者以一种由SARS-CoV-2 引起的传染COVID-19为例,讨论了药物重定位策略如何加速治疗开发以应对COVID-19大流行的危机。
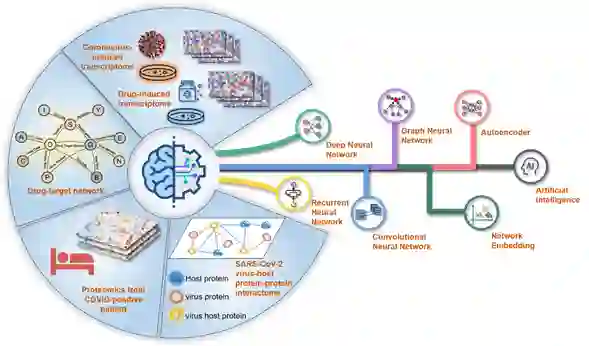
图5:基于深度学习的药物重定位基础设施
4
总结与展望
深度学习已被广泛应用于多个生物医学研究社区,包括药物重定位。与依赖于显式物理方程的物理模型不同,深度学习方法通过设计模式识别算法来映射小分子之间的数学关系,在不需要大量计算资源的情况下能更有效地处理大数据集。然而,深度学习方法的局限性在于模型训练需要大规模、高质量的数据集,以及揭示预测背后的生物学意义的可解释性。虽然传统的机器学习方法可以很好地解决某些领域的特定任务,但随着数据的爆炸式增长和 AlphaFold2的成功落地,有理由相信深度学习将为药物带来里程碑式的发展。
参考资料
Pan X, Lin X, Cao D, Zeng X, Yu PS, He L, et al. Deep learning for drug repurposing: Methods, databases, and applications. WIREs Comput Mol Sci. 2022;e1597.
https://doi.org/10.1002/wcms.1597