——介绍——
分子模拟的粗粒化 (CG) 通过将选定的原子分组为珠子简化了粒子的表示,并大大加速模拟。然而,这种CG过程会导致信息丢失,这使得精确的反向映射,即从CG坐标恢复细粒度 (FG) 坐标,成为一个长期的挑战。来自MIT的Rafael Gomez-Bombarelli团队和唐建团队在2022年的ICML上联合发表了"Generative coarse-graining of molecular conformations"来解决这个问题。 ——背景——
粗粒化分子动力学模拟 (CGMD) 可以通过更简单的组合规则显著加快化学空间的计算发现。对于具有几十万个原子的大分子模拟,CGMD能够达到观测蛋白质折叠等长时间尺度的事件。然而,这样的加速是以丢失FG原子细节为代价的,这对于研究原子级结构的性质和相互作用或在FG尺度上继续模拟是非常重要的。如何从CG坐标准确地恢复到FG坐标仍然是一个具有挑战性的问题。 传统的反向映射有随机投影和基于几何规则的。然而,这些方法通常只能产生较差的初始几何结构,还需要后续的MD来精修结构,从而产生相当大的计算成本,甚至可能导致与原始结构相比有较大的结构偏差。这些方法还需要维护体系特定的片段库,而仅适用于预定义的映射模式,因此其泛化性不强。而目前的几个机器学习模型在反向映射的FG几何结构上都不够理想。 ——方法——
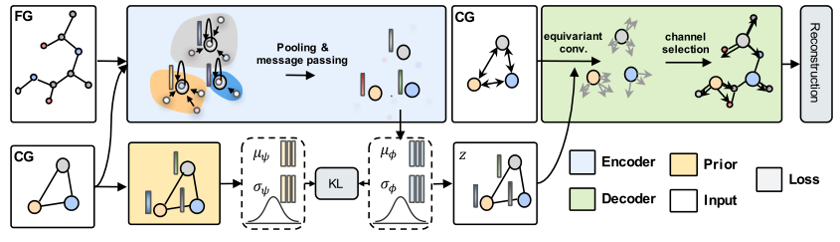
作者提出了一种新模型CGVAE (Coarse-Graining Variational Auto-Encoder, 图1),该模型严格嵌入了反向映射变换的重要概率性质和几何一致性要求,将FG不确定性编码到隐空间中,并通过等变卷积将其解码回FG结构。具体来说,编码器部分负责提取FG和CG的结构特征,经过消息传递和pooling后映射到CG空间,得到CG尺度的隐变量,进一步得到后验分布。另外,仅考虑CG结构,再训练一个先验模型,得到先验分布,并利用此先验分布和后验分布计算KL散度,作为一个损失函数,是一个较典型的条件VAE编码器。编码器方面,作者利用vector neuron对边做卷积,预测坐标的相对位移。为了考虑手性,额外引入pseudoscalars和pseudovectors。最后再经过channel selection,基于CG-FG的映射,从CG节点的隐空间嵌入挑选出对应FG原子的嵌入向量,拼接后形成FG尺度的特征,并额外加入辅助损失函数来保证合理的键长,得到最终的重构损失函数。
图1. CGVAE的总体架构
——实验结果——
作者使用丙氨酸二肽和chignolin的MD轨迹作为数据集,五折交叉验证进行训练和评估。如下图2所示,反向映射后FG结构的质量是可观的。
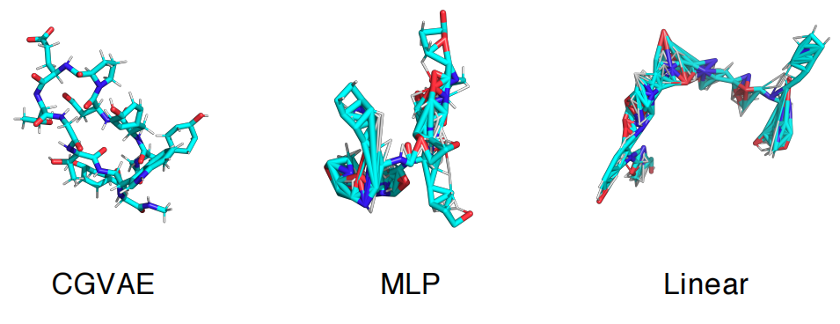
图2. 左图为CGVAE在丙氨酸二肽和chignolin的粗粒化结构和反向映射的全原子结构;右图为不同分辨率(CG珠子的数目)下重构的全原子和GT结构的RMSD比较 作者还将模型与其他几个较为简单的baseline (MLP和Linear) 作了比较,下图2是用于比较的可视化结果。
图3.CGVAE和其他两个baseline的重构CG结构的可视化比较 ——总结——
在这项工作中,作者提出了CGVAE模型,用于将CG结构反向生成FG结构。作者计划在未来将CGVAE这套框架扩展到更复杂的凝聚相系统中,在分子内和分子间水平上进行精确的反向映射。有研究兴趣或者有应用需求的读者可以访问源码: https://github.com/wwang2/CoarseGrainingVAE参考文献:Wujie Wang, et al. Generative coarse-graining of molecular conformations. arXiv:2201.12176.点击左下角的"阅读原文"即可查看原文章。
作者:幻 幻审稿:顾仲晖编辑:黄志贤 GoDesignID:Molecular_Design_Lab( 扫描下方二维码可以订阅哦!) 本文为GoDesign原创编译,如需转载,请在公众号后台留言。