编译 | 董靖鑫 审稿 | 程玉今天给大家介绍的是IBM研究团队发表在arxiv上的一项有关分子生成的研究。作者提出一种可以编码3D蛋白质-配体接触的图生成模型,该模型利用条件VAE从而可以预测在靶标结合口袋内的分子相互作用,从而实现特定活性的分子生成。实验结果表明,与基于配体2D生成方法相比,该方法生成的分子与多巴胺D2受体的结合口袋更契合。该研究展现了蛋白靶标的结构信息如何增强分子生成。
1 介绍 小分子生成模型基于一系列深度学习架构设计的,其通常是要生成具有所需物化性质的分子,以增强化学家的创造力,并扩展传统数据方法或物理方法的规模。大部分模型使用SMILES作为主要的分子表示,因为这样可以从自然语言处理(NLP)技术中借鉴成功的经验,但1维字符串远不能提供小分子的物理直观表示。在现实中,小分子是由存在于三维构象空间中的原子间连接的复杂网络构成。虽然复杂的基于文本的深度学习架构,如Transformer,有时可以从简单的字符串中学习空间关系,但编码分子结合和三维性质表示的架构提供了一种更自然的方法来捕捉深度学习中的化学和物理信息。最近的一些围绕着小分子的二维图表示展开的研究也取得了进步。然而,配体结合最好的理解是在三维受体结构和蛋白配体结合位点内分子间接触的背景下。最近的一些生成工作已经融入了来自靶标结合位点的信息(基因组或蛋白质序列)以及更具体结构数据(3D体素,空间形状或分子碎片表示)。
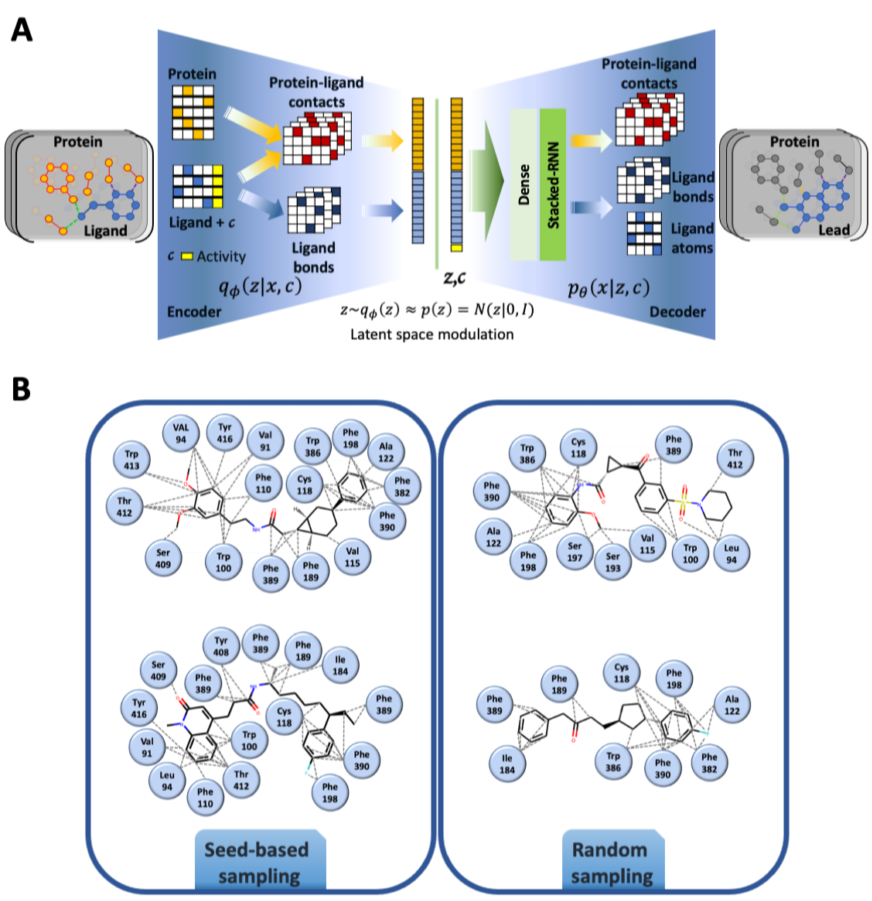
2 模型 本文的模型将三维蛋白质-配体复合物显式编码为分子图,扩展成一个关系图结构,而不同于以前仅配体的图生成模型(图1A)。以分子和接触图为输入,该模型建立在一个由图卷积编码器和RNN辅助图解码器组成的**条件变分自编码器架构(cVAE)**上。
通过优化三个目标函数来训练模型: (1)配体分子图和蛋白质接触图的重建损失, (2)高斯先验和编码后验之间的正则化KL散度, (3)捕获生成的蛋白质-配体相互作用的精确度的JS散度。
图1 gen3D模型架构。A.3D蛋白-配体相互作用网络;B.基于种子和随机采样生成的预测蛋白质-配体相互作用(虚线)的分子示例。
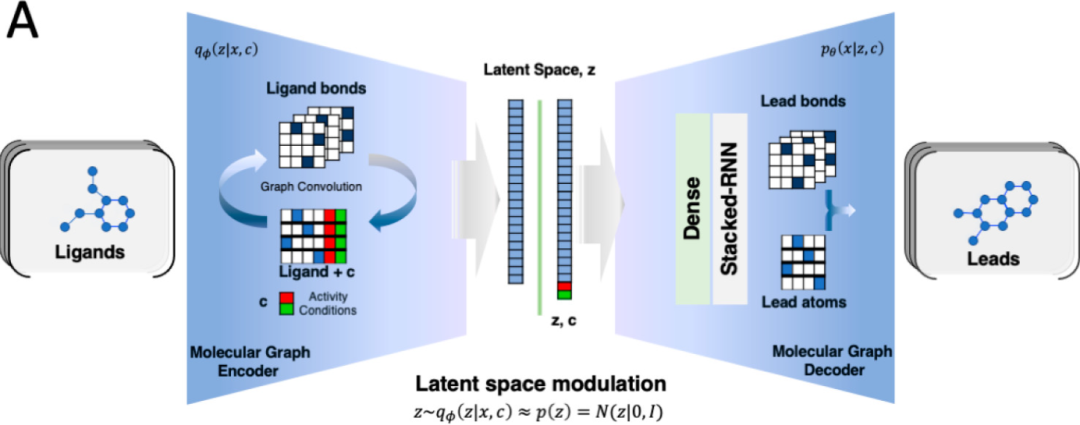
为了确定该3D方法的优势,作者在一个共同的靶标(多巴胺D2受体)、共同的训练集和测试集上训练gen3D(本工作)和gen2D,并尽可能在一个共同的图cVAE架构上训练。
图2 gen2D模型架构。 (https://doi.org/10.1021/acs.jcim.1c01545)
3 实验 3.1 实验设置
生成策略 (1)随机生成,通过在潜在空间直接随机抽样;(2)基于种子的生成,通过在编码种子的潜在空间邻域随机抽样。
对接 对gen3D和gen2D按照上述两种生成策略生成的分子进行了大规模的对接模拟,当存在立体中心时,枚举所有可能的对映体。
3.2 对接结果
图3 来自种子和随机抽样的gen3D和gen2D分子的对接分数,包括前3位的平均对接分数(左)和活性条件和非活性条件分子的平均差异(右)。
基于种子的采样策略在gen3D产生的top位对接得分比gen2D平均低近一个整体能量单位(~0.8千卡/摩尔),第二位和第三位保持相同的趋势(图3)。活性条件下也产生了gen3D比gen2D对接更一致的结果。**实验表明,用3D蛋白质接触训练提高了生成的分子和预期靶标口袋之间的互补性,**这是通过对接分数衡量的。
随机抽样策略在gen3D和gen2D之间产生了统计上差不多的对接分数,无论是绝对的,还是活性和非活性条件分子之间。这种在随机抽样情况下缺乏三维优势可能是由于三维潜在空间的复杂性增加,需要更高的维数来编码蛋白质-配体接触。
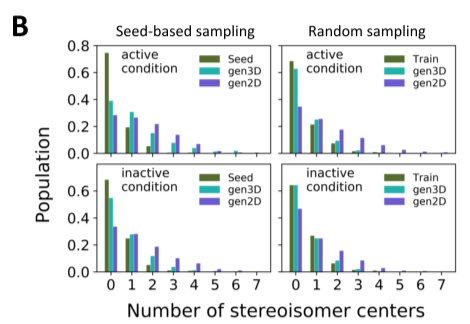
3.3 立体化学性评估
图4 从gen3D和gen2D生成的分子的立体异构中心计数分布。
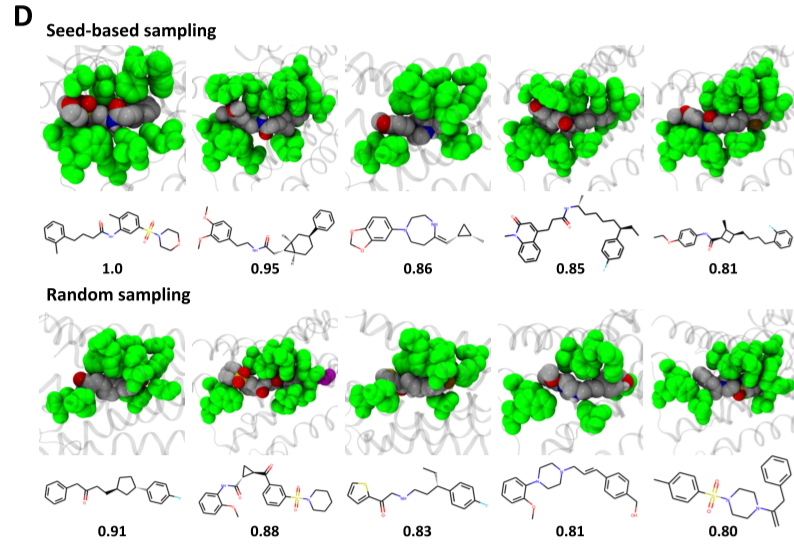
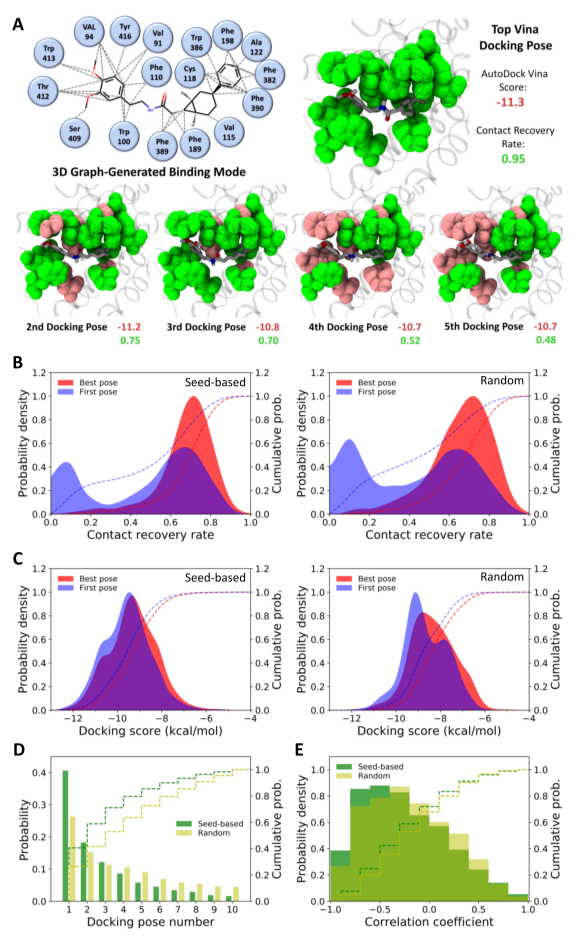
图5 由Gen3D模型生成的选定分子的对接结合方式。生成的分子以经典元素颜色显示,生成预测的接触残基以绿色显示。
gen3D模型在立体化学方面也产生了更理想的结果。gen3D在生成策略和活性条件下产生与参考种子和训练集更一致的立体中心计数;相比之下,gen2D方法倾向于更高的立体中心计数,使合成和构象选择复杂化(图4)。有趣的是,gen3D方法可以隐式学习匹配参考数据中的立体中心计数,并表明通过训练蛋白质-配体复合物施加的约束限制了生成的分子的构型多样性,使其形状与靶标的结合口袋互补,从而提高对接分数(图3和图5)。
3.4 蛋白质配体接触与对接结合位置的匹配度评估
图6 对接结合位置接触的匹配率。
超过90%的基于种子采样生成的分子和85%的随机抽样生成的分子的最佳结合位置能与至少50%的预测接触匹配。因此,由gen3D预测的几乎所有结合模式都能以合理的匹配率由对接程序进行采样。作者还发现对接分数和接触匹配率有着负相关的关系。gen3D模型可以成功地学习生成的配体对特定结合口袋的适应性,这是通过对接模拟获得。
4 总结 本文的方法预测了生成分子的假定结合模式,捕捉了配体结合位点的结构信息。实验结果表明,训练生成模型明确的三维蛋白质配体复合物可以使得小分子与靶标的结合口袋更契合。这些3D生成方法已经开始了解蛋白质-配体结合的底层物理原理,这是加速基于结构的分子设计药物发现的重要步骤。
参考资料 Kang S, Weber J K, Morrone J A, et al. In-Pocket 3D Graphs Enhance Ligand-Target Compatibility in Generative Small-Molecule Creation[J].