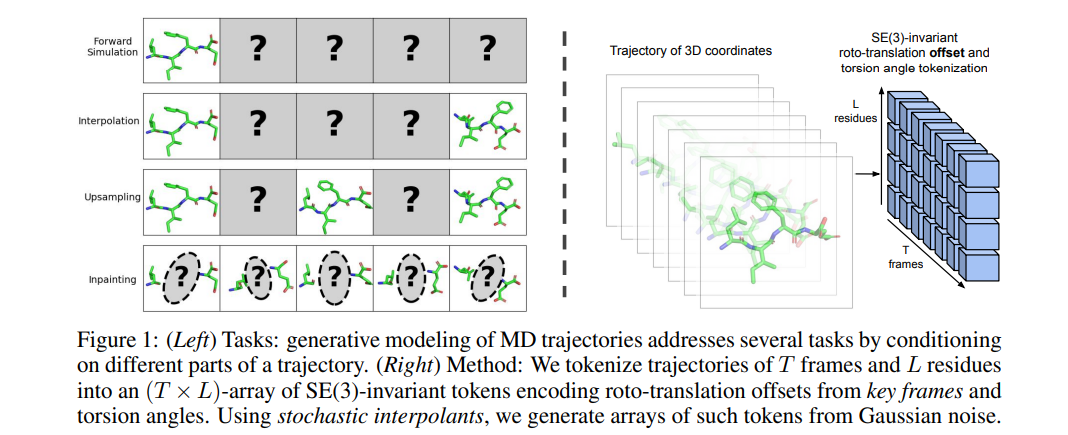
分子动力学(MD)是一种研究微观现象的强大技术,但其高昂的计算成本引发了对基于深度学习的替代模型的广泛兴趣。我们提出了分子轨迹生成建模这一范式,用于从数据中学习灵活的多任务MD替代模型。通过对轨迹中适当选择的帧进行条件化,我们展示了这些生成模型可以适应多种任务,如前向模拟、转移路径采样和轨迹上采样。通过对分子系统的一部分进行条件化并补全其余部分,我们还展示了朝着动态条件下的分子设计迈出的第一步。我们在四肽模拟上验证了这些能力的完整集合,并展示了我们的模型能够生成合理的蛋白质单体集合。总之,我们的工作展示了生成建模如何从MD数据中解锁价值,应用于各种现有方法甚至MD本身难以解决的下游任务。代码已发布在 https://github.com/bjing2016/mdgen。
成为VIP会员查看完整内容
相关内容
Arxiv
42+阅读 · 2023年4月19日
Arxiv
223+阅读 · 2023年4月7日
Arxiv
86+阅读 · 2023年4月4日
Arxiv
152+阅读 · 2023年3月29日