编译 | 程昭龙
审稿 | 林荣鑫,王静本文介绍由浙江大学基础医学院的郭国骥、韩晓平和良渚实验室的王晶晶共同通讯发表在 Nature Genetics 的研究成果:目前研究人员在生成和分析基因组方面做了大量努力,但大多数物种仍缺乏预测基因调控和细胞命运决定的遗传模型。在该研究中,作者利用自主构建的高通量单细胞测序平台Microwell-seq绘制了斑马鱼、果蝇和蚯蚓的全身单细胞转录组图谱,并探究了八种代表性的后生动物细胞类型的跨物种可比性,揭示了脊椎动物细胞类型保守的调控程序。作者开发了一种基于深度学习的模型Nvwa,用于在单细胞分辨率下预测基因表达和识别调控序列。作者还系统地比较了细胞类型特异性转录因子,以揭示脊椎动物和无脊椎动物细胞类型的保守遗传调控。该工作有助于为研究不同生物系统的调控语法提供宝贵的资源和新的策略。
简介
单细胞是生命的基本单位。高通量单细胞RNA测序(scRNA-seq)增强了研究人员识别细胞类型的能力。随着scRNA-seq技术的发展,scRNA-seq分析已被用于绘制各种物种的全生物体细胞图谱,包括人类、斑马鱼、果蝇、小鼠、线虫和涡虫。细胞类型是多细胞生命的基本组成部分,由转录因子(TF)等核心调控因子控制。最近,细胞类型被提出作为具有准独立进化变化潜力的“进化单位”。具有共同谱系祖先的细胞类型共享核心调控TF,其可能在物种进化过程中发生分化。单细胞图谱为系统比较不同物种的细胞类型和调节因子提供了前所未有的机会。
虽然TF的表达可以用scRNA-seq来测量,但目前尚不清楚基因组是如何在细胞图谱背后编码不同的时空遗传程序的。深度神经网络是建模高维数据中复杂关系的强大方法,有助于学习在特定条件下从基因组序列到基因表达的映射。目前已经开发了几种模型来预测DNA序列中的基因表达或染色质谱,如Xpresso、DeepSEA、Basset、 Enformer和AI-TAC。这些深度学习模型在识别复杂序列模式方面显示出了强大的能力。然而,此类模型尚未应用于多物种的综合图谱,并且细胞图谱水平的深度神经网络有可能识别出跨生物共享的新细胞类型特异性调控。
在该研究中作者构建了斑马鱼、果蝇和蚯蚓的全身单细胞图谱。并收集了八种代表性的后生动物图谱,研究了细胞类型和TF的跨物种相似性。然后,作者开发了一种基于深度学习的模型Nvwa,以从单个细胞的DNA序列预测基因表达。最后,作者还解释了细胞类型特异性的序列规则,并表征了跨物种细胞类型的保守调控程序。
结果
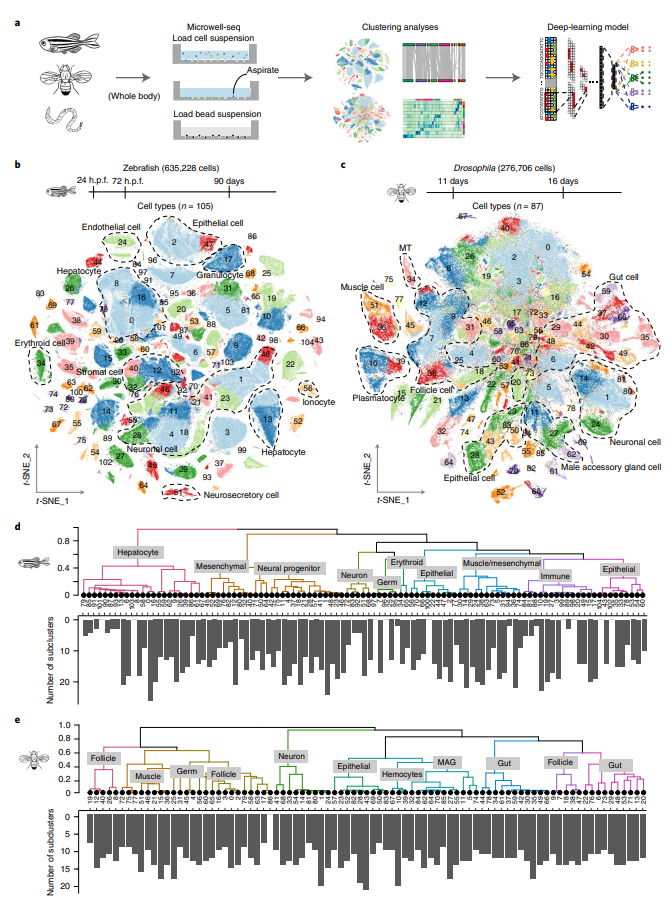
构建斑马鱼、果蝇和蚯蚓的单细胞图谱 之前,作者使用Microwell-seq构建了人类和小鼠的全生物体细胞图谱。在这项研究中,作者使用可以消除组织特异性批次效应的scRNA-seq策略构建了斑马鱼、果蝇和蚯蚓的全身细胞图谱(图1a)。其中,斑马鱼图谱收集了635,228个单细胞数据,果蝇图谱涵盖了276,706个单细胞数据,蚯蚓图谱包含了95,020个单细胞数据。
通过对图谱数据进行无监督聚类,发现了105种主要的斑马鱼细胞类型(图1b)、87种主要的果蝇细胞类型(图1c)和62种主要的蚯蚓细胞类型,它们具有不同的基因表达程序。作者还根据典型细胞类型特异性标记的标准化表达水平对每个细胞类型进行注释。总共105种斑马鱼细胞被分为11个主要细胞谱系:内皮细胞、上皮细胞、红系细胞、生殖细胞、肝细胞、免疫细胞、肌肉细胞、神经元细胞、分泌细胞、基质细胞和其他细胞。作者还对105种主要细胞类型中的每一种进行了子聚类分析,并在层次结构中识别出1285个细胞类型子聚类(图1d)。
果蝇的细胞图谱中,87种细胞类型被分为12个主要的细胞谱系:上皮细胞、神经元细胞、血细胞、卵泡、肠细胞、生殖细胞、雄性副腺、马氏小管(MT)、肌肉细胞、增殖细胞、脂肪体和其他细胞。同时,作者使用MetaNeighbor将构建的单细胞图谱与一个平行的蝇细胞图谱项目进行了比较。在87种果蝇细胞类型中,约93.1%与组织特异性注释一致。最后,对87种主要细胞类型中的每一种进行子聚类分析,在层次结构中共识别出1085个子聚类(图1e)。
图1 使用Microwell-seq构建斑马鱼和果蝇细胞图谱
对于蚯蚓细胞图谱,62种细胞类型被分为8个主要的细胞谱系:消化腺细胞、上皮细胞、神经元细胞、体腔细胞、肌肉细胞、红细胞、生殖细胞和其他细胞。最后,子聚类分析在蚯蚓层次结构中共识别出462个子聚类,这些子聚类在功能上是有意义的。
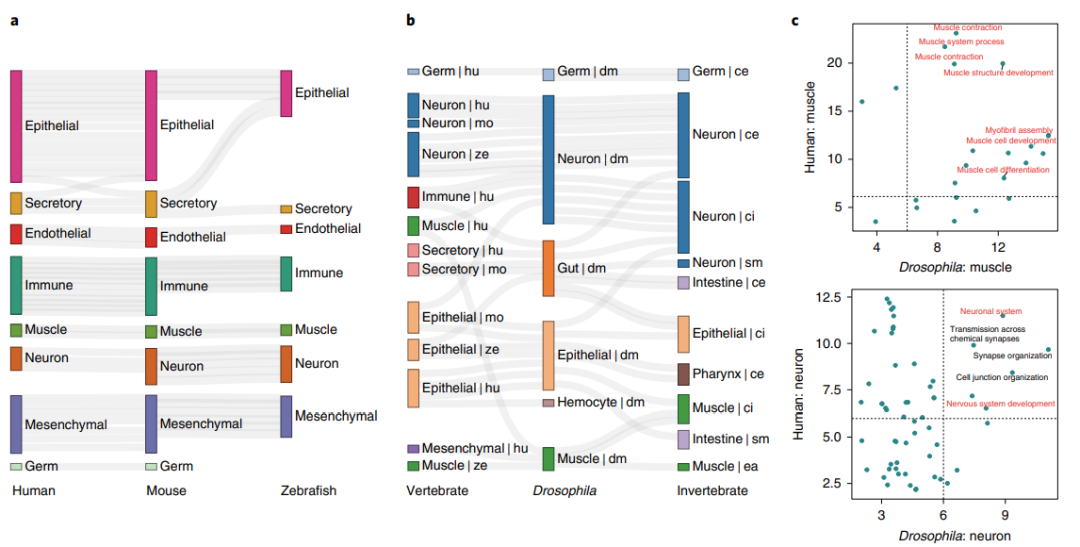
细胞图谱中的跨物种比较 利用流式细胞术和群体分析在生物体水平上研究和建模基因调控模式一直是一个挑战。通过统一的单细胞信使RNA测序(mRNA-seq)平台,作者构建的细胞图谱数据资源为研究跨物种细胞分类的遗传调控提供了前所未有的机会。因此,作者旨在分析细胞类型特异性遗传调控网络,并通过数据集成和机器学习来评估跨物种遗传调控的保守性。为了获得高质量的细胞,作者设置了一个更高的截止值,以生成一个数据集,其中斑马鱼和果蝇平均每个细胞大约有1000个基因,蚯蚓平均每个细胞大约有400个基因。作者总共从八种物种中获得了480种细胞类型,涵盖了主要的细胞谱系,包括上皮细胞、免疫细胞、神经元细胞、基质细胞、肌肉细胞、分泌细胞、红系细胞、生殖细胞、内皮细胞和增殖细胞谱系。然后使用伪细胞算法制作伪体细胞计数矩阵或基于马尔可夫亲和力的细胞图插补 (MAGIC),以插补缺失的基因表达。
为了检验细胞类型的跨物种相似性,对八个转录组数据集进行了成对SAMap分析。作者比较了MAGIC、单细胞和伪细胞三种数据集的跨物种比较结果,结果发现脊椎动物的细胞类型是保守的。基于MAGIC数据集,85.9%同源细胞类型对可以基于单细胞和伪细胞数据集重新识别。为了降低结果的假阳性率,作者设置了严格的阈值来构建跨物种图谱。经分析可知脊椎动物的细胞类型是保守的,特别是免疫细胞、基质细胞、神经元细胞、上皮细胞、内皮细胞和生殖细胞(图2a)。在果蝇的跨物种图谱中,作者还发现几乎来自同一细胞谱系的所有细胞类型都显示出很强的联系(图2b)。为了进一步验证跨物种图谱的结果,作者对肌肉和神经元中同源细胞类型之间的富集基因对进行了功能富集分析(图2c),作者发现富集的基因对具有一致功能,这与之前的研究结果是一致的。
图2 八个物种的跨物种分析
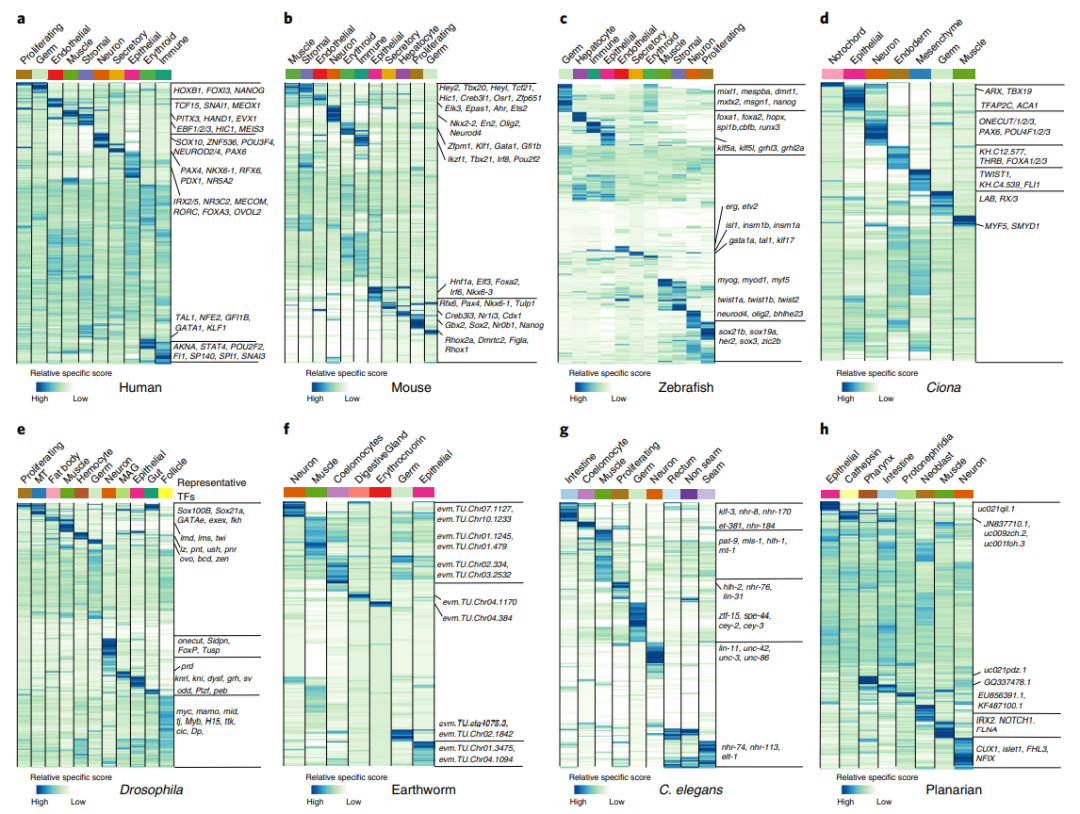
为了评估脊椎动物和无脊椎动物在调控水平上的调控保守性和细胞类型差异,作者计算了每个物种的TF特异性得分(图3a-h)。总的来说,作者在八个物种中共鉴定出2342个细胞谱系特异性TF。基于八个物种间同源基因的转换,可以观察到同源TF中更多保守特征。同源TF分别覆盖了人类、小鼠和斑马鱼所有细胞类型特异性TF的91.42%(70个中的64个)、98.75%(80个中的79个)和75%(104个中的78个)。总之,作者的研究为保守遗传调控基因的跨物种筛选提供了保守遗传调控的详细信息。
图3 利用scRNA-seq数据比较物种内部和物种间的调节TF
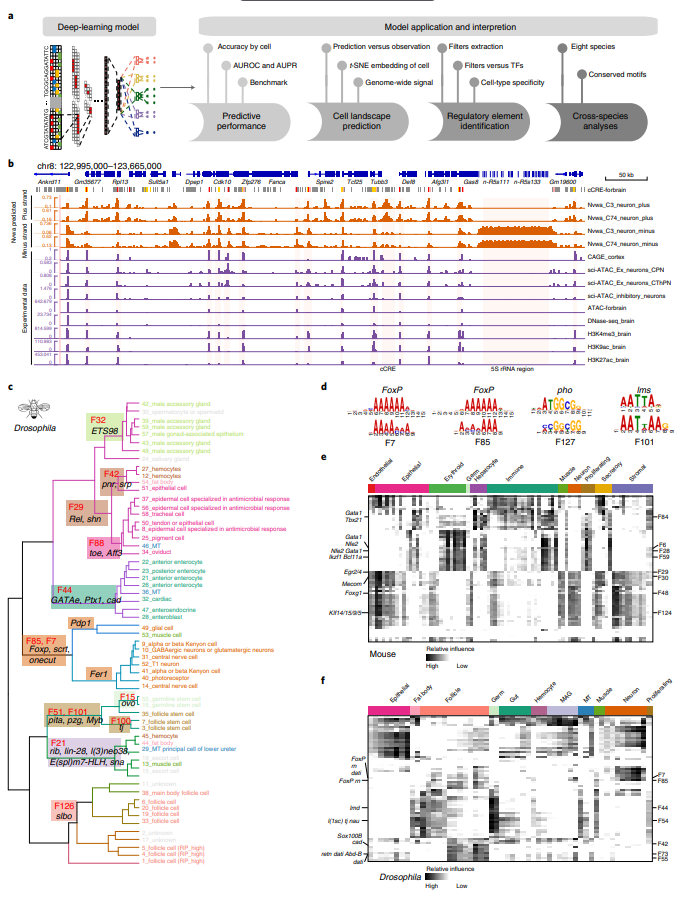
Nvwa根据DNA序列预测基因表达 TF作为调控网络中的重要功能节点,可以识别特定的DNA序列来控制染色质状态和转录。然而,确定DNA序列元件与细胞类型特异性基因调控相关的分子机制仍然具有挑战性。为了更好地理解基因组中编码的调控元件,作者开发了一种基于深度学习的模型Nvwa。训练Nvwa模型以从单热调控序列的输入中预测单个细胞各基因的表达。对预测的细胞图谱进行评估,以验证模型的性能。此后,将学习的序列规则以调控元件的形式进行解释,如序列基序及其预测影响。Nvwa配备了单细胞分辨率,可以进一步识别特定细胞类型与深度学习衍生序列基序之间的关联(图4a)。综上所述,Nvwa可以仅在单细胞水平上利用基因调控序列预测基因表达并识别特定于细胞类型的候选调控因子。
作者首先独立训练了八个物种的Nvwa模型,并评估了Nvwa能否正确预测单细胞基因表达。Nvwa表达预测的准确度是通过检测数据中受试者操作特征曲线(AUROC)下的平均面积和精确召回曲线(AUPR)下的面积来评估的。Nvwa稳健地预测了八个物种的基因表达,其总体AUROC为0.78,AUPR为0.59。通过比较不同细胞类型的性能,表达预测正确性最高的总是生殖系的细胞。Nvwa在预测单细胞基因表达方面进行了优化,在人类和果蝇数据集中优于Basset、DeepSEA、Beluga和Basenji等标准架构。此外,通过集成相关物种的序列进行多基因组训练,可以进一步提高Nvwa模型的准确性。Nvwa模型预测再现了细胞之间的关系,包括细胞类型的相似性和多样性,预测结果与在同一细胞类型中观察到的表达更为相似。细胞类型特异性进一步通过t分布随机邻居嵌入(t-SNE)和预测表达位点在保留基因上的调整互信息(AMI)评分得到证实。总的来说,这些评价证实了Nvwa可以从DNA序列中正确预测单细胞水平的基因表达。
Nvwa可以进一步扩展到扫描全基因组转录活性信号,尽管只训练了基因调控序列(平均约占基因组的13%)。Nvwa模型沿着整个染色体扫描序列,通过识别调控DNA序列来预测信号。通过检查Nvwa全基因组预测,作者观察到它们与实验测量的功能基因组数据相关。此外,通过可视化基因组浏览器轨迹,可观察到Nvwa预测与多种细胞类型和物种中实验定义的信号之间的一致性(图4b)。总的来说,实验分析从外部验证了Nvwa预测性能的鲁棒性。在其应用中,Nvwa模型可以作为在硅片中进行功能基因组研究的辅助工具。
图4 深度学习模型框架的应用和解释
Nvwa确定特定细胞类型的调控程序 为了理解为什么Nvwa可以正确预测单细胞基因表达,作者检查了学习模型的过滤器,其代表了对相应细胞类型重要的特定序列基序。正如预期的那样,在TSS周围的窗口内系统地移动输入序列表明,近端启动子区域(±1 kbp)的信息量最大。然后,作者基于特征映射和TF-MoDISco方法从每个第一层卷积滤波器中提取深度学习的基序,并发现这两种方法给出了一致的结果。作者还计算了序列基序的细胞类型特异性,并使用影响评分进行量化。结果表明,与不同TF相关的过滤器也参与了细胞类型的识别和细胞活性(图4c)。这些结果启发作者进一步分析模型过滤器及其与细胞类型特异性基序和TF的关系。
Nvwa衍生的序列基序可以分配到已知的TF结合位点(TFBS)。作者还观察到,带注释的滤波器与已知的TFBS高度相似(图4d)。在交叉验证分析中,大多数注释滤波器具有较高的再现性和信息含量,这表明Nvwa解释的鲁棒性。一些影响分数较高的未注释过滤器可能捕获了较短的序列模式。
除了生物学注释,作者还检查了Nvwa序列基序的细胞类型特异性。对于小鼠和果蝇,50%-80%的细胞类型特异性Nvwa基序通过相应的单细胞ATAC-seq数据被重新识别。作者还发现,细胞类型特异性过滤器与相应TF的已知作用一致(图4e,f)。作者还在果蝇中鉴定了过滤调节子,这证实了由相同过滤器调控的靶基因具有相似的细胞谱系特异性表达模式(图5a,b)。总之,这些结果表明,Nvwa可以利用与特定细胞类型相关的TF的深度学习衍生基序,使得能够直接从序列中筛选细胞类型特异性调控因子。
图5 Nvwa 确定特定细胞类型的调控程序
Nvwa基序的跨物种比较 为了进一步分析物种间的遗传网络,作者比较了基于深度学习的基序在物种间的保守性和差异性。作者在八个物种特异性模型中共识别出663个细胞类型特异性过滤器。约94.9%的细胞类型特异性过滤器至少与来自其他物种特异性模型的一个过滤器同源(图6)。并且同源过滤器倾向于保持物种间相似的细胞类型特异性。深度学习基序的跨物种比较显示出揭示特定细胞类型下保守调控因子富集的潜力。
图6 细胞谱系特异性过滤器的保守水平分布
总结
在这项研究中,作者利用自主构建的高通量单细胞测序平台Microwell-seq构建了斑马鱼、果蝇和蚯蚓的全生物细胞图谱。在没有组织特异性批次效应的情况下测量了整个细胞的平衡状态。这些细胞图谱为研究物种,特别是节肢动物和环节动物的细胞分类提供了前所未有的机会。在这项研究中,作者总共分析了八种具有代表性的后生动物物种,以生成一个详细目录,来说明动物进化过程中细胞平衡状态的保守性和多样性。为了区分趋同进化和协同进化,作者筛选了具有细胞类型特异性的细胞谱系特异性TF。该研究为在单细胞分辨率下深入理解比较基因组学提供了一个框架。未来的研究可能会收集更多的后生动物物种,以追踪重要细胞类型的出现和研究细胞进化。
同时,作者开发了一个基于深度学习的框架Nvwa,仅从DNA序列预测细胞图谱水平的基因表达。Nvwa具有与特定细胞状态相关的预测调控功能,这使作者能够直接从序列中筛选细胞类型特异性的调控因子。此外,Nvwa仅使用基因组序列就可以模拟多细胞生物的复杂表达模式。Nvwa从未使用任何表观基因组数据进行训练,但其全基因组活性预测与使用功能基因组学确定的候选调控元件相关。这些结果有两个含义。首先,利用基因组共享的基本规则,深度神经网络可以模拟多细胞基因表达图谱。第二,谱系特异性转录组在很大程度上由调控DNA序列决定。
虽然Nvwa为研究进化过程中细胞类型特异性调控程序提供了一个新的视角,但Nvwa模型的解释和应用仍需谨慎。首先,超参数和模型体系结构,特别是第一层卷积滤波器控制了序列模式解释的简并性和灵敏度之间的权衡,应该根据用户的特定目的进行调整。例如,可以增加滤波器数量以提高序列基序检测的灵敏度。其次,使用Nvwa进行全基因组预测可以帮助研究人员进行功能基因组研究,并填充高度重复的基因组区域。但由于Nvwa尚处于概念验证阶段,其预测结果与具体实验数据并不完全一致;因此,Nvwa应该在实践中作为辅助工具使用。第三,本研究聚焦于TF调控因子,并将深度学习衍生的序列模式解释为TF基序。然而,仍然有新的序列模式不能分配到已知的数据库。第四,通过功能实验验证调控元件非常重要。最后,基因调控机制复杂,模型的体系结构、预测性能和调控逻辑解释仍有待完善。
总之,作者生成了斑马鱼、果蝇和蚯蚓的全身单细胞转录组图谱,并开发了一种基于深度学习的模型Nvwa,来预测基因表达并识别单细胞水平的调控序列,作者还揭示了进化过程中保守调控程序的作用。该研究将为破解多物种调控图谱提供宝贵的资源。 参考资料
Li, J., Wang, J., Zhang, P. et al. Deep learning of cross-species single-cell landscapes identifies conserved regulatory programs underlying cell types. Nat Genet (2022). https://doi.org/10.1038/s41588-022-01197-7
数据 https://figshare.com/s/ecc05b1051fb5678fd3e http://bis.zju.edu.cn/nvwa/