KAUST高欣组研发基于深度学习的研究RNA和RBP相互作用的全新方法|Nat. Commun.
一项研究提出了一种基于深度学习的研究 RNA 和 RNA 结合蛋白(RBP)相互作用的全新方法“NucleicNet”,通过使用深度学习和 FEATURE 特征提取框架,该方法可以同时提供 RBP 和 RNA 相互作用时的结构信息以及大规模的结合性信息。
来源 沙特阿卜杜拉国王科技大学
编辑 戚译引
RNA 结合蛋白(RBP)是基因表达调控中不可或缺的一类生物分子,对于转录后调控尤为关键。比如,Argonaute 是 RNA 干扰(RNAi)的核心分子, PUF 蛋白可以直接影响 mRNA 的表达。深入地理解 RBP 的功能,尤其是它们如何和 RNA 结合并相互作用,可以进一步帮助我们研究转录后调控等一系列问题。
目前,有两类常用的实验方法被用来研究 RBP 和 RNA 的相互作用,分别是基于 assay 的大规模化验分析,以及面向结构的 RBP 和 RNA 结合复合物的结晶分析。两种实验方法都各有优势和不足:基于 assay 的分析可以大规模地测试 RBP 和 RNA 的结合强度及选择性,但是却无法揭示它们作用时候的结构细节,尤其是无法鉴别 RBP 和 RNA 是通过碱基直接相互作用,还是通过主链间接相互作用;另一方面,结晶分析可以揭示作用细节,却因为成本和实验属性等各方面原因无法进行大规模的实验,同时只解决了很多种可以与该 RBP 结合的 RNA 序列中的一种,因此不具有统计学意义。
能够同时提供统计学和结构性两种信息的方法,无论是基于实验的还是基于计算的,都可以极大地促进 RBP 的研究。遗憾的是,之前的计算方法同样无法同时提供这些信息。
之前的计算方法可以分为两类。一类是基于结构或者序列的预测器,可以用来预测 RBP 上的氨基酸位点是否为 RNA 结合位点。但是,这些预测器无法提供更精细的预测结果,比如结合位点上结合的是 RNA 的哪个基团(磷酸、核糖、腺嘌呤、鸟嘌呤、胞嘧啶、尿嘧啶)。这些现有的方法也没法预测 RBP 对于特定 RNA 的结合选择性和强度。另一类是是基于 assay 结果的计算方法,如 DeepBind。和 assay 实验方法一样,这类方法可以预测 RBP 和 RNA 的结合强度,但是它们无法给出作用的结构细节。
沙特阿卜杜拉国王科技大学高欣(Xin Gao)教授课题组(共同一作为 Jordy Lam,李煜)与香港科技大学黄旭辉教授课题组及南方科技大学陈炜教授课题组合作,在 Nature Communications 发表题为“A Deep Learning Framework to Predict Binding Preference of RNA Constituents on Protein Surface”的文章,提出了一种基于深度学习的研究 RNA 和 RBP 相互作用的全新方法。这种方法的预测结果可以同时提供 RBP 和大规模 RNA 的结合选择性信息,以及 RBP 和 RNA 结合的结构信息。
图片由研究团队提供。
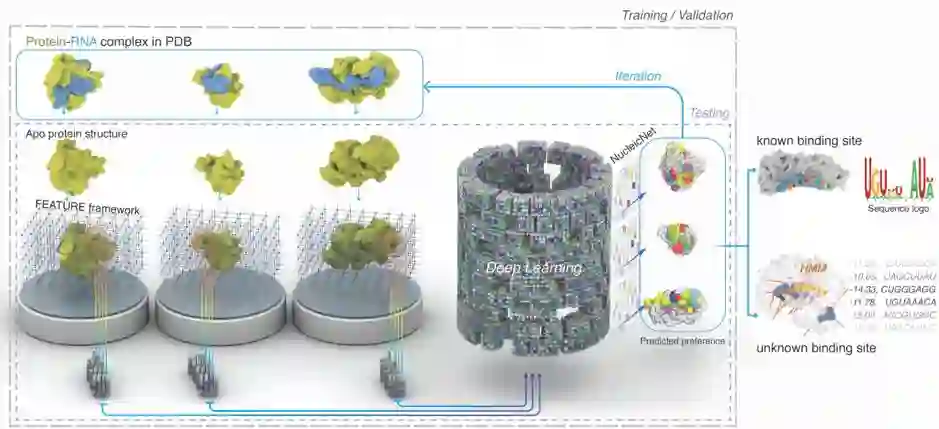
如上图所示,NucleicNet 从蛋白质的结构出发,首先在被研究蛋白质的表面产生空间点阵,然后预测空间点阵中的每一个点结合 RNA 各个基团(磷酸、核糖、腺嘌呤、鸟嘌呤、胞嘧啶、尿嘧啶)的概率。对于每一个空间点,该方法使用斯坦福大学 Russ Altman 课题组(也是本文作者之一)所研发的 FEATRURE 框架去提取和该点有关的结构以及理化性质信息。这些信息会被输入到一个深度学习模型之中,从而得到该点结合 RNA 各个基团的概率。不同点同 RNA 基团的结合情况合并到一起,就可以得到 RBP 与 RNA 结合的结构信息。同时,预测概率可以去评价每一个潜在的结合RNA,因为计算的并行性,我们可以在短时间内得到 RBP 和大量 RNA 的结合性。
对于已知的 RBP,NucleicNet 可以对所有可能结合的 RNA 序列进行打分和排序;对于未知的 RBP,NucleicNet 可以预测该蛋白与 RNA 结合的可能性,可能的结合位点,以及相应的结合 RNA,因此可以用来识别新的 RBP。
该文章同时使用了机器学习的标准评价方法以及实验方法去验证该方法的有效性。以机器学习的标准评价方式来看,该方法的准确性比基准方法提高了 35%。同时,在预测蛋白质上的氨基酸位点是否结合位点这个任务上,该方法也比之前那些专门做这个预测的方法要好,尽管 NucleicNet 并不是专门为了这个任务而提出并训练的。
该文章使用了三种生物实验去验证了方法的有效性。第一种实验是 RNACompete 实验。该实验使用 RBP 在大规模的 RNA 库中去竞争性地结合 RNA,从而得到该 RBP 和 RNA 的结合选择性。结果显示,NucleicNet 预测出的结合选择性和 RNACompete 实验结果得到的结合选择性非常吻合(Pearson 相关系数可以高达 0.8)。
第二个实验是让 NucleicNet 去区分能和 Ago 形成成熟的 RISC complex 的 Guide RNA strand 以及被释放的 Passage RNA strand。直观来看,Guide RNA strand 同 Ago 的结合性应该比 Passage RNA strand 要高,因为 Guide RNA strand 最终可以和 Ago 结合形成了稳定的复合物。预测的结果显示,在 222 例实验中,NucleicNet 可以正确预测其中 76% 的实验,给 Guide RNA strand 更高的结合预测分。值得一提的是,NucleicNet 的训练数据不含有任何 Assay 的信息,因此其预测结果与 Assay 实验的高吻合度说明了 NucleicNet 从结构数据中学习蛋白质和 RNA 结合的统计学规律的能力。
最后一个实验是验证 NucleicNet 预测的 Ago 和 siRNA 的结合强度分是否和该 siRNA 的敲除效率成正相关。结果显示,在 37 例基因中,22 例实验呈正相关。也就是说,虽然很多因素会影响到 siRNA 的敲除效率,Ago 和 siRNA 的结合程度会在一定程度上影响到敲除效率。同时,NucleicNet 有可能帮助大家设计敲除效率更高的 siRNA。
总的来说, NucleicNet 是一个研究 RBP 和 RNA 结合的全新方法。通过使用深度学习和 FEATURE 特征提取框架,该方法可以同时提供 RBP 和 RNA 相互作用时的结构信息以及大规模的结合性信息。大量的计算实验和生物实验验证了该方法的有效性。除了提出了一个研究 RBP 和 RNA 结合的全新方法,该文章提出的框架同样适用于其他类似的问题,比如蛋白质和小分子以及药物作用。该框架同样为新药物的研发提供了新的思路。
NucleicNet 为用户提供了网络服务器,点击了解更多:https://www.cbrc.kaust.edu.sa/NucleicNet/
本文来自微信公众号“科研圈”。如需转载,请在“科研圈”后台回复“转载”,或通过公众号菜单与我们取得联系。