作者 | 杨柳青 审核 | 刘洛涛 今天给大家分享的是印度德里英德拉普拉斯塔信息技术研究所和澳大利亚昆士兰前列腺癌研究中心发表在Nature Communications上的论文《Gene expression based inference of cancer drug sensitivity》。本文介绍了一种使用基因表达数据预测癌症治疗药物敏感性的深度神经网络模型——Precily。作者将药物的结构特性与基因表达的通路特异性相结合作为特征来训练模型,该模型在体外和体内预测药物反应都取得了较好的表现。 摘要**
**
肿瘤异质性是癌症治疗的主要绊脚石,也是癌症患者产生不同药物反应的主要原因。现阶段使用的癌症药物主要针对常见的癌症治疗靶点,但并不是所有的癌症和抗癌药物都与常见的、研究透彻的肿瘤标志物有关,而且在不考虑耐药性的情况下实施靶向治疗可能会降低患者的生存率。所以对癌症患者进行预先绘制分子图谱和提前推断药物反应是必要的。作者介绍了一种使用基因表达数据推断癌症治疗反应的深度神经网络模型——Precily,展示了将通路活性与药物描述符结合起来作为特征的优势。作者将Precily应用于高通量数据集中与癌细胞系相关的bulk RNA-seq和scRNA-seq测序数据,并且使用作者自己的前列腺癌细胞系和暴露在不同治疗条件下的异种移植数据集来评估模型对药物反应的可预测性,证明了该方法的适用性。
****方法
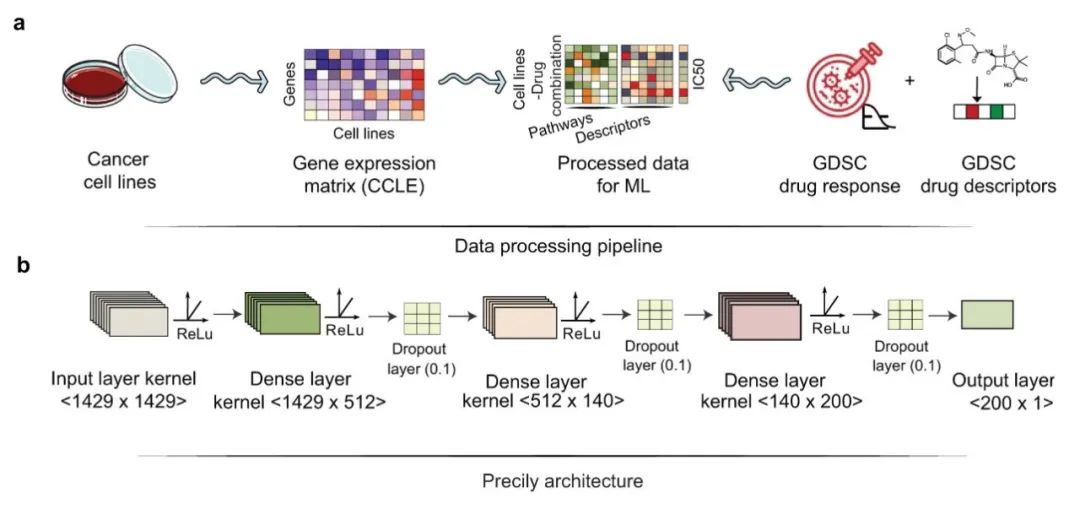
**1. 输入数据预处理
(1)分子描述符:基于从CCLE数据库中筛选的550个细胞系在GDSC数据库中获得了192种化合物的药物反应信息,基于1517名TCGA患者的用药情况获得了215种化合物的临床反应信息,对这些化合物进行SMILES检索,过滤掉没有SMILES描述符的药物分子,最后获得550个CCLE细胞系对应的173个化合物的SMILES和1443个TCGA患者对应的139个化合物的SMILES。使用smiles2vec工具将这些SMILES转换为大小为100的向量。 (2)通路活性分数:从分子特征库(MSigDB)中选取包含1329个基因集的C2典型通路集合作为输入的参考基因集,以log2(TPM+1)基因表达矩阵作为输入,使用基因集变异分析(GSVA)R软件包计算GSVA得分,得到通路得分矩阵。将通路得分矩阵与药物特征向量嵌入相结合,最终CCLE细胞系训练数据集包括80056个细胞系药物组合行和1429个特征,其中1329个路径和载体大小为100的药物特征作为解释变量,LN IC50作为反应变量列。对于TCGA患者数据,将单个癌症类型的基因表达谱转化为通路得分,根据共同通路合并每个癌症类别中药物反应信息可用的样本的GSVA得分。最终矩阵包括3108个患者药物组合和1427个特征(通路得分和药物描述符),反应变量为患者是否对药物有反应(有反应者=1,无反应者=0)。2. 训练模型 作者制定了用通路得分矩阵和药物分子描述符作为决策变量,LN IC50值作为反应变量的有监督回归任务。作者利用Keras框架构建和训练了深度神经网络(DNN),DNN体系结构包括一个能输入所有特征的输入层,隐藏层和输出层,每层以ReLU作为激活函数。作者将CCLE训练数据集分为90%的训练集(72262个细胞系-药物对)和10%的测试集(7794个细胞系-药物对),以便细胞系之间没有重叠。使用五折交叉验证进行超参调优,使用Adam优化器来优化学习率。最小化均方误差损失。
**
结果
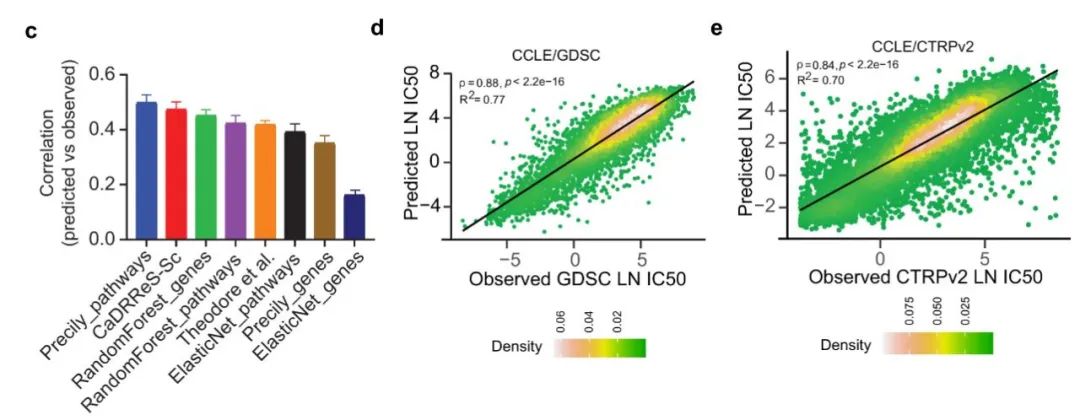
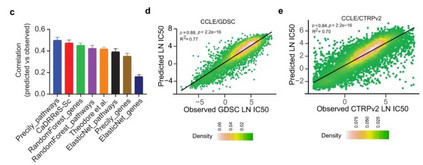
**1. Precily能够在癌细胞系中预测可重复的药物反应 作者在未利用化合物结构信息的情况下将Precily与两种广泛应用的、基于基因表达预测药物敏感性的方法(CaDRReS方法和Theodore等人的方法)和传统的机器学习方法进行比较,证明基于Precily-pathway的预测与基本事实的相关性最高,其次是CaDRReS-Sc(图c)。但仅基于基因表达谱的预测方法是不理想的,因为它没有利用化合物的结构信息。将药物结构信息与pathway汇总进行预测时,Precily的药物敏感性预测值获得了0.88(R2=0.68,p value<2.2e-16)的Pearson 相关系数值(图d)。除此之外,作者利用CTRPv2数据库中的另一批小分子抗癌药物与CCLE的细胞系组合进行预测,初步得到的皮尔逊相关系数为0.84(R2=0.70;p value<2.2e-16)(图e)。上述分析证明,Precily对于预测癌细胞系对抗癌药物治疗的敏感性具有合理的准确性和可重复性。
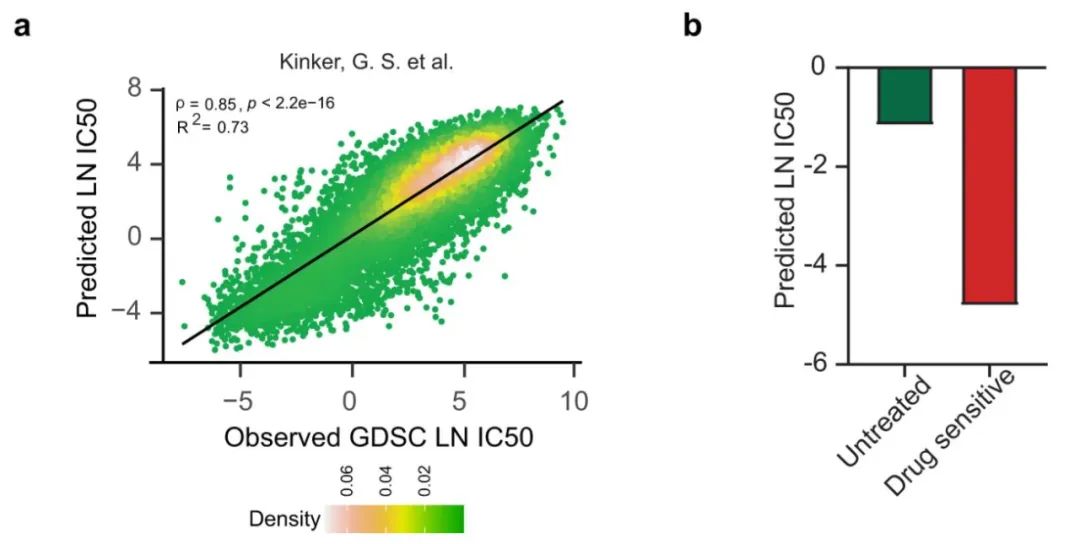
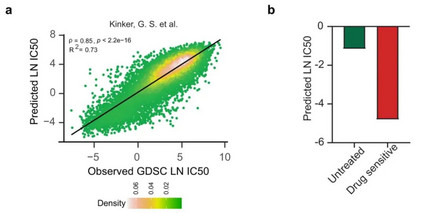
- 利用单细胞表达谱预测药物反应 为了证明Precily在单细胞表达谱水平上预测药物反应的潜力,作者使用了两个现有研究的单细胞转录组数据集进行分析。首先使用Kinker G.S.等人提供的207个癌细胞系scRNA-seq数据,其中116个细胞系与CCLE数据集重叠。作者重新训练了CCLE/GDSC模型,使得Kinker,G.S.等人的细胞系数据集从未用于模型训练,将重新训练后的模型用于Kinker G.S.等人的数据集,模型预测值与真实值的皮尔逊相关系数=0.85(R2=0.73;p value<2.2e-16)(图a)。在另外一项关于转移性乳腺癌的研究,Precily也正确预测了药物治疗反应(图b)。
- 前列腺癌细胞系不同治疗策略分析 作者将Precily应用于自己研究的五个未经药物处理的前列腺癌(PCa)细胞系的bulk RNA-seq数据,每个细胞系进行两个生物学重复。并预测了GDSC数据库中针对不同细胞通路的155种药物在上述十个样本的药物反应。发现雄激素受体(AR)阳性的PCa细胞系(LNCaP、DuCaP和VCAP)与AR阴性的细胞系(DU145和PC3)相比对药物相更敏感,且根据药物与通路的关系清楚预测了LNCaP细胞对PI3K/mTOR信号通路靶向药物的潜在敏感性(图a、b、c)。另外,作者预测在雄激素受体(AR)激动剂双氢睾酮(DHT)和AR拮抗剂比卡鲁胺(BIC)、恩杂鲁胺(ENZ)和阿帕鲁胺(APA)存在的不同情况下,LNCaP细胞系对药物的敏感性变化(图d、e、f),从而暗示了Precily在确定潜在的前列腺癌联合疗法方面具有指导意义。
- 患者临床反应的可预测性作者基于TCGA数据集中患者肿瘤细胞RNA-seq数据、患者用药情况及用药反应,使用自动机器学习(AutoML)H2O.ai的现成R库构建药物反应分类器,用于预测患者对药物治疗是否有反应。AutoML共评估了34个模型(包括机器学习、深度学习、增强模型和集成模型),并提供了“极端随机树”(XRT)作为最佳模型,且XRT在测试数据集中的AUC-PR为0.85(图a)。作者的分析结果也表明患者的药物反应概率与生存风险之间具有相关性。另外,作者将TCGA模型应用于一个与黑色素瘤相关的独立数据集时能够正确预测三位患者的耐药性,从而评估了TCGA模型的可用性和准确性。
更多、更详细的实验方法与结果请查看原文。 论文链接:https://www.nature.com/articles/s41467-022-33291-z 代码链接:https://github.com/SmritiChawla/Precily