今天给大家介绍Utrecht University的Alexandre M. J. J. Bonvin团队发表在Bioinformatics上的文章《DeepRank-GNN: A Graph Neural Network Framework to Learn Patterns in Protein-Protein Interfaces》。文章介绍了DeepRank-GNN,一个用于学习蛋白质-蛋白质界面信息的图神经网络框架,并在文中展示了两个功能,分别是对接模型的评估与生物与人工晶体PPIs的分类,且均取得了颇有竞争力的表现。
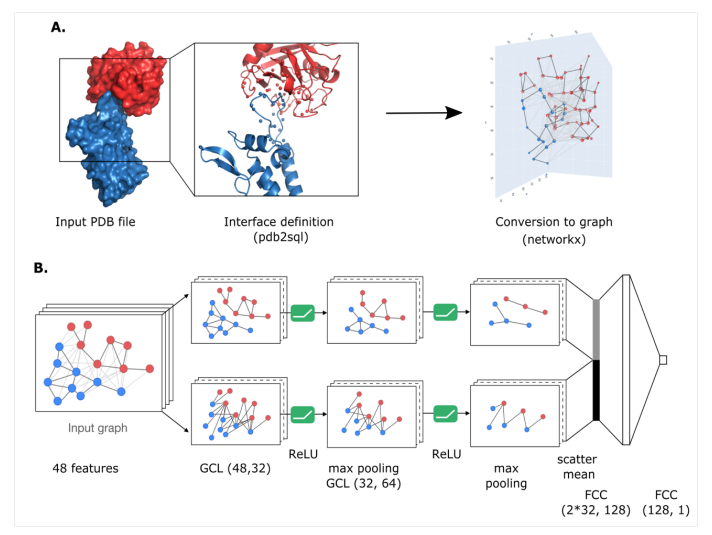
1 DeepRank-GNN介绍 DeepRank-GNN继承了DeepRank的思想,是一个即插即用的Python依赖包, 旨在对蛋白质-蛋白质复合物数据集进行端到端的训练,但不同的是DeepRank-GNN基于图神经网络,此外还做了进一步的优化。相较于DeepRank来说,本文提出的方法不仅消除了旋转不变性,而且在效率上取得了大幅的提升。
图1 DeepRank-GNN框架图
2 方法
DeepRank-GNN总览 DeepRank-GNN以PDB的3D坐标为输入,通过pdb2sql工具定义界面,形成残基级别的图数据,并提供了丰富且可自定义的节点特征化方式与边特征化方式,最终以HDF5的形式存储。在骨干网络的选择上,本文提出的方法提供了多样的选择,用户可以选择使用文章提供的预训练模型,或者自定义特定的网络结构。训练目标也同样,用户可以在分类目标与回归目标之间自由切换。
应用1-对接模型的打分。 作者在BM5数据集上训练模型,在CAPRI数据集上测试模型效果。在这一部分,作者设计了一个基于GNN的网络结构并命名为GINet(如图1B所示),以fnat (the fraction of native contacts)为训练目标构建回归任务,并计算了AUC、命中率与成功率作为评估标准。
应用2-生物与人工晶体PPIs分类 作者在MANY数据集上训练模型,在DC数据集上测试模型效果。神经网络结构沿用了上一部分的GINet构建分类任务,在Accuracy、Specificity、Sensitivity和Precision等指标上评估了模型表现。
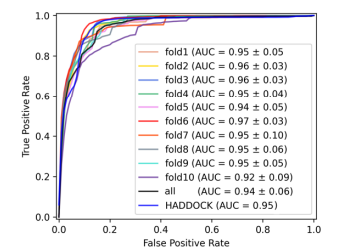
图2 DeepRank-GNN与HADDOCK打分函数在BM5数据集上的比较
3 结果
应用1-对接模型的打分。 在BM5数据上进行10折交叉验证后,作者发现其中8折的表现都好于HADDOCK打分函数(如图2所示),其AUC值均大于等于0.95,但表现出了明显的数据集依赖性。为了更好的体现出模型优秀的打分能力,作者进一步计算了fnat与预测值的斯皮尔曼相关系数,均值0.49±0.14鲜明地体现了模型的优异性能。作者还在CARPI数据集上与其余软件进行了比较,如表所示,DeepRank-GNN在AUC于Top100的成功率上均取得了最好的表现。最后,作者验证了方法在性能上的优势,相较于DeepRank,本文提出的方法在存储与速度上都展现出大幅的超越。
应用2-生物与人工晶体PPIs分类 作者将在MANY数据集上训练好的模型在DC数据集上进行测试,获得了82%的准确率、81%的特异度、83%的灵敏度以及82%的精确率。考虑到DC数据集与MANY数据集有重叠,在去重后各项指标分别为81%、82%、79.2%与80.3%,只产生了轻微的影响。在于外部软件的比较上,DeepRank-GNN超越了PISA与PRODIGY-CRYSTAL,仅次于DeepRank。
4 总结 作者团队开发了一个从蛋白质-蛋白质界面数据中学习和预测相互作用模式的计算框架DeepRank-GNN,并为用户提供了丰富的自定义选项。在对接模型打分的任务中,DeepRank-GNN展现出了具有竞争力的表现,强调了几何性质与物化性质同样具有表达能力。在生物与人工晶体的辨别中,本文提出的方法同样达到了与SOTA模型相近的结果,但在效率上更胜一筹。总之,DeepRank-GNN是一个高效学习蛋白质-蛋白质界面模式的软件,其模块化与可扩展的特性允许社区更进一步地拓展,推动蛋白质-蛋白质界面学习的发展。
参考资料 Manon Réau, Nicolas Renaud, Li C Xue, Alexandre M J J Bonvin, DeepRank-GNN: A Graph Neural Network Framework to Learn Patterns in Protein-Protein Interfaces, Bioinformatics, 2022;, btac759, https://doi.org/10.1093/bioinformatics/btac759