作者 | 王郅巍
审核 | 付海涛
今天给大家介绍的是来自中山大学智能工程学院的陈语谦教授团队发表在Briefings in Bioinformatics上的文章"3DGT-DDI: 3D graph and text based neural network for drug–drug interaction prediction".传统的DDI预测模型只考虑分子的2D拓扑结构,忽略了其3D结构信息。本文提出一种新的方法,3DGT-DDI,由3D图神经网络和预训练的文本注意机制共同组成。该方法创新之处在于利用了3D分子图结构和位置信息来增强模型对DDI的预测能力。实验表明,3DGT-DDI的预测性能优于其他基线模型。此外,该模型还通过DrugBank数据集上的权重可视化证明了自身的可解释性。
1.摘要
药物-药物相互作用(DDI)发生在药物组合过程中。识别潜在的 DDI 有助于我们研究联合用药或不良反应背后的机制,从而避免副作用。尽管已有许多挖掘和预测潜在DDI的方法被提出,但它们都忽略了药物分子的3D结构信息,没有充分考虑分子亚结构在DDI中的贡献。基于此,本文提出了一种新的深度学习架构,3DGT-DDI,由3D图神经网络和预训练文本注意机制组成,用3D分子图结构和位置信息来增强模型对DDI的预测能力,使模型能深入探索药物亚结构对DDI关系的影响。结果表明,3DGT-DDI的预测性能优于其他最先进的模型。此外,对文本和分子图结构的特征权重可视化证明了3DGT-DDI的可解释性。
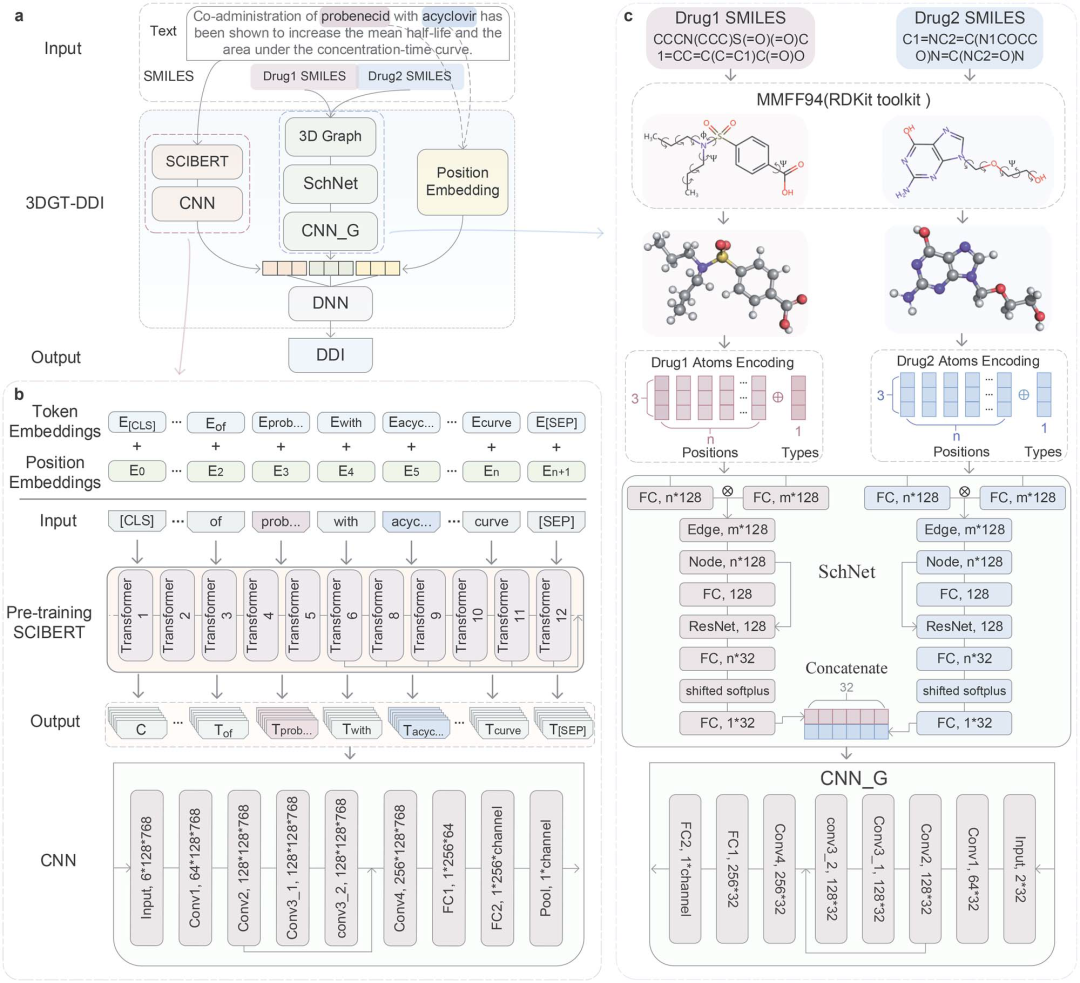
2.模型介绍 3DGT-DDI的整体框架如下:
该模型主要由3部分组成:3D图神经网络、预训练的文本注意力机制和位置嵌入。 2.1 3D图神经网络
3D图用表示,其中为全局特征向量,为节点特征向量,为边集,为节点的位置信息。一般的 3D 图模型消息传递更新方程类似于: 3DGT-DDI使用SchNet作为3D图模型,输入原子的位置表示和类型,进行几何建模: 1. 输入药物的SMILES,生成3D构象,利用MMFF94优化得最优构象的原子位置表示和类型; 2. 代表个原子的特征表示; 3. SchNet的滤波器函数将位置(距离)映射到具体的滤波器值,从而更新原子特征: 4. 聚合上述信息,SchNet的消息更新函数如下: 5. 最后,利用CNN融合SchNet得到的特征,得最终特征表示: 2.2 文本及位置嵌入
输入文本信息,通过融合SCIBERT的多个隐藏层得到文本嵌入,此外还加入位置嵌入,以便提取更完善的上下文信息。 1. 输入文本 2. 对于每一个,生成三个向量Query,Key,Value: 3. 计算Attention: 4. 最后,融合SCIBERT的多个隐藏层,利用CNN进行卷积,获得最终的文本嵌入: 其中,为融合的隐藏层数。
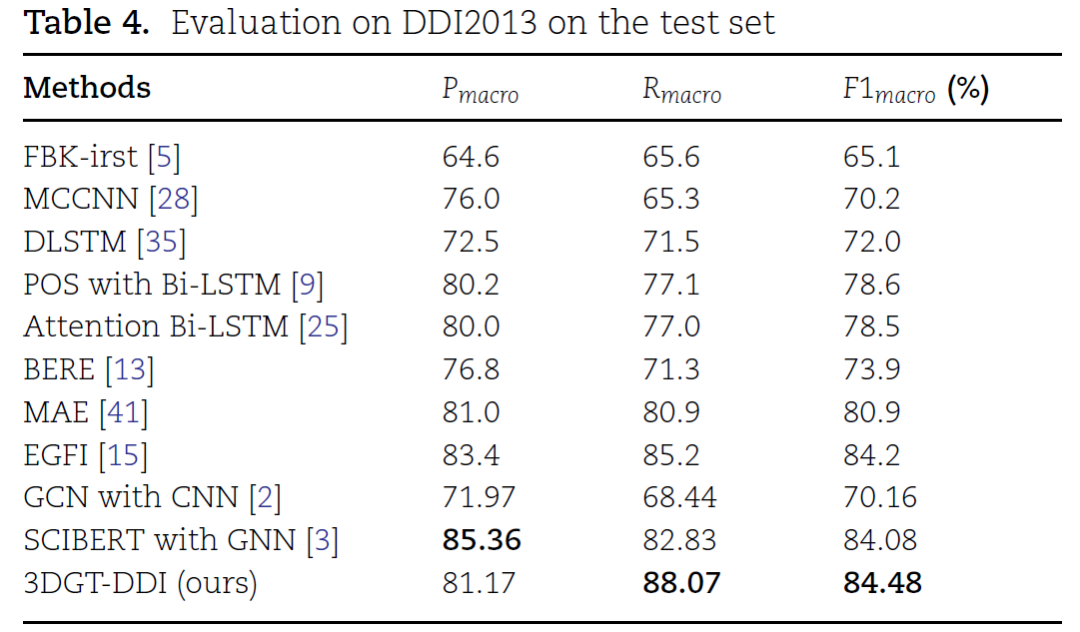
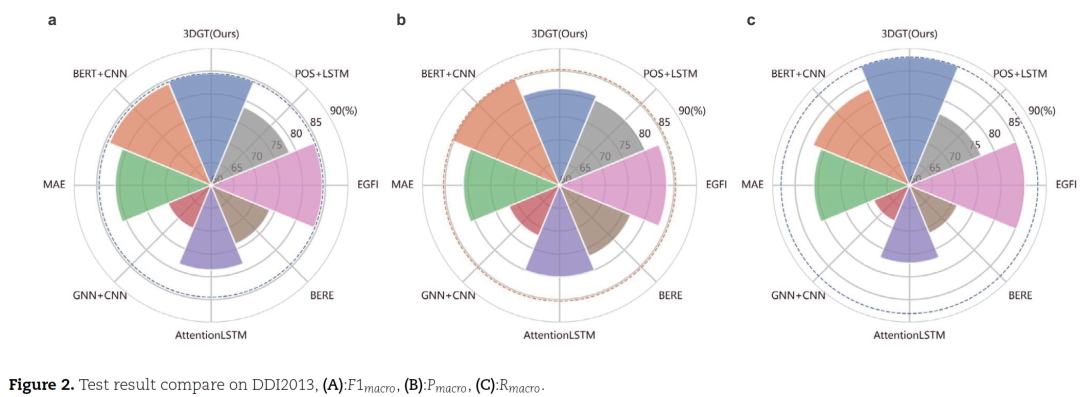
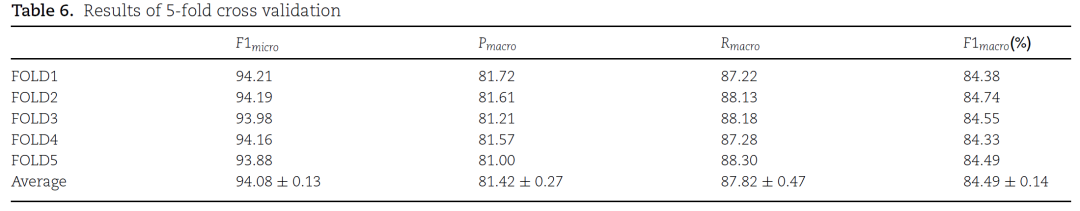
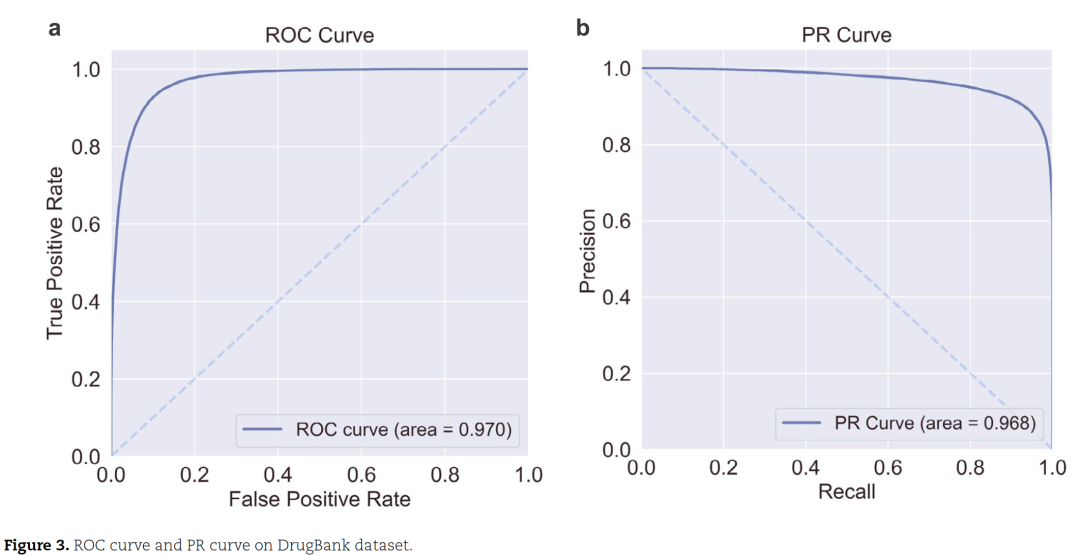
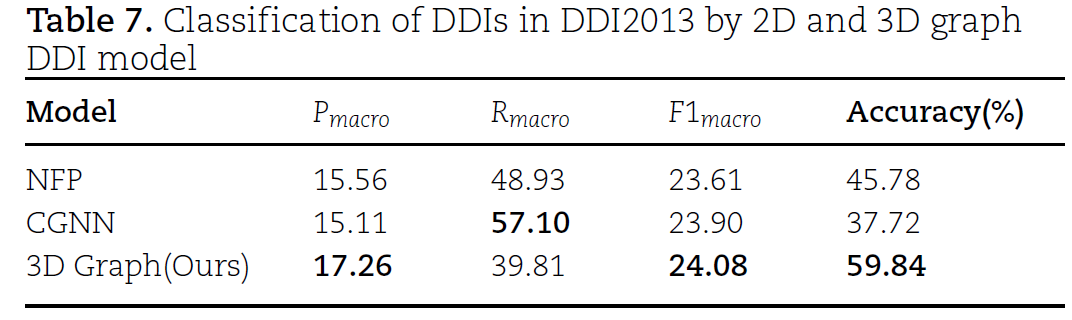
3.实验 本文在DDI extraction 2013和DrugBank数据集上进行实验。 3.1. 模型性能
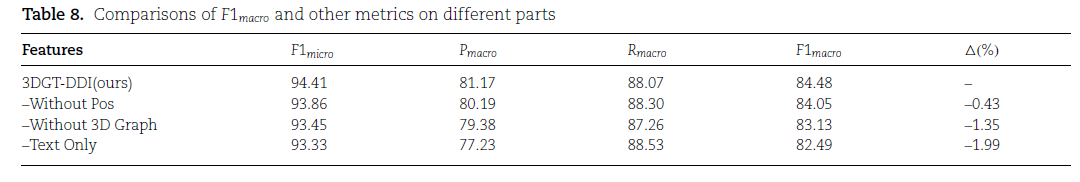
结果表明,3DGT-DDI的预测性能优于目前最先进的模型。 3.2. 消融实验
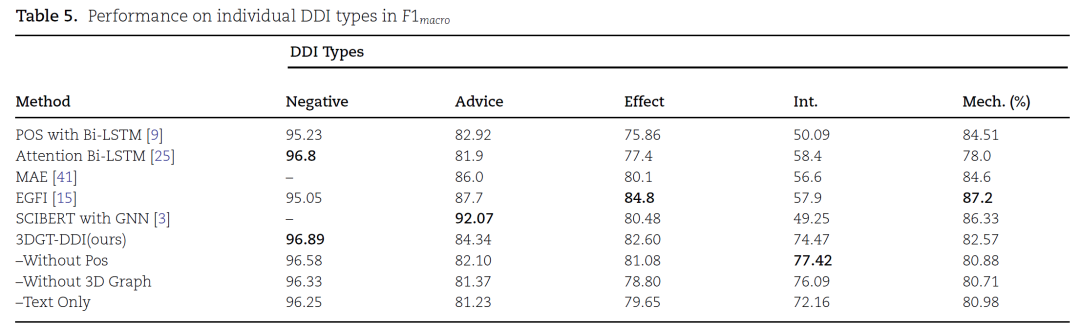
结果表明,使用所有组件(3D图神经网络、预处理的文本注意力机制和位置嵌入)的3DGT-DDI模型表现最好。 3.3. 误差分析
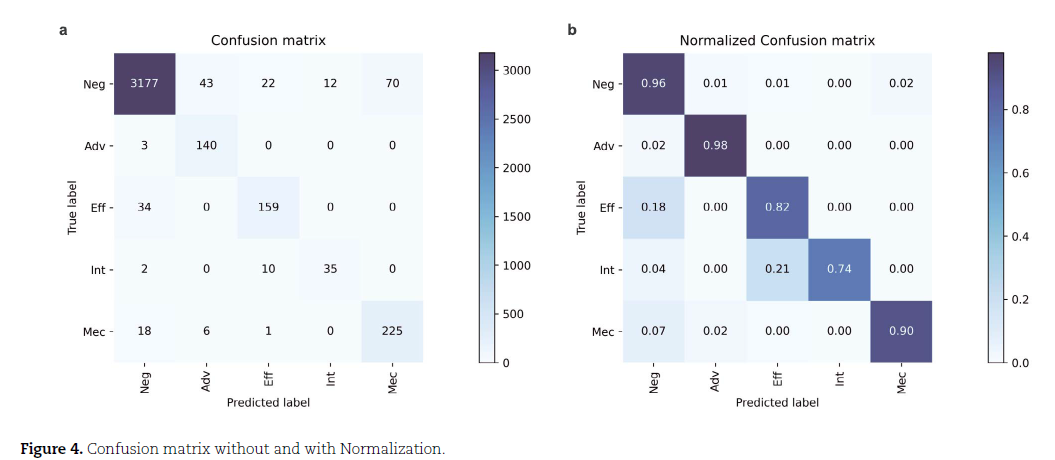
3.4. 可解释性
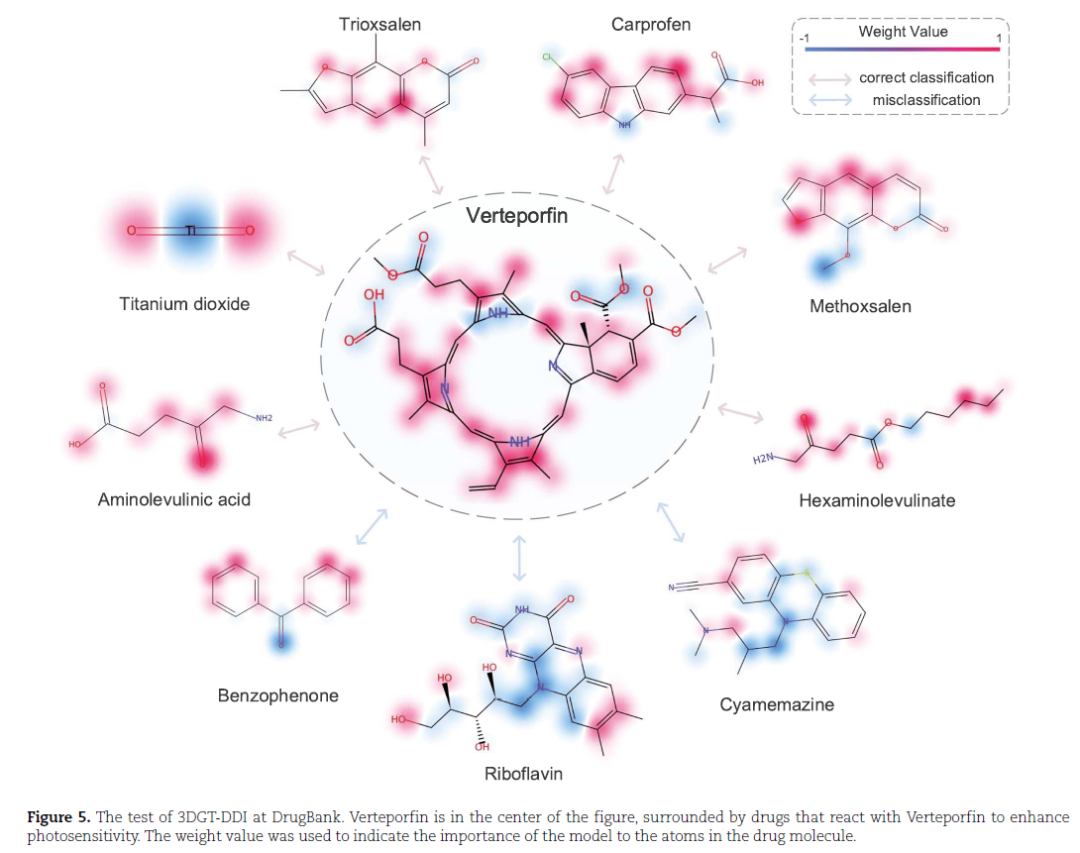
3D 图网络的最后一层输出上,分子上每个原子的注意力权重代表其对分类的影响。 光敏剂Verteporfin位于图的中心,周围是与其发生反应以增强光敏性的药物:
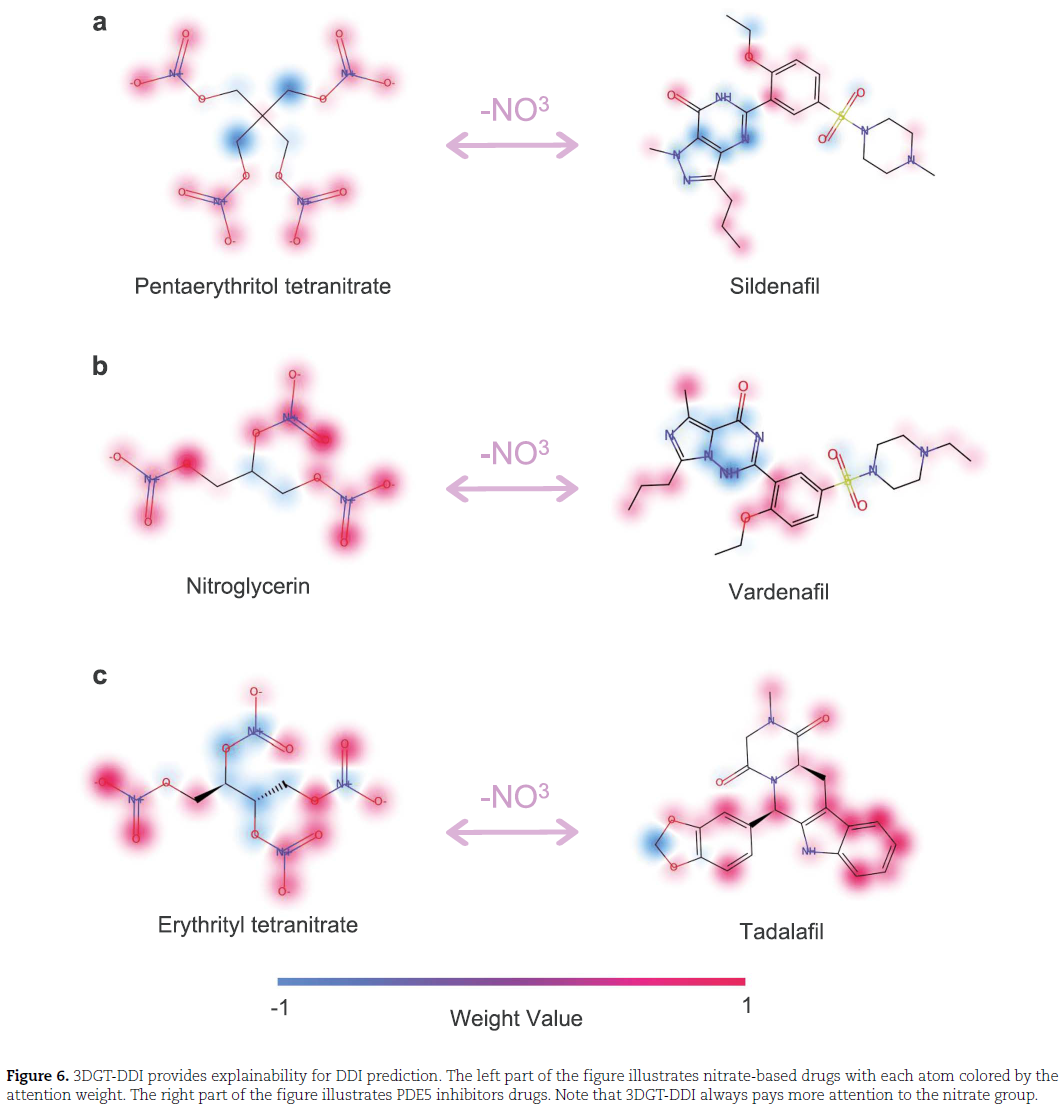
硝酸盐药物与PDE5抑制剂产生相互作用:
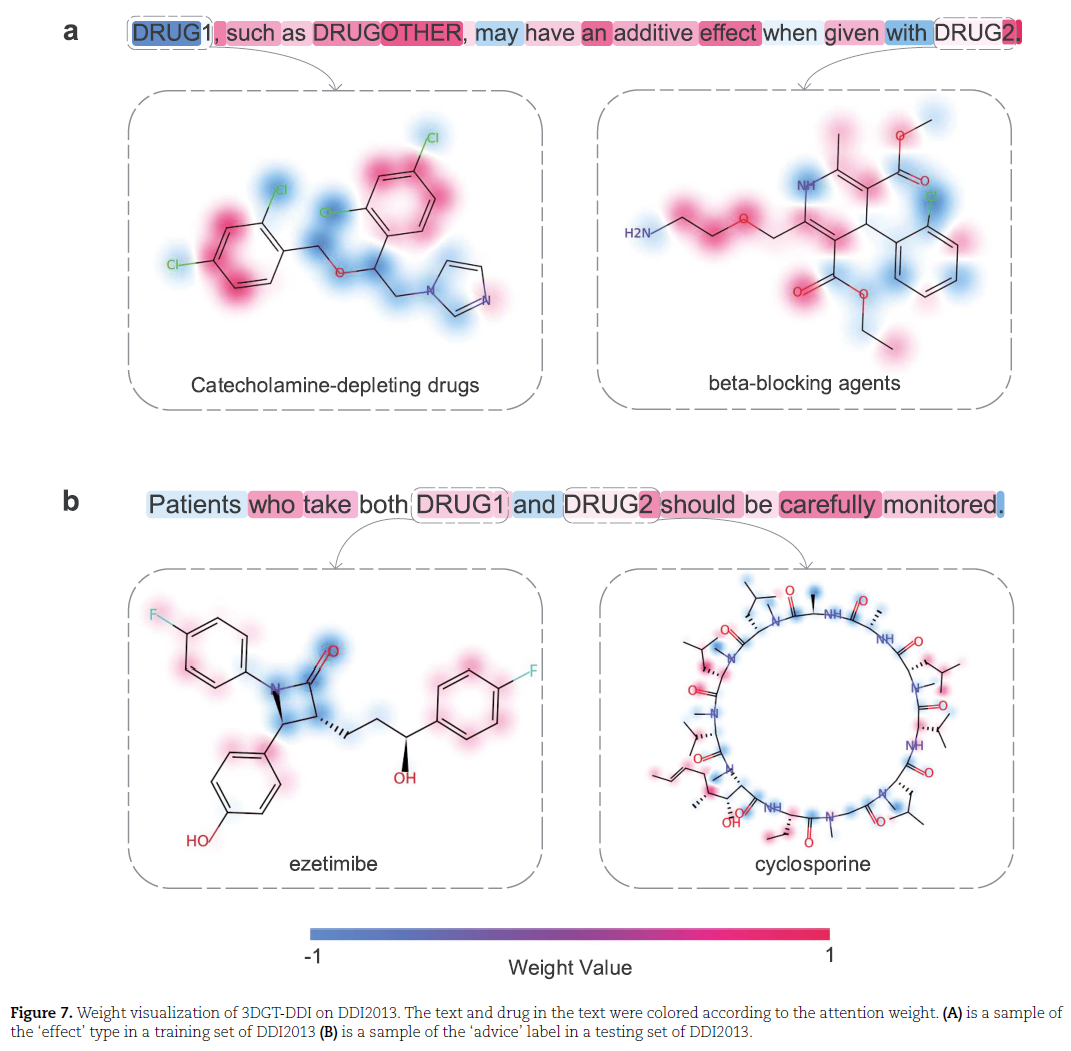
结合文本信息及药物结构的注意力权重:
4.总结
本文提出了一个基于注意力的深度学习框架3DGT-DDI,由预训练的文本模型和3D图模型组成,用于预测和深入解释DDI。与传统方法使用分子2D信息不同,本文引入了3D信息,将其与文本药物描述信息相结合。实验表明,3DGT-DDI性能优于其他基线模型;且通过可视化文本和分子图结构的特征权重,3DGT-DDI具有较好的可解释性。总的来说,3DGT-DDI学习了药物分子的3D结构,并结合了药物分子的文本信息,提高了DDI预测效果;可视化DDI可以有效帮助医学研究人员更深入地了解联合用药或不良反应背后的机制。
参考文献
文章地址 https://academic.oup.com/bib/article-abstract/23/3/bbac134/6576451