|导语|
图神经网络因其对图结构数据的强大表达能力而受到越来越多的关注,但它们仍然因为缺乏可解释性而受到质疑。当前可解释性人工智能方法受限于数据集,仅在社交网络等现实世界数据集上进行定性评估,缺少定量评估和比较。同时,可解释性方法生成的解释是否达到预期目的并提供可靠的答案也仍待探索。中山大学杨跃东教授团队联合星药科技研发团队在Cell Press旗下Patterns期刊发表了题为“Quantitative evaluation of explainable graph neural networks for molecular property prediction”的文章,**该研究建立了五个分子可解释性基准数据集,定量评估了六种常用的可解释性方法与四种图神经网络模型的组合,并与药物化学家在可解释性任务上进行了直接比较。**这是首次将可解释性方法与人类专家在可解释性任务上进行定量实验比较的研究。同时,基于模型学到的解释,研究人员开发了一种数据驱动的分子结构指纹,可作为分子属性预测中经典分子指纹的补充。相关成果[1]已于11月正式发表。
近年来,由于图结构数据的强大表达能力,图神经网络的研究越来越受到关注,其应用领域包括了社交网络、推荐系统和自然科学等。尽管前景广阔,图神经网络仍然因为其缺乏可解释性而广受质疑,因为这些模型通常被认为是“黑匣子”。可解释的人工智能方法(XAI)已被用于解释图神经网络模型。然而,因为缺少用于定量评估的真实数据集,这些XAI方法通常仅在社交网络等现实世界数据集上进行定性评估。 相比之下,可解释性的先验知识通常存在于科学数据集中,特别是在化学和药物发现领域[2]。例如,在毒性预测中,关键子结构(也称为结构预警, structural alert)通常是与毒性发生概率增加密切相关的片段。而在性质断崖的情形下,则更为复杂,因为一些子结构的轻微变化会导致分子性质发生显著变化。这些现实而复杂的场景和数据,为可解释性方法的发展带来巨大挑战的同时,也带来了一定机遇。 **在这项研究中,研究团队建立了五个分子可解释性基准数据集,包括两个合成数据集和三个实验数据集,用于定量评估 XAI方法与图神经网络的可解释性。**通过这些基准,研究人员定量地评估了六种可解释性方法与四种图神经网络的组合,**并与七位不同经验水平的药物化学家在可解释性任务上进行了实验比较。结果表明,当前的可解释性方法可以为药物化学家提供可靠且信息丰富的答案。**而基于学到的可解释子结构特征,研究人员开发了一种数据驱动的指纹,可作为分子属性预测中经典指纹的补充,在多个分子属性基准数据集上得到了验证。
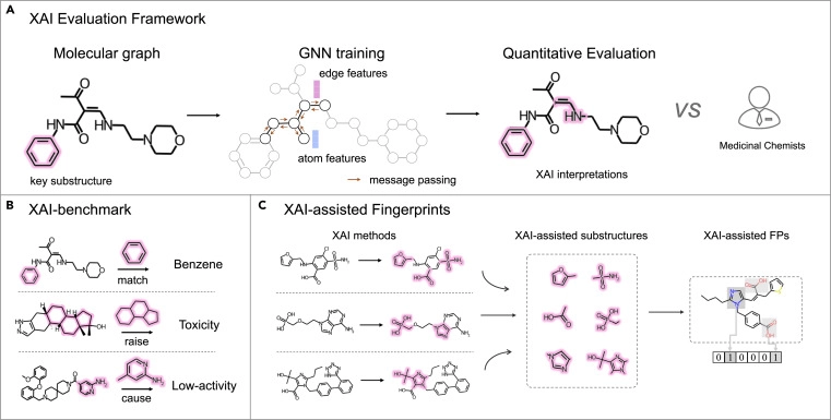
图1. 可解释性数据集以及评估框架示意图 定量评估XAI方法在图神经网络中的可解释性
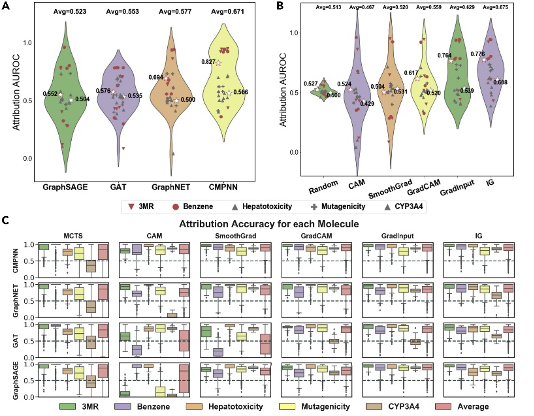
为了定量评估XAI方法在图神经网络中的可解释性,研究人员在五个基准数据集上评估了六种常用的可解释性方法与四种图神经网络的组合,如图2所示。当比较不同图神经网络的可解释性能时,CMPNN得到了最高的Attribution-AUROC值(0.671), 而 GraphNET 和GAT分别排名第2和第3。当比较不同 XAI方法时,IG方法表现最佳,得到了最高 Attribution-AUROC值为0.675,其次是 GradInput 和 GradCAM,其Attribution-AUROC值分别为0.629和0.559。可视化结果由图3展示。
图2. 可解释性定量评估实验结果图
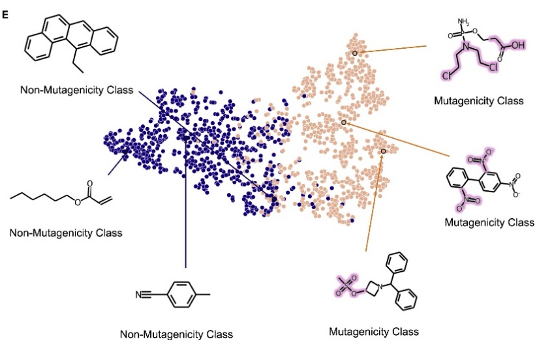
图3.可视化结果图
对比药物化学家与可解释AI模型在识别分子肝毒性上的能力
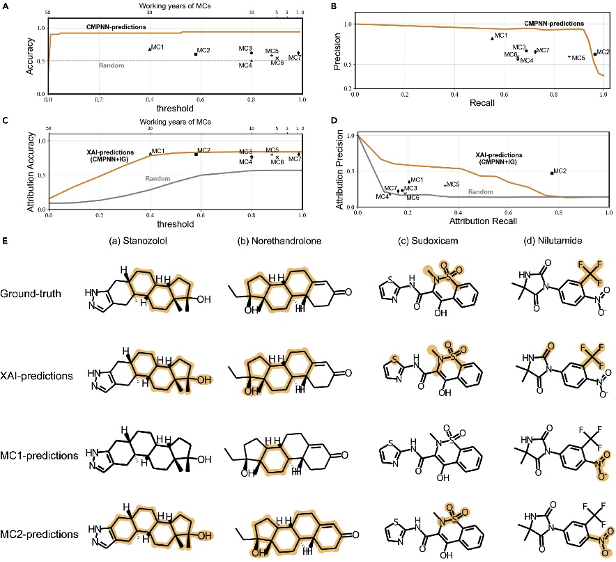
为了评估可解释性方法的真实能力,研究人员邀请了7位不同经验水平的药物化学家进行了实验比较。研究人员从肝毒性数据集的外部测试集中随机选择了50 个未标记的分子,并要求XAI模型和药物学家预测这些分子是否具有肝毒性及其引起该毒性的关键子结构。 如图4所示,XAI模型在预测肝毒性的准确度上,要优于所邀请的药物学家,而药化学家对于肝毒性的预测准确度与工作年限呈正相关。在肝毒性的子结构识别方面,XAI模型的预测精度略低于一位工作经验长达20年的药化学家。研究人员也通过代表性的案例分析了药化学家与XAI模型对肝毒性子结构预测的偏向和趋势。
图4. 与人类专家的定量对比实验结果图 基于可解释子结构的分子指纹
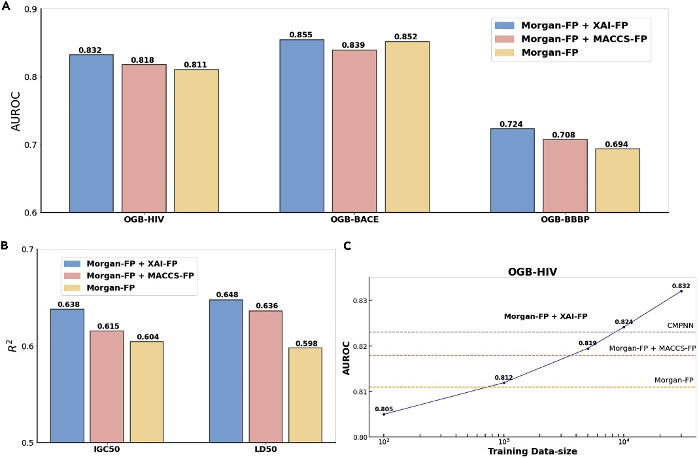
研究人员利用可解释性方法识别的关键子结构,构建了一种数据驱动的分子结构指纹,在五个分子属性预测的公开基准上进行了实验测试。结果如图5所示,该分子指纹在三个分类任务中分别比单独使用经典分子指纹Morgan-FP高出了2.59%、0.35%和4.32%,在回归任务上分别高出8.36% 和5.63%。与此同时,研究人员通过不同数据量下的对比实验,验证了增加训练样本有望提高该分子指纹的表征性能,表明了这种数据驱动方法的优势。
图5. 可解释性分子指纹实验结果图 结语
本研究建立了五个分子可解释性基准数据集,定量评估了可解释性方法与图神经网络的组合,并与药物化学家在可解释性任务上进行了直接比较。这是首次将可解释性方法与人类专家在可解释性任务上进行定量实验比较的研究。实验评估表明,当前的可解释性方法与图神经网络可以在识别分子属性和关键子结构方面提供可靠且信息丰富的答案,并且模型得到的解释也可为分子属性的预测提供帮助。文末,作者对当前可解释性方法的一些局限性也作了讨论。未来,本研究建立的基准数据集和定量评估框架,也将更好地评估、比较模型的可解释性,进而推动可解释性方法的发展。 作为应用前沿人工智能技术赋能药物研发的代表性企业,星药科技在AI驱动分子设计、虚拟筛选、性质预测、复合物结构预测、合成生物学研究等领域的落地转化方面进行了大量探索并取得了一定的成果[3][4][5][6]。目前,星药科技已通过自研平台Pyxir®和M1确定了两种新型的治疗自身免疫性疾病的临床前候选化合物(PCC)[7],验证了其AI药物研发技术能力的闭环。接下来星药科技将秉承初心,继续聚焦在能为医药产业带来更多增益的差异化管线,尤其是未成药、难成药靶点的管线研发。 参考资料 [1]. Rao, J., Zheng, S., Lu, Y., and Yang, Y. (2022) Quantitative Evaluation of Explainable Graph Neural Networks for Molecular Property Prediction. Patterns, 100628. [2]. Jiménez-Luna, J., Grisoni, F., & Schneider, G. (2020). Drug discovery with explainable artificial intelligence. Nature Machine Intelligence, 2(10), 573-584. [3]. Wang, P., Zheng, S., Jiang, Y., Li, C., Liu, J., Wen, C. & Yang, Y. (2022). Structure-Aware Multimodal Deep Learning for Drug–Protein Interaction Prediction. Journal of Chemical Information and Modeling, 62(5), 1308-1317. [4]. Chen, J., Zheng, S., Song, Y., Rao, J., & Yang, Y. (2021). Learning Attributed Graph Representations with Communicative Message Passing Transformer. IJCAI 2021. [5]. Wang, J., Zheng, S., Chen, J., & Yang, Y. (2021). Meta learning for low-resource molecular optimization. Journal of Chemical Information and Modeling, 61(4), 1627-1636. [6]. Lu, W., Wu, Q., Zhang, J., Rao, J., Li, C., & Zheng, S. (2022). TANKBind: Trigonometry-Aware Neural NetworKs for Drug-Protein Binding Structure Prediction. bioRxiv. [7].https://mp.weixin.qq.com/s/nXUgKL5qb6vZgGKXUegLqA