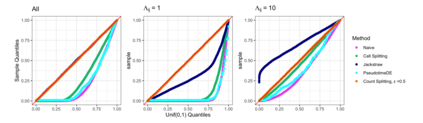
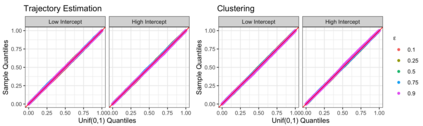
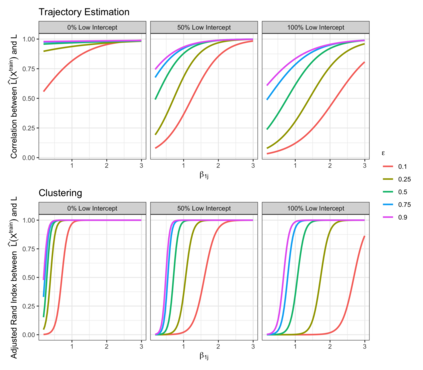
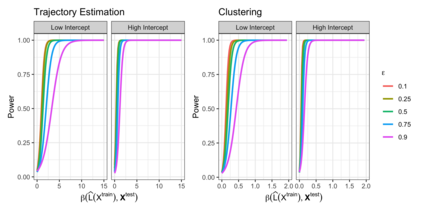
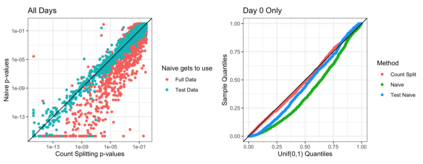
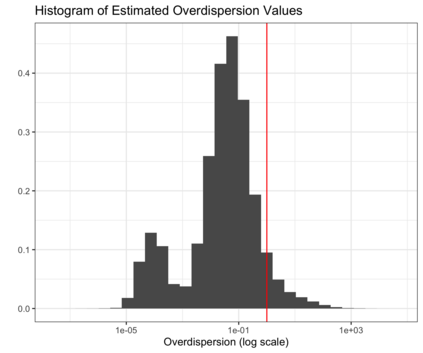
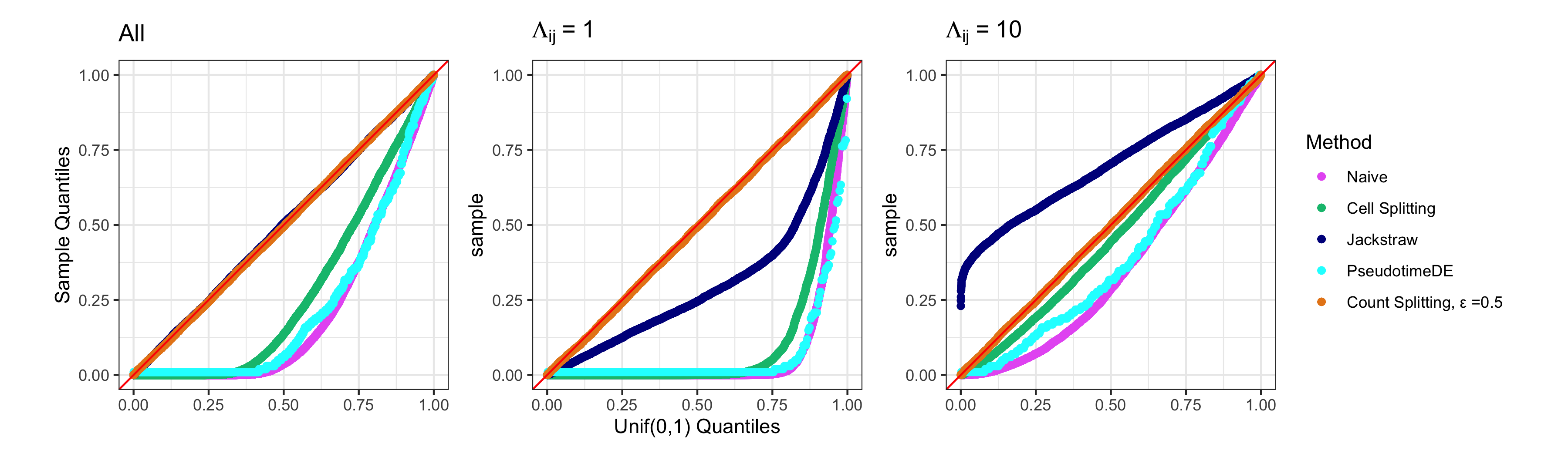
In the analysis of single-cell RNA sequencing data, researchers often characterize the variation between cells by estimating a latent variable, such as cell type or pseudotime, representing some aspect of the individual cell's state. They then test each gene for association with the estimated latent variable. If the same data are used for both of these steps, then standard methods for computing p-values and confidence intervals in the second step will fail to achieve statistical guarantees such as Type 1 error control. Furthermore, approaches such as sample splitting that can be applied to solve similar problems in other settings are not applicable in this context. In this paper, we introduce count splitting, a flexible framework that allows us to carry out valid inference in this setting, for virtually any latent variable estimation technique and inference approach, under a Poisson assumption. We demonstrate the Type 1 error control and power of count splitting in a simulation study, and apply count splitting to a dataset of pluripotent stem cells differentiating to cardiomyocytes.
翻译:在分析单细胞RNA测序数据时,研究人员往往通过估计一个潜在变量,例如细胞类型或假时间,来描述细胞之间的差异,这些变量代表了单个细胞状态的某些方面。然后,他们测试每个基因与估计的潜伏变量有关。如果对这两个步骤都使用同样的数据,那么在第二步计算p值和信任间隔的标准方法将无法实现诸如类型1错误控制等统计保证。此外,在此情况下,诸如可用于解决其他环境类似问题的样本分割等方法不适用。在本文中,我们引入了点数分割,一个灵活的框架,使我们能够根据几乎任何潜在变量估计技术和推论方法,在Poisson假设下,在这个设置中进行合理的推论。我们在模拟研究中演示了类型1错误控制和计数分割的功率,并将点数分割用于对卡迪欧细胞的多精干干细胞数据集。