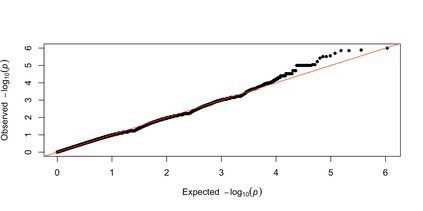
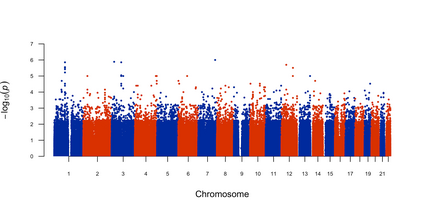
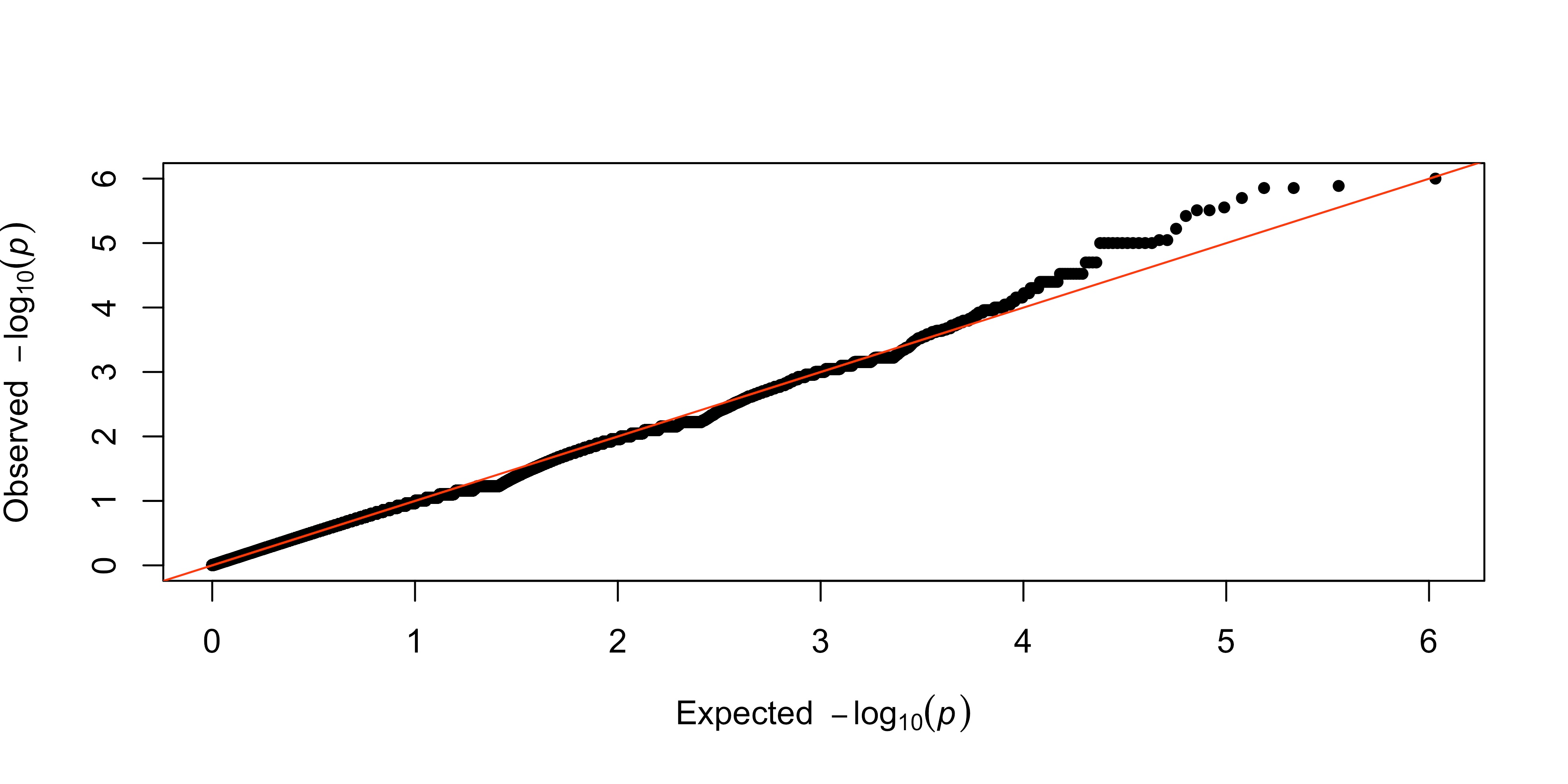
In genome-wide association studies (GWASs), there is an increasing need for detecting the associations between a genetic variant and multiple traits. In studies of complex diseases, it is common to measure several potentially correlated traits in a single GWAS. Despite the multivariate nature of the studies, single-trait-based methods remain the most widely-adopted analysis procedure, owing to their simplicity for studies with multiple traits as their outcome. However, the association between a genetic variant and a single trait sometimes can be weak, and ignoring the actual correlation among traits may lose power. On the contrary, multiple-trait analysis, a method analyzes a group of traits simultaneously, has been proven to be more powerful by incorporating information from the correlated traits. Although existing methods have been developed for multiple traits, several drawbacks limit their wide application in GWASs. In this paper, we propose a multiple-trait adaptive Fisher's (MTAF) method to test associations between a genetic variant and multiple traits at once, by adaptively aggregating evidence from each trait. The proposed method can accommodate both continuous and binary traits and it has reliable performance under various scenarios. Using a simulation study, we compared our proposed method with several existing methods and demonstrated its competitiveness in terms of type I error control and statistical power. By applying the method to the Study of Addiction: Genetics and Environment (SAGE) dataset, we successfully identified several genes associated with substance dependence.
翻译:在全基因组联系研究(GWAS)中,越来越需要发现基因变异和多种特性之间的联系。在复杂疾病的研究中,通常需要同时测量某个基因变异和多种特性之间的关联。尽管研究具有多变性质,单轨基方法仍然是最广泛采用的分析程序,因为其结果具有多种特性,基因变异和单一特性之间的关联可能比较弱,而忽视基因变异和多种特性之间的实际关联可能会丧失力量。相反,多轨分析、同时分析一组特性的方法通过纳入相关特性的信息而证明更为有力。尽管现有方法具有多种特性,但单轨基方法仍然是最广泛采用的分析程序,因为其结果是具有多种特性的研究简单易行,然而,我们建议采用多轨适应性适应性渔业公司(MTAFAF)方法来测试遗传变异异和多种特性之间的关联,同时忽略每个特性之间的实际关联性关联性。提议的方法可以适应连续和双轨分析一组特性,同时通过纳入相关特性的信息,证明更强有力。尽管现有方法针对多种基因变异性的研究方法,但以可靠的方式进行了模拟。我们用各种统计变本研究。