三维基因组中的开放染色质互作网络(一)
北京大学生命科学学院 贾璐萌、李亭亭(李程研究组)
真核生物细胞核中的染色质通过折叠成高度动态、复杂的高级结构,调控基因的转录、复制,以及损伤修复等重要功能。理解染色质在细胞核内如何折叠,基因组的三维空间结构如何调控基因转录、复制和修复等生物学功能,以及探索核染色质在遗传、发育、分化、癌变等生物学过程中的变化规律是当前三维基因组学(Three-Dimensional Genomics)研究领域的主要内容 (Dekker and Mirny, 2016)。
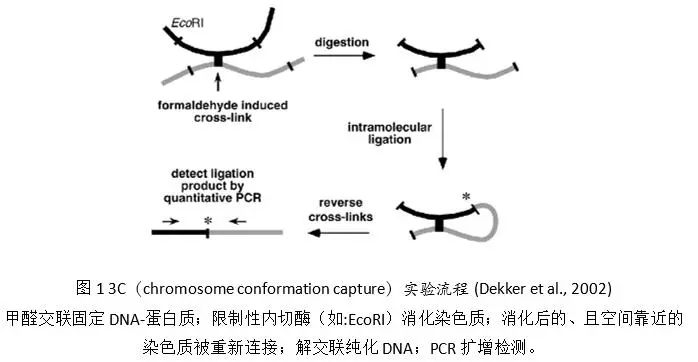
目前研究染色质三维构象的主要方法均是在3C(Chromosome Conformation Capture)实验技术的基础上开发的 (Dekker et al., 2002)。3C实验技术最初于2002年由Dekker等提出 (Dekker et al., 2002)。在3C技术中,染色质构象首先被甲醛交联固定;随后,基因组被限制性内切酶消化;存在相互作用的染色质由于空间接近,在重新连接酶消化后的基因组时,存在相互作用的染色质将被连接到一起;解交联纯化DNA后,针对感兴趣的两个特定基因组区域分别设计引物,并对重连接产物进行PCR扩增;最后通过PCR条带的强度,可对这两个区域的相互作用情况进行定性或定量评估(图1)。3C技术适用于评估两个目标区域之间的空间相互作用情况。为了在一次实验中同时捕获到更多染色质区域的相互作用,2006年,研究人员在3C技术的基础上开发了适用于捕获染色质某一区域与全基因组其他区域间相互作用(一对多)的4C技术 (Simonis et al., 2006; Zhao et al., 2006)以及同时捕获染色质多个区域之间相互作用(多对多)的5C技术 (Dostie et al., 2006)。
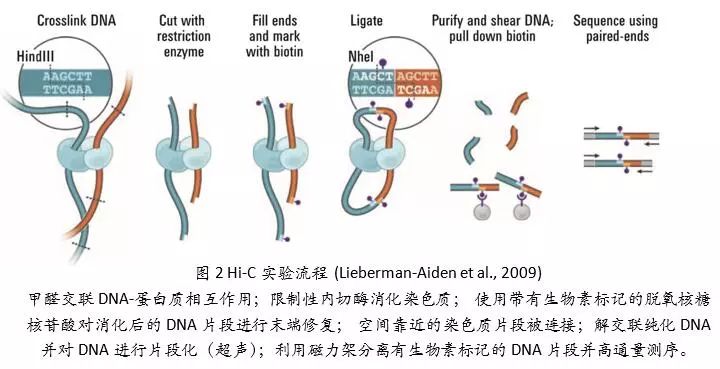
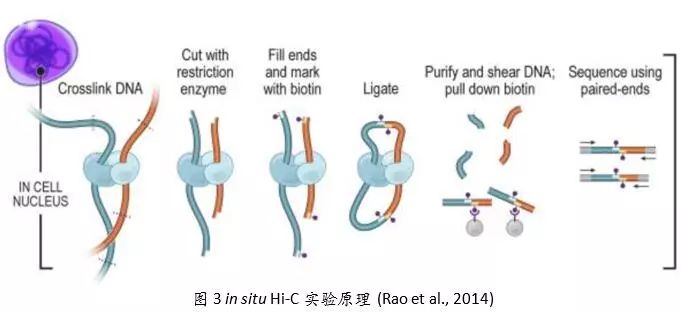
随着二代测序技术的发展,基于3C实验技术、并结合了高通量测序技术的Hi-C技术 (Lieberman-Aiden et al., 2009)被开发(图2)。在Hi-C技术中,限制性内切酶消化后的染色质在末端补平时连入biotin,用于标记重组信号,重组片段通过生物素富集后建库测序。Hi-C技术可以同时捕获全基因组染色质间的相互作用。至此,人类首次染色质空间相互作用图谱被绘制:在染色体上,转录活跃的分区(compartment A)与不活跃的分区(compartment B)交错排列,每个分区长度从几Mb到几十Mb大小不等,其中分区A富集了基因密度高的区域和活跃染色质修饰标记,而分区 B则多是基因荒漠区,染色质组织结构更紧密 (Lieberman-Aiden et al., 2009)。在每个染色体内部还存在更小尺度(平均约800 kb)的拓扑相关结构域(Topological Associated Domains, TAD)(Dixon et al., 2012; Nora et al., 2012),TAD内部的DNA元件之间形成了较为紧密的相互作用,而不同TAD之间的相互作用则较弱。相邻TAD的边界上结合有染色质结构蛋白,如CTCF蛋白、cohesin蛋白复合体等,这些蛋白起到组织染色质结构并隔离两个相邻的TAD之间互作的功能 (Dixon et al., 2012)。2014年,Aiden等开发出了用于原位捕获全基因组水平染色质相互作用的in situ Hi-C (Rao et al., 2014)(图3)。与之前的Hi-C技术相比,in situ Hi-C 替换消化染色质的六碱基识别序列限制性内切酶(通常为HindIII)为四碱基识别序列限制性内切酶(通常为MboI/DpnII),提高了对染色质的切割效率,使更多的染色质空间相互作用被捕获。同时,实验的酶切-连接反应均在细胞核内进行,减少了染色质之间的随机连接。这使得in situ Hi-C技术不仅能得到更高比例的有效数据(reads),同时还提高了染色质相互作用矩阵的分辨率。In situ Hi-C不仅可以观测到A/B compartment,TAD等较大尺度的染色质结构,通过提高测序深度,将染色质相互作用矩阵精度提高至1kb,还可以观测到存在于TAD内部的、由CTCF蛋白介导的环状结构(loop)。这些loop调节增强子和启动子间的相互作用,从而调控基因表达 (Rao et al., 2014)。
尽管in situ Hi-C可以将染色质相互作用矩阵的精度提高到1kb,但它需要产生极大的测序量。以人类基因组为例,欲观测人类细胞核染色质间相互作用的loop结构,需要测到TB级的总数据量。如此庞大的测序量不仅花费大,而且数据处理也更为复杂。因此,Hi-C更适用于研究大尺度上的染色质结构,如TAD和A/B compartment,而不适用于研究转录调控元件之间的相互作用。
参考文献:
Dekker, J., and Mirny, L. (2016). The 3D Genome as Moderator of Chromosomal Communication. Cell 164, 1110-1121.
Dekker, J., Rippe, K., Dekker, M., and Kleckner, N. (2002). Capturing chromosome conformation. Science 295, 1306-1311.
Dixon, J.R., Selvaraj, S., Yue, F., Kim, A., Li, Y., Shen, Y., Hu, M., Liu, J.S., and Ren, B. (2012). Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature 485, 376-380.
Dostie, J., Richmond, T.A., Arnaout, R.A., Selzer, R.R., Lee, W.L., Honan, T.A., Rubio, E.D., Krumm, A., Lamb, J., Nusbaum, C., et al. (2006). Chromosome Conformation Capture Carbon Copy (5C): a massively parallel solution for mapping interactions between genomic elements. Genome research 16, 1299-1309.
Lieberman-Aiden, E., van Berkum, N.L., Williams, L., Imakaev, M., Ragoczy, T., Telling, A., Amit, I., Lajoie, B.R., Sabo, P.J., Dorschner, M.O., et al. (2009). Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science 326, 289-293.
Nora, E.P., Lajoie, B.R., Schulz, E.G., Giorgetti, L., Okamoto, I., Servant, N., Piolot, T., van Berkum, N.L., Meisig, J., Sedat, J., et al. (2012). Spatial partitioning of the regulatory landscape of the X-inactivation centre. Nature 485, 381-385.
Rao, S.S., Huntley, M.H., Durand, N.C., Stamenova, E.K., Bochkov, I.D., Robinson, J.T., Sanborn, A.L., Machol, I., Omer, A.D., Lander, E.S., et al. (2014). A 3D Map of the Human Genome at Kilobase Resolution Reveals Principles of Chromatin Looping. Cell 159, 1665-1680.
Simonis, M., Klous, P., Splinter, E., Moshkin, Y., Willemsen, R., de Wit, E., van Steensel, B., and de Laat, W. (2006). Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C). Nature genetics 38, 1348-1354.
Zhao, Z., Tavoosidana, G., Sjolinder, M., Gondor, A., Mariano, P., Wang, S., Kanduri, C., Lezcano, M., Sandhu, K.S., Singh, U., et al. (2006). Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nature genetics 38, 1341-1347.
Genome Biology
2016 Journal Metrics
Citation Impact
11.9 - 2-year Impact Factor
13.6 - 5-year Impact Factor
2.8 - Source Normalized Impact per Paper (SNIP)
10.5 - SCImago Journal Rank (SJR)
更多关于Genome Biology的信息,请访问
长按并提取二维码浏览BMC期刊列表
点击“阅读原文”阅读英文原文
BMC是施普林格∙自然旗下机构。作为开放获取出版先锋,BMC不断推出一系列高质量的同行评议期刊,包括BMC Biology 、BMC Medicine等涵盖范围较广的期刊,以及Malaria Journal、Microbiome和BMC系列期刊等专门刊物。BMC以“科研永不止步”为信条,致力于不断创新,以更好地满足作者群体的需要,确保所发表论文的完整性,并积极推广开放研究。