AI从底物和酶的结构中预测米氏常数,量化酶活性

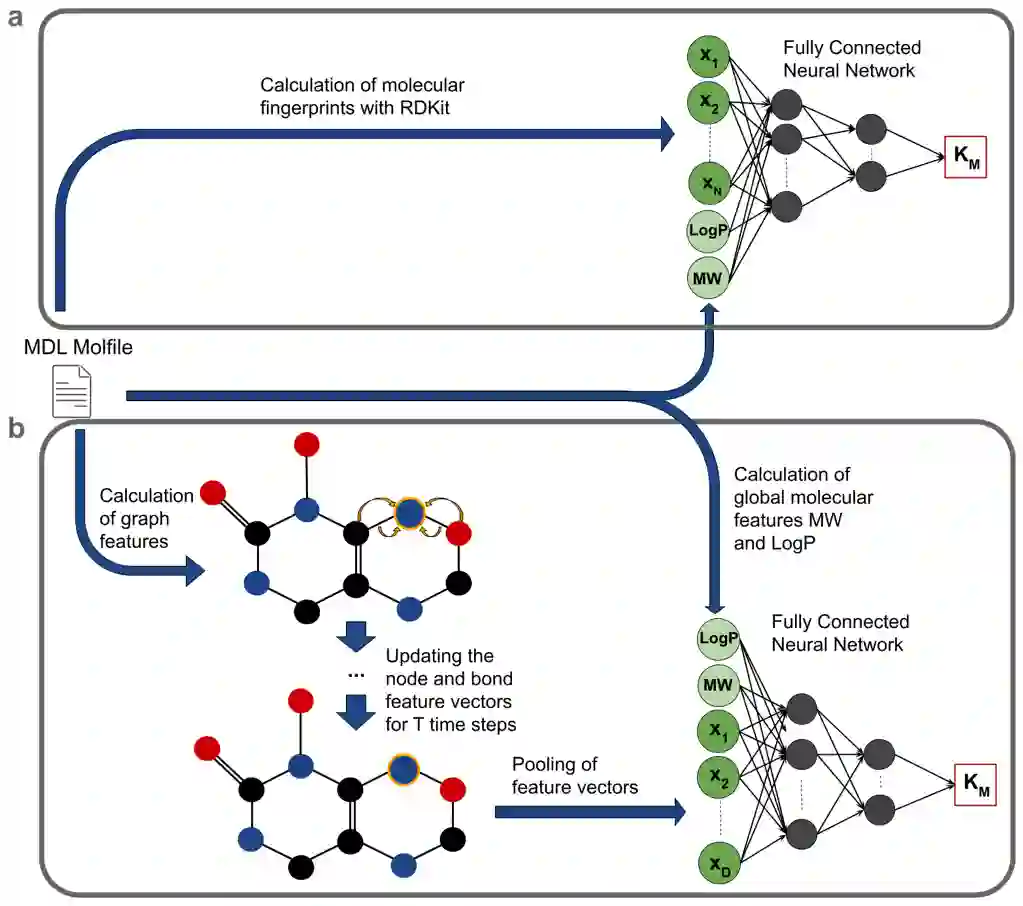
模型概述。
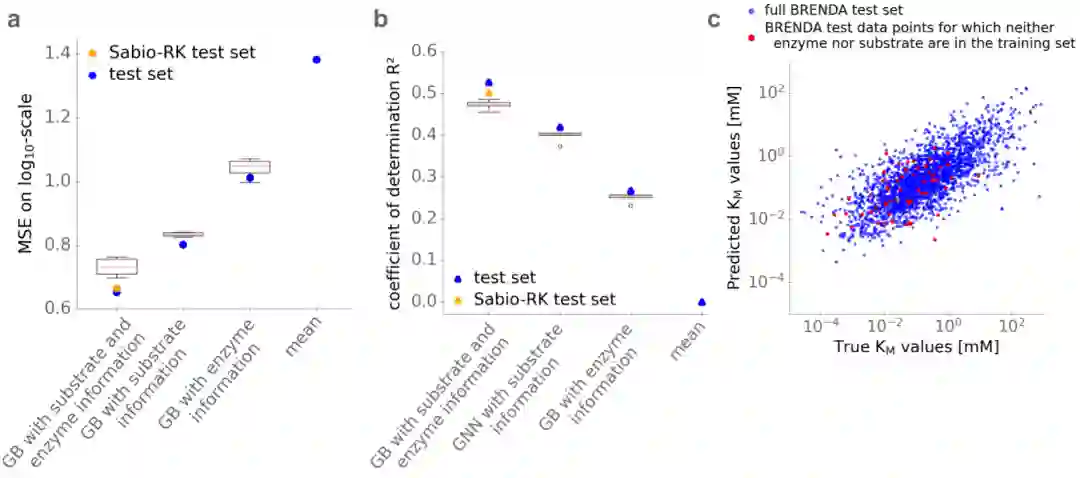
优化模型的性能。

培训集尺寸对模型性能的影响。
人工智能 × [ 生物 神经科学 数学 物理 材料 ]
「ScienceAI」关注人工智能与其他前沿技术及基础科学的交叉研究与融合发展。
欢迎关注标星,并点击右下角点赞和在看。
点击阅读原文,加入专业从业者社区,以获得更多交流合作机会及服务。
登录查看更多
相关内容
Arxiv
0+阅读 · 2022年4月16日