靶向蛋白质降解的蛋白-蛋白相互作用预测
蛋白质-蛋白质相互作用(PPI)在各种生物学功能中起着重要作用,因此,检测PPI位点对于了解疾病和开发新药至关重要。PPI预测与开发采用靶向蛋白质降解的药物特别相关,因为他它们的功效依赖于涉及两种蛋白质的稳定三元复合物的形成。然而,检测PPI位点检测方法既昂贵又耗时。
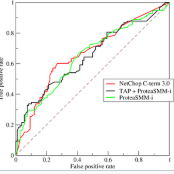
作者提出了一种基于图表示学习概念的新型深度学习框架DGRL[1],该架构可以基于蛋白质的表面表征来预测蛋白质的相互作用位点和相互作用。该模型在MaSIF数据集上达到了最先进的性能(AUC)。
作者还收集了更多样化蛋白质相互作用的新数据集,并表明作者的模型可以很好地推广到这些新数据。该模型还可以预测与靶蛋白降解相关的PPI,作者证明了模型在可用的三元复合物数据上,达到了PPI预测的高精度。
模型细节
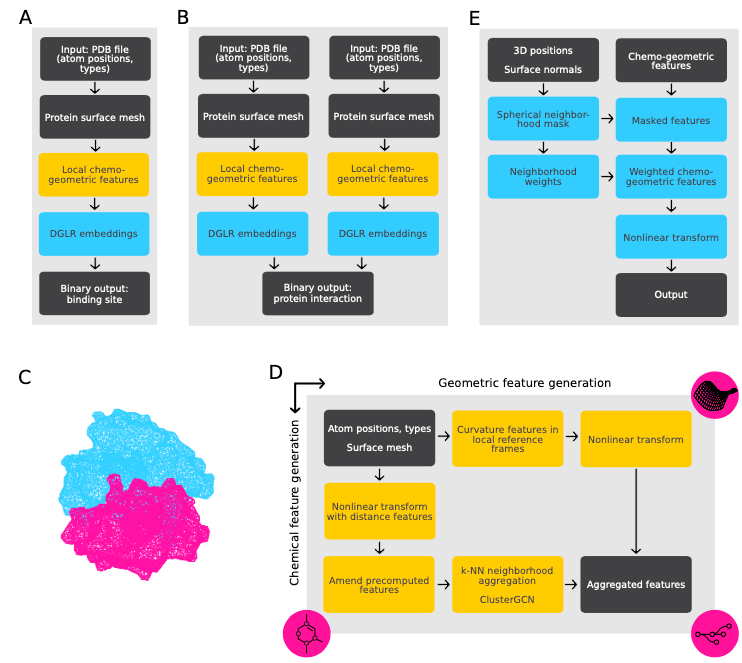
模型细节:A. 结合位点的预测的总体工作流程。该模型输入PDB文件,对蛋白质表面上不同点是否是活性位点的二分类预测。主要处理步骤是表面网格生成,然后计算局部化学几何特征。B. 两种蛋白质相互作用预测的总体工作流程。每种蛋白质在在流程中单独处理,类似于结合位点预测。最后一个步骤组合学习的表征,输出二分类。D. 化学几何特征生成的详细信息。化学和几何特征在单独的流程中生成。几何特征生成(水平)包括在所考虑的点的邻域中估计的曲率特征的嵌入学习。化学特征计算(垂直)包括具有可加的距离依赖性和预计算的化学特征的嵌入学习,这些嵌入在邻域内使用单个基本cluster-GCN层进行聚合处理。E. DGRL处理的详细信息。所考虑的点周围的球形邻域内的化学几何形状使用从3D位置和表面法线生成的学习权重影响此时的特征。加权特征使用多层感知器(MLP)进行处理,从而产生最终输出。
模型DGRL[1],使用Pytorch、PyTorchGeometric和PyKeOps工具。
结论
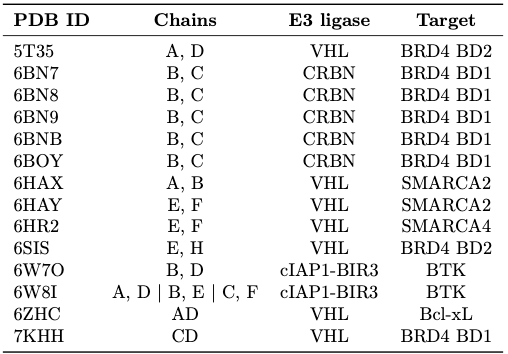
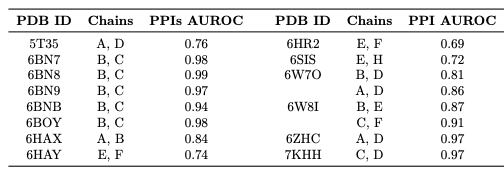
作者在16个三元复合物上评估了训练模型,参与三元复合物的蛋白质的预处理与一般的PPI预测任务相同。模型在三元复合物数据上评估了相互作用预测的AUC,并在不同的数据集上进行了训练。
正如预期的,模型在三元复合物数据的平均AUC达到了0.87。从这个最终模型的高精度来看,将所提出的几何深度学习架构与新数据相结合,可以准确地确定参与三元复合物的两种蛋白质之间是否会发生相互作用。
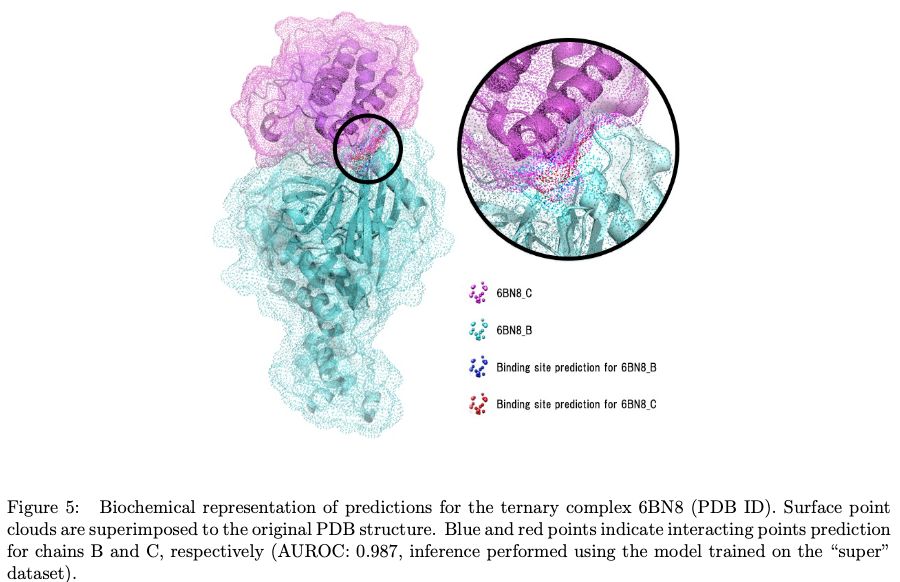
为了证明该算法检测最佳结合位点的能力,下图显示了假设的表面点云,以确定6BN8三元复合物(无降解剂)的两个链的结合位点。发现该算法可以正确检测每种蛋白质的结合位点和两种蛋白质之间的主要相互作用位点。
作者的研究结果表明,在初步筛选过程中使用PPI预测模型可以作为过滤步骤的一部分以识别潜在相互作用的POI和E3连接酶,进行后续的深入分析。
这种方法可以利用机器学习驱动方法的优势来限制需要在计算成本高昂的对接模拟或时间和资源密集型实验室实验中评估的潜在候选者的数量。
总体而言,这些结果表明,使用DGRL进行PPI预测是靶向蛋白质降解的有前途的工具,并且可以用作过滤机制,例如,作为选择可能的感兴趣蛋白质和E3连接酶对的过滤机制,这将在给定合适的降解分子的情况下,可以形成三元复合物。
参考文献
[1] William L Hamilton. Graph representation learning. Synthesis Lectures on Artifical Intelligence and Machine Learning, 14(3):1–159, 2020.