基于结构化 SVM 进行序列标注
作者 seaboat ,本文作者为雷锋网 AI 研习社撰写的独家稿件,未经雷锋网 AI 研习社许可不得转载。
关于SVM
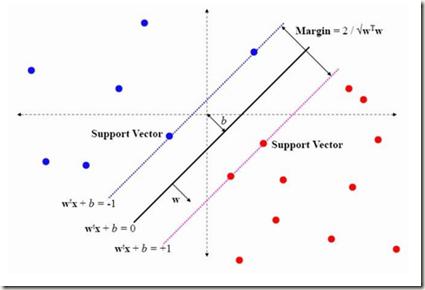
SVM 即支持向量机,常用于二分类模型。它主要的思想是:
1.它是特征空间上间隔最大的线性分类器;
2.对于线性不可分的情况,通过非线性映射算法将低维空间的线性不可分的样本映射到高维特征空间,高维特征空间能够进行线性分析。
什么是结构化
其实机器学习中,如果按照输出空间不同可以分为:
二元分类 (binary classification)
多元分类 (multiclass classification)
回归问题 (regression)
结构化预测 (structured prediction)
其中前面三类都是我们常见且经常用的,第四种结构化预测重点体现在结构化上,前面三类的输出都是标签类别或者回归值之类的单变量,而结构化预测输出是一种结构化的数据结构,比如输入一句话,输出是一颗语法树。此外,结构还可以是图结构、序列结构等。
结构化 SVM
把前面的 SVM 与结构化结合起来就是结构化 SVM 了。它为了处理更加复杂的彼此之间互相存在依赖关系的结构数据,对传统 SVM 进行了改进,可以说结构化 SVM 是在传统 SVM 的基础上扩展出来的。结构化 SVM 使用时主要涉及学习和推理两个过程,与大多数机器学习算法一样,学习其实就是确定模型的参数的过程,而推理就是根据学习到的模型对给定的输入进行预测的过程。
假设给定了训练集

学习过程
学习结构化数据就是要找到上述的一个判别函数,使之在判别函数确定后,对给定的输入
x
,能选择最大化函数 f 值的 Y 作为输出。假定函数 f 的形式为,
其中判别函数

接着还得再定义一个损失函数Δ:Y×Y→R,它应该满足



所以我们的目标是要找到一个 
其中,



但实际情况中经验风险为 0 可能会导致过拟合现象,这时要考虑容忍训练集中某些样本错误分类,从而引入松弛变量,于是优化问题变为:
约束条件引入损失函数的影响,得
那么现在不管是经验风险为 0 还是不为 0,剩下要做的事就是求解上述优化问题,即根据上述各个式子中的约束条件解得最优值 W。怎么求解还是个难题,如果样本数较少且 Y 状态数较少,能用传统的二次优化求解。
而实际情况中样本数和状态数都较多,于是产生的约束条件规模非常大,总数量为
IOB 标记
常用的标注策略有 IOB 标记,即块的第一个符号处标注为 B,块内部的符号标注为 I,块外的符号标注 O。其中 B 为 Begin,表示开始;I 为 Intermediate,表示中间;O 为 Other,表示其他。比如:
我明天去北京。
OBIOBI
实现例子
使用 dlib 库实现结构化 SVM 序列标注功能,以下仅仅是一个简单的功能。对 “我 昨 天 在 学 校 看 到 小 明”,“小 红 刚 刚 才 去 晚 自 习” 中的人名进行标注,并且使用 BIO 标记方式,通过训练后对 “我 昨 天 在 学 校 见 到 大 东” 句子进行人名提取。
输出分别为
小 明
小 红
大 东
#include <iostream>
#include <cctype>
#include <dlib/svm_threaded.h>
#include <dlib/string.h>
using namespace std;
using namespace dlib;
class feature_extractor
{
public:
typedef std::vector<std::string> sequence_type;
const static bool use_BIO_model = true;
const static bool use_high_order_features = true;
const static bool allow_negative_weights = true;
unsigned long window_size() const { return 3; }
unsigned long num_features() const { return 1; }
template <typename feature_setter>
void get_features(
feature_setter& set_feature,
const sequence_type& sentence,
unsigned long position
) const
{
if (sentence[0].compare("小") == 0 || sentence[0].compare("明") == 0 || sentence[0].compare("红") == 0 || sentence[0].compare("张") == 0 || sentence[0].compare("陈") == 0)
set_feature(0);
}
};
void make_training_examples(
std::vector<std::vector<std::string> >& samples,
std::vector<std::vector<std::pair<unsigned long, unsigned long> > >& segments
)
{
std::vector<std::pair<unsigned long, unsigned long> > names;
samples.push_back(split("我 昨 天 在 学 校 看 到 小 明"));
names.push_back(make_pair(8, 10));
segments.push_back(names); names.clear();
samples.push_back(split("小 红 刚 刚 才 去 晚 自 习"));
names.push_back(make_pair(0, 2));
segments.push_back(names); names.clear();
}
void print_segment(
const std::vector<std::string>& sentence,
const std::pair<unsigned long, unsigned long>& segment
)
{
for (unsigned long i = segment.first; i < segment.second; ++i)
cout << sentence[i] << " ";
cout << endl;
}
int main()
{
std::vector<std::vector<std::string> > samples;
std::vector<std::vector<std::pair<unsigned long, unsigned long> > > segments;
make_training_examples(samples, segments);
structural_sequence_segmentation_trainer<feature_extractor> trainer;
sequence_segmenter<feature_extractor> segmenter = trainer.train(samples, segments);
for (unsigned long i = 0; i < samples.size(); ++i)
{
std::vector<std::pair<unsigned long, unsigned long> > seg = segmenter(samples[i]);
for (unsigned long j = 0; j < seg.size(); ++j)
{
print_segment(samples[i], seg[j]);
}
}
std::vector<std::string> sentence(split("我 昨 天 在 学 校 见 到 大 东"));
std::vector<std::pair<unsigned long, unsigned long> > seg = segmenter(sentence);
for (unsigned long j = 0; j < seg.size(); ++j)
{
print_segment(sentence, seg[j]);
}
}
NLP 工程师入门实践班:基于深度学习的自然语言处理
三大模块,五大应用,手把手快速入门 NLP
海外博士讲师,丰富项目经验
算法 + 实践,搭配典型行业应用
随到随学,专业社群,讲师在线答疑
▼▼▼
新人福利
关注 AI 研习社(okweiwu),回复 1 领取
【超过 1000G 神经网络 / AI / 大数据,教程,论文】
开发者自述:我是怎样理解支持向量机(SVM)与神经网络的
▼▼▼