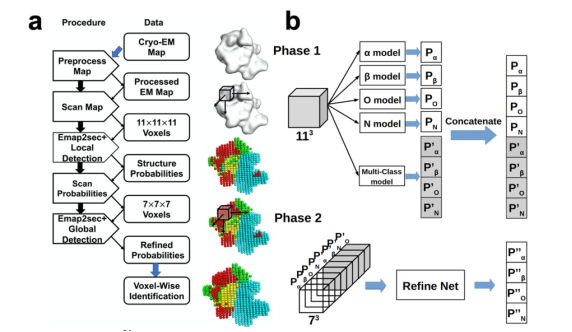
本文介绍的是由普渡大学西拉法叶校区计算机科学系的Xiao Wang, Eman Alnabati, Tunde W. Aderinwale等人发表在Nature Communications上的文章《Detecting protein and DNA/RNA structures in cryo-EM maps of intermediate resolution using deep learning》。越来越多的大分子结构的密度图,包括蛋白质和DNA/RNA复合体,已经通过低温电子显微镜确定。尽管最近在接近原子分辨率的地图经常被报道,仍然有大量的地图确定在中等或低分辨率,提取结构信息不是小事。在这里,本文报道了一种新的计算方法,Emap2sec+,它可以在5到10 Å分辨率的低温电镜图中识别DNA或RNA以及蛋白质的二级结构。Emap2sec+采用深度残差卷积神经网络。Emap2sec+在低温-电磁图的每一个体素上分配具有关联概率的结构标签,这将有助于在电磁图中建模结构。 1.摘要近年来,利用低温电子显微镜(cryo-EM)测定生物分子结构取得了迅速进展。存放在公共数据库EMDB中的确定的低温EM图的数量正在呈指数级增长;此外,其中高分辨率(如优于4 Å)地图的比例呈稳步上升趋势。尽管低温电磁技术取得了显著的进展,但仍有相当一部分地图是用中分辨率或低分辨率确定的。不同的因素控制着EM图的分辨率,包括EM样品的构象或成分的不均匀性、低电子剂量引入的噪声水平以及二维粒子图像的不准确对齐。在此之前,本文开发了一种名为Emap2sec的方法,用于检测中间分辨率EM图中的蛋白质二级结构元素。Emap2sec显示了良好的结果,为中分辨率的地图结构解释提供了一种新的方法。然而,Emap2sec的检测仅限于蛋白质图谱。在这里,本文扩展了该方法,以检测蛋白质二级结构元素和中分辨率的EM图的核酸。新方法Emap2sec+使用了比它的前任Emap2sec更先进的卷积神经网络架构Resnet,并对EM图中的每个体素执行四类分类:α-螺旋、β-链、线圈(其他结构类型)或DNA/RNA。蛋白质与核酸的相互作用是许多基本生物过程的核心,包括转录、翻译、细胞分裂和复制。尽管研究蛋白质-核酸相互作用的结构很重要,但用于DNA/RNA结构建模的工具并不多。Emap2sec+具有较高的核苷酸检测精度,同时保持了与Emap2sec相当的蛋白质二级结构检测性能。 2.数据集本文在两个数据集上测试了Emap2sec+,一个是分辨率为6 Å和10 Å的108个蛋白质和核酸复杂结构的模拟EM图数据集,另一个是分辨率为5 Å和10 Å的84个实验EM图数据集。 3**.**** Emap2sec+架构**
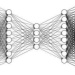
图1**.架构逻辑图**要使用Emap2sec+,输入的低温-电磁图的网格大小需要调整为1.0 Å。Emap2sec+采用从输入EM图中提取的输入体素113 Å3,并在体素的中心输出检测到的结构,该结构可以是DNA/RNA或蛋白质二级结构(α-螺旋、β-片和本文呢所说的“其他结构”)。因此,Emap2sec+将体素分为四个不同的结构类。输入体素以2 Å的步幅滑动到地图中的三个正交方向,并在移动后的每个位置计算输出。由于网格空间为1.0 Å,因此对地图中的每一个其他网格点都进行了结构分配。Emap2sec+的深度神经网络架构如图1所示。Emap2sec+有两个阶段:在第一阶段,Emap2sec+对输入体素执行5个独立的评估。在5个评估中,4个评估来自二进制分类器,其中每个评估器输出体素包含特定结构类(例如,α-螺旋中的残基)的概率。第五种评估是通过多类分类器进行的,它输出四个结构类的四种概率。文本稍后将展示将二元分类器与多类分类器结合使用比仅使用多类分类器性能更好。然后,阶段2网络从阶段1网络中的5个分类器中获取8个概率值。第二阶段网络的输入体素大小为143 Å3,移动步幅为2 Å。由于第一阶段网络在每个其他网格点上分配了概率值,第二阶段输入体素包含73个网格点的概率值。第二阶段网络输出查询体素的中心网格点的四个结构类的最终概率值。由于阶段1和2的网络使用相同的跨步2 Å,阶段2覆盖阶段1的结果。该网络的目的是在决定查询体素时考虑检测到的相邻体素的结构,从而对EM图中的结构分配具有平滑效果。
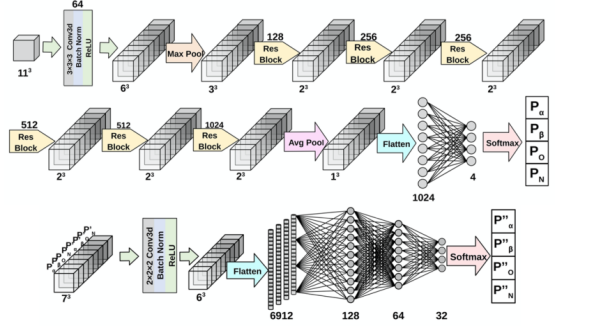
图2.详细网络架构第一阶段网络中五个分类器的详细架构如图1c所示。对于一个大小为113 Å3的输入体素,应用一个卷积块,它由一个包含64个大小为33 Å3的过滤器的卷积层、一个3D批处理归一化层 (批大小设置为256)和一个ReLU激活(生成64个63 Å3的体素)组成。然后,体素被传递到maxpooling层,大小为23 Å3。之后,每个体素连接到6个3D残块,使用大小为33 Å3的128,256,256,512,512和1024过滤器。在最后一步,应用一个大小为23 Å3的平均池化层,得到一个1024值的特征向量,该特征向量连接到一个完全连接的层,以给出最终的概率值。第二阶段网络包含73个相邻体素的概率值。使用卷积块处理输入,卷积块由一个具有32个大小为2的过滤器的3D卷积层、一个3D批处理归一化层和一个ReLU激活层(批处理大小为256)组成,然后是一个完全连接的网络。卷积层的步幅为1。最后,对四种结构类型的输出概率值进行softmax运算。对模拟地图数据进行网络训练,用于模拟地图,对实验地图进行网络训练。 4.** 实验结果**本文研究了Emap2sec+在108个非冗余模拟地图(分别位于6和10 Å)以及一组84个实验地图上的性能。使用独立的训练集分别训练模拟和实验地图的神经网络。
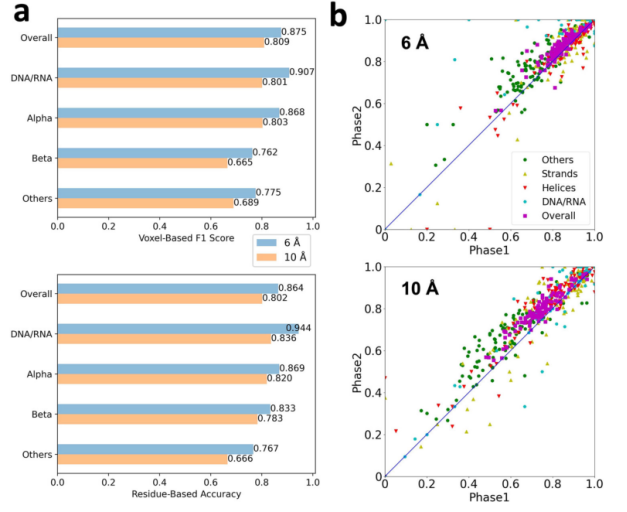
图****3.****模拟图实验结果模拟地图数据集上的结构检测性能。该数据集包含108个以两种不同分辨率计算的结构,6Å和10Å。a:6Å(蓝色)和10Å(橙色)地图的基于体素的F1评分和基于Q4残基/核苷酸的准确性b:在6 Å和10 Å计算的108个测试模拟地图的第1和第2阶段网络输出的第4季度的比较。绿色,其他结构;黄色三角形,β链;红色三角形,α螺旋;青色,DNA / RNA;品红色,Q4评分比较基于体素的准确性和Q4, Q4在所有结构类中都更高。考虑到基于残基/核苷酸的分配是通过体素的多数票进行的,这表明相邻体素的分配趋于一致,这有利于用户在密度图中直观地识别蛋白质和DNA/RNA结构。此外,本文注意到α螺旋和β链的基于段的准确性非常高,分别为0.950和0.940的6Å图。这些结果强烈表明,Emap2sec+的结构分配将能够帮助cryoEM图的主链跟踪和域结构分配。10Å地图的结果比6Å地图的结果差6 - 13%。在四个结构类中,α螺旋的差异最小,在6Å和10Å地图之间,基于体素的F1评分差距为7.5%,Q4为5.6%。尽管有所下降,但值得注意的是,基于体素的F1总分和10 Å的Q4总分维持在0.8的高位。因此,本文可以得出结论,即使是10Å分辨率的模拟地图,也含有丰富的结构信息。
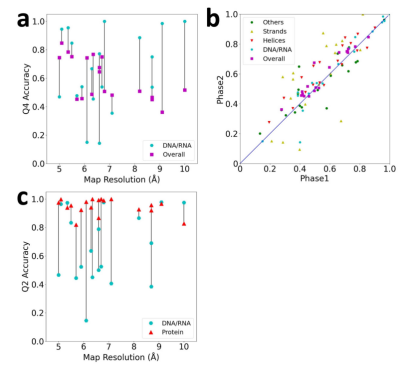
图****4.****实验图实验结果19张实验图的结构类检测: a.实验地图相对于地图分辨率的Q4精度。整体Q4用洋红色的方块表示,DNA/RNA的Q4用青色的圆圈表示。线连接同一地图映射的值。B.第一阶段和第二阶段网络基于残差的精度比较。c.Q2区分实验图谱中蛋白质和DNA/RNA类的二元分类精度。注意,DNA/RNA的值可能与报告四类分类结果的面板a不同。由于蛋白质类别的概率计算为三个二级结构类别的概率之和,因此四类分类中的DNA/RNA分配可以更改为二类分类中的蛋白质。
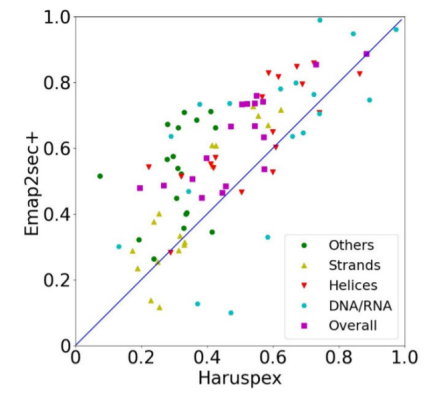
图5.与相关工作的比较在本文的工作之前,用于检测DNA/RNA和蛋白质结构的方法非常有限。流行的结构建模工具最初是为x射线晶体学开发的,ARP/wARP (v8.0)8、Phenix7和Brickworx26,只适用于仅包含蛋白质或DNA/RNA的地图。这项工作的前身Emap2sec仅用于蛋白质二级结构检测。如前所述,Emap2sec+在蛋白质结构分配上的表现优于Emap2sec。最近的一项工作haruspex是最接近Emap2sec+的,因为它使用了深度神经网络来检测DNA/RNA和蛋白质结构。然而,它们的工具和目标应用的目的是完全不同的;Haruspex的设计目的是在4 Å的高分辨率地图中检测结构,或更好地检测从低温- EM构建的结构模型中的潜在错误,或协助建模过程,而Emap2sec+是为6-10 Å的地图提供结构线索,否则结构信息是不容易检测到的。 当考虑所有19张地图的F1总分平均值时,Emap2sec+的值为0.620,高于Haruspex的0.488。有一张地图上Haruspex的F1总分更高。在单个结构类水平上,Emap2sec+对α螺旋、β链、其他结构和DNA/RNA的平均F1分分别比Haruspex高0.105、0.066、0.197和0.043。这两种方法在DNA/RNA类的表现最接近,其中Haruspex在19个地图中的8个显示了更高的F1分。由于这两种方法具有不同的设计和目的,本文的比较只是为了说明它们在本质上的不同,并了解Emap2sec+的性能。Emap2sec+在α螺旋、β链、其他结构和DNA/RNA图谱上分别表现出较高的F1分。 5. 总结本文汇报了Emap2sec+,这是一种基于深度学习的方法,可以在中等分辨率下检测EM map中的结构(5-10 Å),它通过实现核酸检测和提高蛋白质二级结构检测精度,大幅升级了之前的Emap2sec。核苷酸的检测效果特别好,即使在分辨率较低的地图上,准确性也没有下降多少。这项工作首次探索了蛋白质-核酸复合体在这一困难分辨率范围内的结构信息。同样的深度学习策略将被用于检测EM图中的其他分子或结构,如氨基酸类型。Emap2sec+将帮助结构分配和建模,实现快速和准确的预测,将是低温电磁结构生物学时代一个有用和强大的工具。