蛋白质数据库(Protein Data Bank)收录着成千上万实验鉴定的蛋白质结构数据,可惜的是,目前PDB仅收录35%的人源蛋白质的结构。近年发布的深度学习蛋白质结构预测工具AlphaFold2(AF2)能够从蛋白序列准确预测蛋白质结构,可以在一定程度上弥补PDB收录结构数量上的不足。然而如何评价AF2的预测结果的准确程度颇为重要。 近期,丹麦科技大学的Kasper P. Kepp教授团队提出使用精确计算残基的相对溶剂可及表面(relative solvent accessible area ,RSA)作为评估指标,可用以评价AF2对蛋白质结构预测的准确度。作者认为残基的溶剂可及表面积包含着蛋白质功能和进化信息,是一个直接用于深度学习模型训练或外部验证的可解释自然特征。为了验证猜想,作者建立了一个AF2预测结构和实验结构相对应的数据对库并针对RSA进行了一系列测试。该工作近期发表于美国化学会出版的计算化学核心期刊Journal of Chemical Information and Modeling[1]。 首先,作者从AF2预测结构数据库(AlphaFold Protein Structure Database)获取人源参考蛋白质组;从PDB中获取使用X-ray晶体学方法鉴定、分辨率<2.0Å的人源蛋白质结构。然后将每个AF2结构先后通过匹配UniProt编号配对、序列比对,产生对应的数据对库。为了探寻可能影响AF2性能的其他因素,作者依据(1)序列一致性比例(2)实验结构的分辨率(3) 实验结构是否为单体为基准,拆分成子数据集,将这些结构进一步整理成分为六个非重叠组。 随后,研究者计算并比较了AF2生成结构(RSAAF)和实验结构(RSAEP)中每个残基主干的RSA,从平均RSAExp值计算出的平均绝对偏差(MAE)、平均符号偏差(MSD)和标准差(SD),发现仅针对蛋白质单体而言,MSD和SD值不依赖于序列一致性或实验结构的分辨率,说明AF2预测单体结构的性能与配体的存在无关。
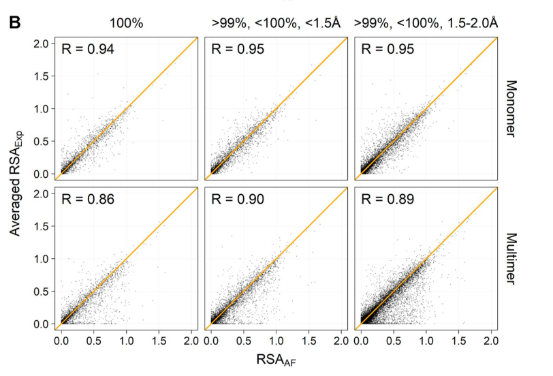
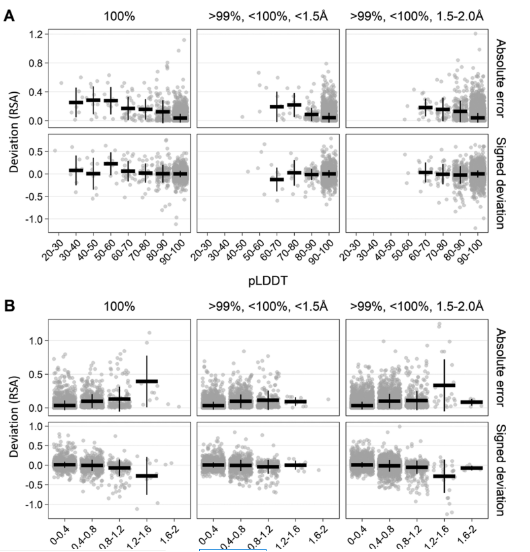
图 1 根据序列一致性、实验结构分辨率和实验结构的单体-多聚体状态分组的结果实验对比的AF2和RSA值 为了确定AF2预测置信度pLDDT与预测****RSA的准确性是否相互影响,作者将结构对上的残基按照pLDDT和RSA分别划分区间,发现RSAAF没有因残基pLDDT值低受到影响(图2A);但在高****RSA的残基上AF2预测置信度较低,AF2对于低****RSA的包埋残基预测更准确。(图2B)
图 2 pLDDT和RSA相关的偏差.(A)对pLDDT函数的偏差;(B)对RSAExp的偏差 然而,与蛋白质单体相比,多聚体结构更加复杂,存在着不同链之间的界面残基,实验结果也显示了这种差异的存在(图3左)。作者识别并移除了链界面可能具有较低溶剂可及性的残基(与其他链残基距离<3.5Å),发现剩余残基RSAAF和RSAExp之间的相关性更强,达到了单体数据对的相关性水平(图3右)。实验结果说明,AF2对多聚体蛋白复合物链界面残基RSA的预测值存在偏高现象。
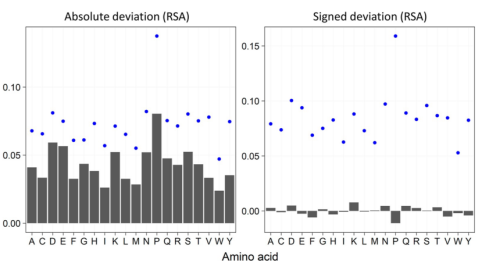
图 3 多聚体实验结构中的实验与AF2 RSA值。界面残留物(左)和非界面残留物(右)的RSAAF和RSAExp的相关性。 研究者还认为AF2预测RSA的准确性也可能取决于氨基酸类型。实验结果(图4)说明最易预测的氨基酸往往是非极性的,例如异亮氨酸((I)、亮氨酸(L)等,该类氨基酸更多处于包埋残基。而位于蛋白质表面区域的极性氨基酸和脯氨酸较难预测,如天冬氨酸(D)和谷氨酸(E)等,尤其是脯氨酸(P)。
图 4 实验和AF2结构之间的一致性取决于残基类型;左:MAE。右:MSD(RSAAF−RSAExp);SDs显示为蓝点小结
****这项工作提出蛋白质局部残基的相对溶剂可及表面(RSA)是一种包含着蛋白质功能和进化信息的自然特征,可用于评估AF2预测性能。通过作者研究发现RSA与AF2预测性能的相关性高度依赖于人源蛋白的单体/多聚体状态;与AF2预测置信度高低、结构中辅因子和配体的存在与否、结构鉴定分辨率均无关。小编认为仅蛋白单体预测而言,该工作提出的RSA特征,可作为目前主流的评估AF2预测性能的RMSD、pLDDT等指标的补充,共同对AF预测结构准确性做出精确的评估。 参考文献[1]Bæk KT, Kepp KP. Assessment of AlphaFold2 for Human Proteins via Residue Solvent Exposure. J Chem Inf Model. 2022;62(14):3391-3400.