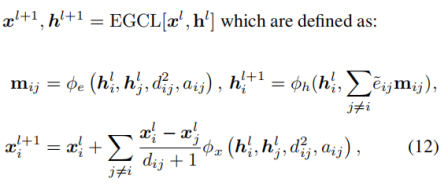本文将给大家介绍一篇由Emiel Hoogeboom等人发表在 Proceedings of the 39th International Conference on Machine Learning的《Equivariant Diffusion for Molecule Generation in 3D》,在本工作中,引入了E(3)等变扩散模型(EDMs)。EDM学习针对坐标和原子类型上的扩散过程的去噪过程。这是第一个直接在三维空间中产生分子的扩散模型。本文的方法不需要特定的原子排序(与自回归模型相比),并且可以比归一化流更有效地进行训练。
摘要文章引入了一个与欧几里得变换等变的三维分子生成的扩散模型。E(3)等变扩散模型(EDM)学习了用一个等变网络来去噪一个扩散过程,该网络在连续特征(原子坐标)和分类特征(原子类型)上共同操作。此外,文章提供了一个概率分析,允许可能性计算的分子使用文章提出的模型。实验结果表明,该方法在生成样本的质量和训练时的效率方面都显著优于以往的三维分子生成方法。 1.研究背景现代深度学习方法正开始对分子科学产生重要影响。在Alphafold在蛋白质折叠预测方面的成功后,越来越多的文章运用深度学习模型来分析或合成分子。分子存在于物理的三维空间中,因此它们受到几何对称性的影响,如平移、旋转和可能的反射。这些对称性被称为在三维空间中的欧几里得群,即E(3)。在分子数据中利用这些对称性对于良好的泛化很重要,并且已经得到了广泛的研究。虽然运用为分类任务,E(n)等变层也可以用于三维分子生成。特别是,它们已经被整合到自回归模型中,这些模型人为地计算了原子的顺序,并且已知在采样过程中难以缩放。另外,连续时间归一化流,如E-NF,对于训练是昂贵的,因为它们必须积分一个微分方程,导致有限的性能和可伸缩性。文章引入了E(3)等变扩散模型(EDMs)。在QM9数据集上训练时,EDM产生的稳定分子是E-NF的16倍,而需要一半的训练时间。这种有利的缩放行为允许EDM在更大的类药物数据集上进行训练,如 GEOM-Drugs数据集。文章的贡献可以总结如下。引入了一种作用于原子坐标和分类特征的等变去噪扩散模型;添加了一个概率分析,它允许可能性计算,这种分析对连续和分类特征是一致的;证明了文章的方法在对数似然性和分子稳定性方面优于以前的分子生成模型。 2.模型
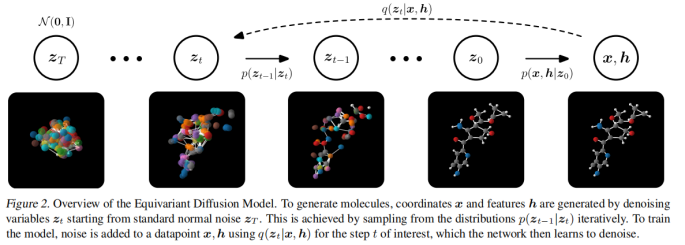
图1.等变扩散模型的概述EDM利用了最近较火的扩散模型来生成分子,过程如图1所示。扩散模型定义一个扩散步骤的马尔可夫链,逐渐向数据添加随机噪声,然后学习逆扩散过程,从噪声中构建所需的数据样本。即模型分为两个过程:扩散过程和去噪过程。
图2.EDM模型训练过程的伪代码针对分子生成,模型训练过程如图2所示。具体过程如下:**步骤1:**定义一个参数T,含义是分子扩散的步数。分子的扩散过程被定义为一个马尔可夫过程,t+1时刻的分子只与t时刻的分子有关,从高斯分布中采用噪声,增加到在t时刻分子上,生成t+1时刻的分子。每一时刻的高斯分布参数是提前生成的,是不可训练参数。**步骤2:**针对一个batch的分子,首先从均匀分布U(0,T)采样batchsize个时刻t,t与分子一一对应。步骤3:针对分子坐标x和分子特征h从标准正态分布中随机采样噪声ε。分子坐标的噪声需要减去重心。不同的特征噪声需要乘不同的尺度因子σ。利用步骤1生成的对应时刻的高斯分布参数和重参数技巧将噪声加在原分子特征上形成新的分子,就是该分子在t时刻的扩散后的样本。**步骤4:**将扩散后的样本和时刻t作为E(n)等变图神经网络(EGNNs)的输入,用该网络预测增加的噪声。**步骤5:**将步骤3采样的真实噪声和步骤4预测的噪声求损失。用来预测噪声的EGNNs网络是一个等变网络,等变网络可以将一个不变分布映射到另一个不变分布。所以要求输入的特征是E(3)不变的,即随着分子的平移、旋转等变化,分子特征的值不变。分子的电荷数(整数标量)、原子类型(one-hot feature)特征本身就是不变的,为了保证分子坐标(实数向量)不变,这里采用的方法是分子坐标减去重心。这样保证了分子的E(3)等变性。EGNNs网络的每一层架构如图3所示,其中的每一个网络的构成如图4所示。
图3.EGNNs网络的层架构
** ****图4.EGNNs中每个网络的具体组成**模型利用的分子特征有电荷数(整数标量)、原子类型(one-hot feature)、分子坐标(实数向量)。由于不同的分子特征形式不同,无法通过对所有特征加相同的高斯噪声来训练模型。对此,文章设计了针对不同特征的加噪方法和损失,并且调整输入特征的尺度来使训练更加稳定。文章将EDM模型的输入定义为[x,0.25h(one-hot feature),0.1h(atom charge)]显著提高了性能。
图5.分子采样过程的伪代码分子的采样过程如图5所示,具体过程如下:步骤1:从标准高斯分布中采样分子特征,构成T时刻的分子。步骤2:在每一时刻中,将该时刻的分子输入到训练好的EGNNs模型中预测该时刻的噪声,代入公式,计算下一时刻的分子,直到时刻t=1得到生成的分子。为了适应不同大小的不同分子,计算了训练集上分子大小的分类分布P(M)。为了从模型P(x,h,M)中采样,首先从P(M)中采样原子数目M,然后利用EDM从P(x,h|M)中采样x,h。 3.实验结果
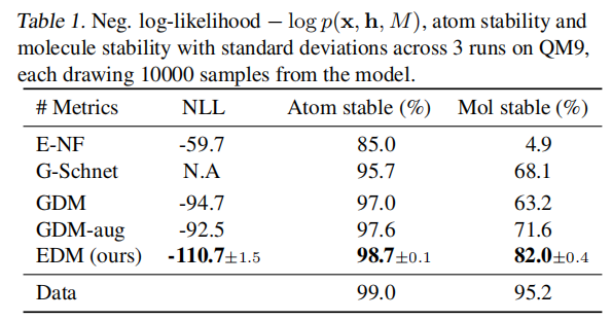
**1.实验一:分子生成—QM9****数据:**QM9是一个标准数据集,包含130k个小分子,最多9个重原子(29个原子,包括氢)的分子性质和原子坐标。评价标准:使用原子对和原子类型之间的距离来预测键类型(单键、双键、三重键或无键)。测量原子的稳定性(具有正确价的原子的比例)和分子的稳定性(生成的所有原子都是稳定的分子的比例)。实验一训练EDM无条件地生成具有三维坐标、原子类型(H、C、N、O、F)和整数原子电荷的分子,实验结果如表1所示。同时计算了生成分子的有效性和独特性,如表2所示。表1 .实验一的实验结果
表2.利用QM9数据集生成分子的有效性和独特性
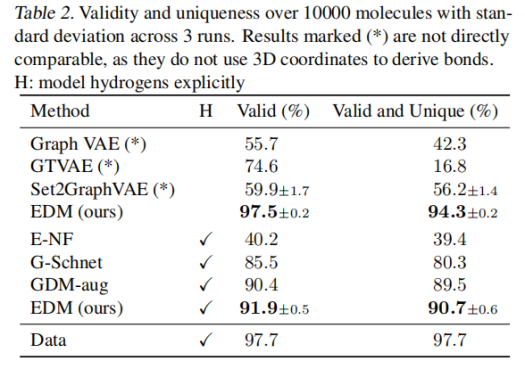
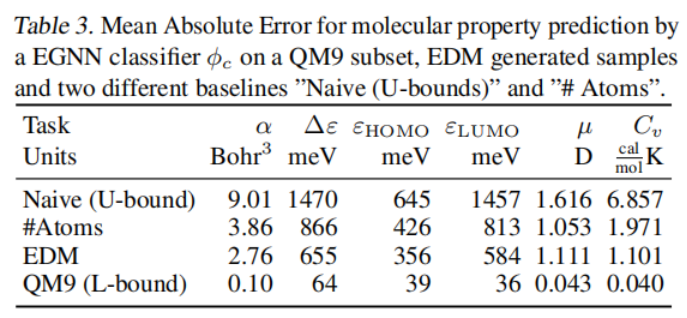
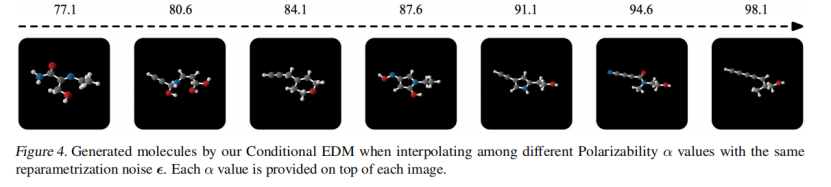
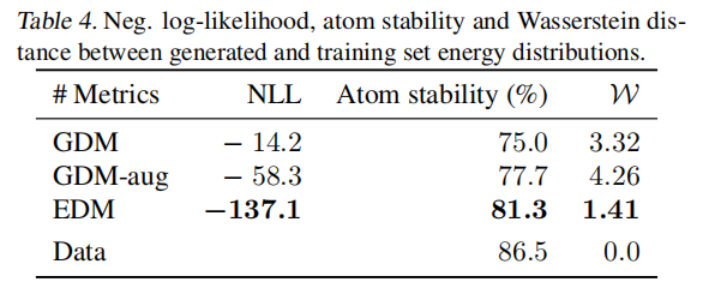
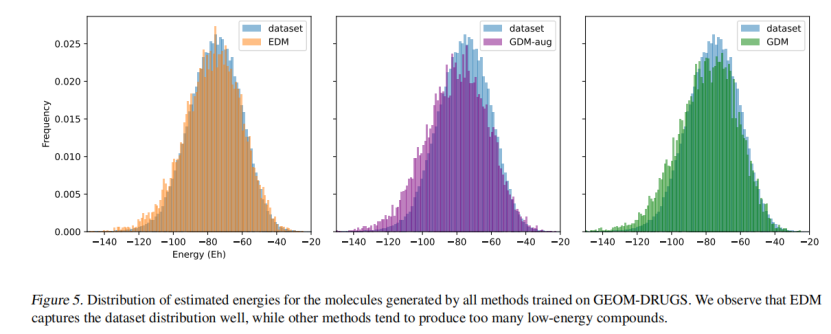
图6 .不同α下的分子生成样例从表3可见EDM在每一性质下效果都比Naive(U-bound)和Atoms好,但是与QM9还有一定的差距以及可以优化的空间。 3.实验三:分子生成—GEOM-DrugsQM9只有小分子,而GEOM-Drugs是一个更大规模的分子构象数据集。它有43万个分子,平均有多达181个原子和44.4个原子。对于每个分子,都有许多构象和它们的能量。从这个数据集中,保留了每个分子的30个最低能量构象。这些模型学习了如何生成这些分子的三维位置和原子类型。所有的模型都使用4层,每层有256个特征,并使用批处理大小为64、学习率为10−4的Adam进行训练。实验结果如表4所示。关于模型能量捕获方面,文章绘制了图7。 表4 实验三的实验结果
图7.DM与非等变对应物的比较从表4得出结论,利用EDM模型在数据集GEOM-Drugs上生成的分子的负对数似然比baseline低,原子稳定性高,效果较好。EDM在所有指标上都优于它的非等变对应物。特别是,从图7可以看出EDM能够很好地捕获能量分布。